Титульный лист и исполнители
Реферат
Отчет выполнен на 103 страницах, содержит текст отчета, 10 таблиц, 26 рисунков, выводы, представленные в конце каждой главы, заключение, список литературных источников.
Целью научно-исследовательской работы является проведение молекулярно-генетических исследований овец отечественных пород различными методами, анализ полученных данных и расширение за счет этого отечественных баз геномных данных в соответствии с современными мировыми научными тенденциями.
В результате проведенного полногеномного генотипирования овец волгоградской породы (n=48) с использованием OvineSNP50 Genotyping BeadChip получены данные, характеризующие 54241 SNP, расположенные в геноме овец. По результатам анализа были определены 40 SNP, ассоциированных с плодовитостью овец, а также определены 14 генов, которые могут быть ответственны за признаки, связанные с воспроизводительной функцией.
Результаты молекулярно-генетических исследований овец волгоградской породы (n=117) показали, что полиморфизм гена GDF9 у овец волгоградской породы представлен двумя аллелями A и G с частотами 0,04 и 0,96 и двумя генотипами AG и GG с частотами 8,55 и 91,45%. Установлено влияние полиморфизма гена GDF9 на массу ягнят при рождении. У маток генотипа AG вес ягненка при рождении в одинцовых пометах (первый окот) был выше на 0,14 кг (4,02%, р=0,09) по сравнению с матками генотипа GG, при этом вес ярочек был выше на 0,17 кг (5,04%, р=0,1) и баранчиков на 0,14 кг (3,90%, р=0,12). Во втором окоте вес ягнят из двоен, полученных от маток генотипа AG был выше на 0,18 кг (7, 11%, р=0,2). Различия между ярочками составили 0,21 кг (8,92%, р=0,2) и баранчиками 0,19 кг (7,11%.р=0,2).
В отчете представлены результаты исследования полиморфизма гена гормона роста (GH) у овец карачаевской породы (n=106). Результаты молекулярно-генетических исследований показали полиморфизм гена гормона роста (GH) у овец карачаевской породы и наличие трех генотипов АА, AВ и ВВ. Частота встречаемости трех генотипов АА, АВ и ВВ установлена в ходе эксперимента в соотношении 51, 40 и 15%, соответственно Установлено влияние полиморфизма гена гормона роста (GH) на мясные качества овец карачаевской породы.
Проведен обзор международных баз геномных данных с точки зрения характера и объема предоставляемой информации, методов доступа к данным, что будет способствовать повышению точности и достоверности анализа молекулярно-генетической информации. Дополнена собственная база данных генетических ресурсов с.-х. животных для анализа и последующей интеграции с международными базами данных.
Ключевые слова: полногеномные исследования, сельскохозяйственные животные, ДНК, SNP, биочип, племенное животноводство, племенная оценка, продуктивные качества, международные базы генетических данных, информационные ресурсы
Введение
В условиях внешнеполитических рисков, оказывающих негативное влияние на экономику сельскохозяйственного производства РФ, решающую роль приобретает его конкурентоспособность. Конкурентоспособность отраслей животноводства в большой степени зависит от возможности практического применения инновационных технологий. Современное российское овцеводство не является исключением. Овцеводство – одна из самых древних отраслей животноводства. Она приносила огромную пользу человечеству на тех стадиях его становления и развития, когда начался переход от простого потребления продуктов природы к их производству. Не теряет своего значения овцеводство и в наше время. И на сегодня, и на ближайшее будущее трудно представить жизнь большинства людей без потребления продуктов овцеводства [2].
Российская Федерация располагает большими возможностями, как для роста численности овец, так и для увеличения производства всех видов продукции отрасли. На её территории имеются естественные пастбища, которые овцы могут рационально использовать для обеспечения потребности в корме без существенных материальных затрат; есть уникальные породы овец, характеризующиеся высоким генетическим потенциалом продуктивности и хорошей адаптацией к различным природно-климатическим условиям; имеется значительный спрос на экологически чистую мясную продукцию овцеводства, как в России, так и за её пределами. Баранина – один из основных видов продукции овцеводческой отрасли. На протяжении последних 20-25 лет потребление баранины в стране остается стабильным (1-1,2 кг/чел. в год), а положительного сдвига в отрасли можно ожидать лишь тогда, когда будет сформирован устойчивый потребительский спрос на этот вид продукции. За первое полугодие 2018г. Россия импортировала 1,7 тыс. тонн, при этом экспорт баранины составил 4,1 тыс. тонн против 41 тонны за аналогичный период прошлого года. Основным потребителем российской баранины стал Иран [17, 20, 34, 40].
В значительной мере это можно объяснить тем, что в мае на полях Астанинского экономического форума страны ЕАЭС и Иран подписали соглашение о создании зоны свободной торговли, в рамках которого зафиксировано, что импортная пошлина на эту продукцию не превысит 5%. Таким образом, принимая во внимание результат экспорта баранины, зафиксирована высокая положительная динамика потребительского спроса со стороны внешнеэкономических торговых партнёров [20]. А это формирует хорошие перспективы мясного овцеводства.
Каждая порода овец характеризуется уникальным генофондом и является итогом длительного, целенаправленного и напряженного труда. В процессе многолетнего интенсивного отбора в породах складываются устойчивые коадаптивные генные комплексы, определяющие специфические признаки той или иной породы и адаптивную норму популяций [22].
После значительного спада поголовья овец во многих районах страны начинается рост их численности на качественно новом уровне. Поэтому требуется более продуманный, основанный на современных технологиях, подход к восстановлению поголовья [63].
Геномная оценка является наиболее эффективным инструментом повышения генетического потенциала сельскохозяйственных животных. Еще большую значимость она приобретает в контексте задач совершенствования отечественных племенных ресурсов и сохранения биоразнообразия сельскохозяйственных животных. Поэтому проблема накопления и расширения информационной базы генетических данных отечественных локальных пород является весьма актуальной, т.к. подобная база является информационной основой для изучения популяционной структуры пород, выявления уникальных районов генома, подверженных позитивной селекции, выявление конкретных генетических вариантов, отвечающих за адаптацию, продуктивные и другие характеристики, представляющие интерес для продуктивного животноводства. Наряду с этим, обладает актуальность и проблема привлечения мировых информационных ресурсов, предоставляющих данные по результатам генетических исследований сельскохозяйственных животных со всего мира, т.к. это, с одной стороны, значительно расширяет информационное поле исследований, что повышает возможности анализа, а, с другой стороны, обеспечивает интеграцию отечественных исследований в мировую науку. Огромный интерес к молекулярно-генетическим методам исследований и их использование в практике селекционера, позволяет предполагать, что определение племенной ценности овец на основе сочетания классических методов оценки и отбора в сочетании с ДНК-маркерами станет наиболее точной и более ранней формой оценки. Поэтому разрабатываемая нами научная тематика является актуальной и представляет существенный практический интерес.
Современное состояние проблемы.
Маркер-ассоциированная селекция.
Одним из способов интенсификации отрасли и повышения его конкурентоспособности является внедрение в производство современных методов селекционно-племенной работы. Основа современной селекции – рациональное использование имеющихся генетических ресурсов племенных животных, эффективное и своевременное воспроизводство стада, получение животных с желаемыми физиологоморфологическими характеристиками и оптимальным уровнем хозяйственно-полезных признаков. В большинстве случаев данные задачи невозможно решить без применения методов маркер-ассоциированной (MAS) селекции [13, 19, 37-39], т. к. классические приёмы определения племенной ценности либо имеют значительный уровень погрешности, либо носят растянутый во времени характер. Маркерная селекция – современное перспективное направление в животноводстве, позволяющее эффективно использовать выявленные гены-маркеры хозяйственно-полезных признаков для повышения эффективности селекционной работы [42].
Практика применения молекулярно — генетического маркирования в животноводстве в настоящее время широко распространена в мире. Последнее время особенно популярны исследования, направленные на поиск взаимосвязи уровня продуктивности, необходимых качественных характеристик животных с определенными генетическими маркерами. При этом в качестве молекулярно-генетических маркеров рассматривают фрагменты генов хозяйственно-полезных признаков, характеризующиеся наличием полиморфизма в своей структуре, удобного к выявлению. Доходность овцеводства определяется прибыльностью производства основных видов продукции, прежде всего, шерсти и баранины. Ранее, в нашей стране экономика овцеводства базировалась на производстве тонкой шерсти и уже потом баранины. Несмотря на изменение экономической значимости этих видов продукции, улучшение качества шерсти, в том числе грубой, остается важной задачей. Для ее решения необходимо улучшение технологии производства продукции и использование новых приемов и методов селекции овец [67, 69, 73, 76, 89, 93].
Для выведения овцеводства из сложной экономической ситуации в первую очередь надо совершенствовать систему и технику ведения этой отрасли, разрабатывать новые более эффективные методы разведения овец, отдавая предпочтения направлениям и породам, лучше оплачивающим затраты на производство продукцией. Особенно важное значение приобретает в таких условиях направление овцеводства, ориентированное на комбинированную продуктивность – производство баранины, ягнятины, молока, шерсти и меховой продукции в одной породе. Создание таких овец – задача селекции. Для ее выполнения необходимо знание как современных требований к продукции овцеводства, так и накопленных зоотехнией сведений об овцах и приемах их воплощения в жизнь [48].
В современном мировом овцеводстве конкурентоспособным, является разведение овец, которые сочетают в себе, например, достаточный уровень мясной и молочной продуктивности. Одной из таких пород является карачаевская, которая распространена в зонах традиционного овцеводства на Северном Кавказе, приближенным к рынкам сбыта мясной овцеводческой продукции, в т.ч. и за пределы Российской Федерации на Ближний и Средний Восток [49].
Для наращивания объёмов производства баранины необходимо использовать все доступные средства: организационные, экономические, зоотехнические и т.д. Среди зоотехнических ведущая роль принадлежит селекции [1].
В настоящее время в животноводстве все большую популярность приобретают ДНК-маркеры, основанные на генах, белковый продукт которых играет значительную роль в формировании или регуляции некоторых физиологических процессов. Сам ген при этом должен обладать различными аллельными вариантами (полиморфизмом), которые связаны с вариативностью уровня продуктивности. «Считывание» этих вариантов и выявление желательных комбинаций позволяет проводить селекцию животных по генотипам [24, 41, 46, 99].
Степень влияния и уровень информативности генов-маркеров зависит от генотипической конструкции популяций («групповой генотип» по Д. Кисловскому). Внедрение в селекционный процесс этих маркеров должно непременно сопровождаться изучением взаимосвязи генотипов с продуктивными качествами на фоне существующего «группового генотипа» популяций, т.е. необходим синтез традиционных и молекулярно-генетических методов [65-66].
Молекулярно-генетические технологии (ДНК-маркеры) являются революционными методами, которые позволяют в десятки раз уменьшить затраты и ускорить темпы селекции. Дезоксирибонуклеиновая кислота рассматривается как главная информационная макромолекула, в структуре которой закодирован весь план развития организма. Интенсивное развитие молекулярной биологии и генетики приводят к появлению методов, позволяющих «считывать» информацию и моделировать линейные генетические конструкции, с учетом их сочетаемости и заданным уровнем продуктивности гибридного поголовья [90].
Полногеномные исследования.
В настоящее время молекулярно-генетические исследования завоевывают все большую популярность в работах, направленных на изучение сельскохозяйственных животных. Это связано с постоянным развитием и совершенствованием методом, которые позволяют решать различные задачи (популяционно-генетический анализ; оценка генетического разнообразия; филогенетические связи; поиск значимых вариантов, ассоциированных с селекционными признаками, поиск «подписей селекции»; определение протяженных гомозиготных областей и т.д.). Реализация этих методов осуществляется посредством считывания нуклеотидных последовательностей. Условно можно разделить методы на позволяющие получить полную нуклеотидную последовательность определенных фрагментов, либо всего генома или методы, основанные на считывании отдельных вариантов генома (от 10 тыс. SNP и более), рандомно расположенные по всему геному.
Один из новых методов, но уже широко используемых, является NGS – секвенирование нового поколения (Next Generation Sequencing), который пришел на смену «метода терминации растущей цепи», предложенного Сэнгером (1977). Метод Сэнгера позволяет «считывать» нуклеотидные последовательности, обладает высокой точностью, но низкой производительность. Следует отметить, что он остаётся достаточно востребованным для получения данных о небольших фрагментах ДНК. В свою очередь технологии NGS значительно сокращают время исследования, позволяя обрабатывать за день до одного миллиарда нуклеотидов. В качестве предшествующего метода, который послужил основой для NGS — секвенирования нового поколения (называемого также массивным или высокопроизводительным параллельным севенированием) можно выделить метод «дробовика» (shotgun sequencing), разработанный в рамках проекта «Геном человека».
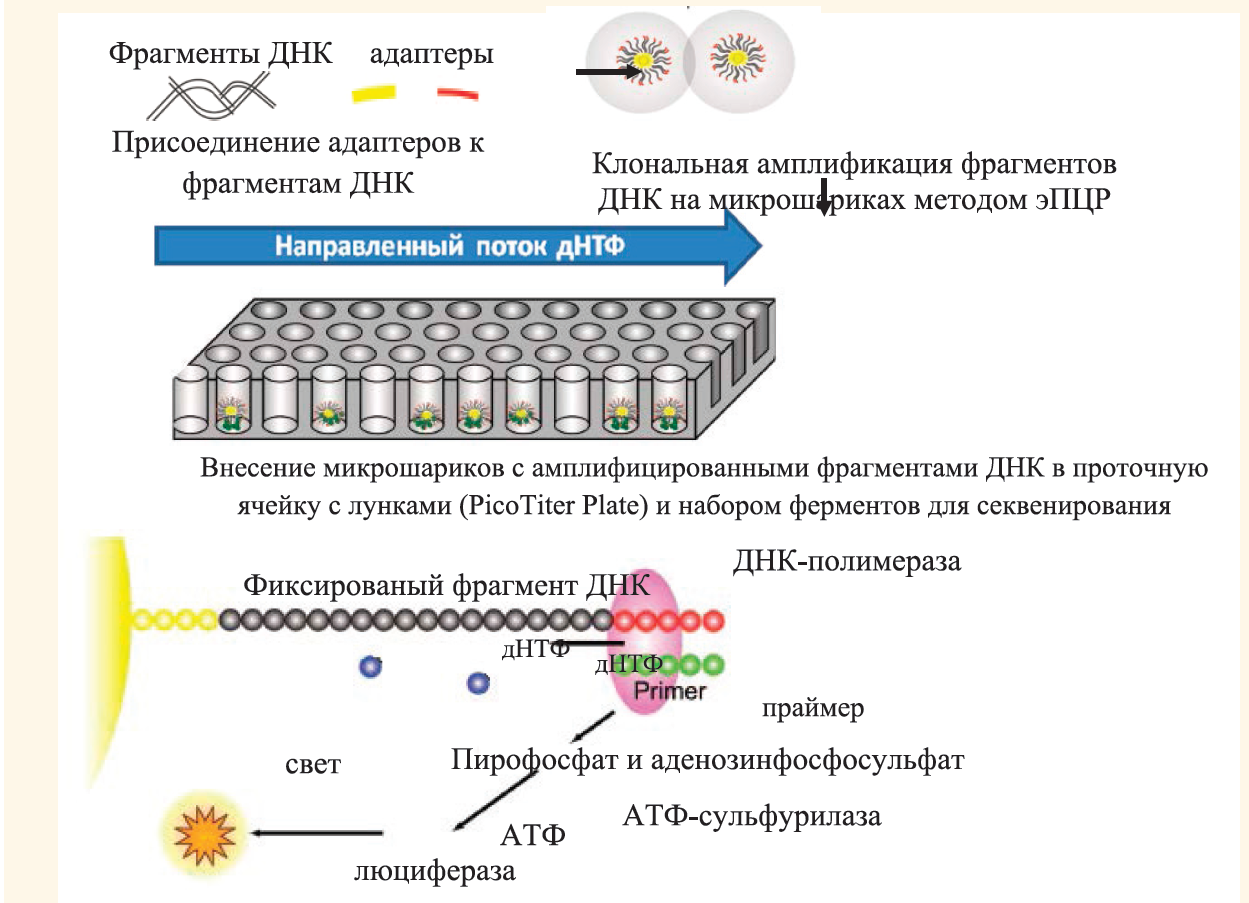
Рисунок 1 – Процесс секвенирования на платформах NGS [2]
Процесс секвенирования на платформах NGS можно разделить на три этапа:
- Подготовка библиотек, осуществляется путем фрагментирования ДНК (ферментативным методом или при помощи ультразвука), присоединения адаптеров (имеют известную нуклеотидную последовательность) и индексов;
- Амплификация полученных фрагментов методом ПЦР (полимеразной цепной реакцией);
- Считывания нуклеотидов изучаемой последовательности и детекция результатов.
На рынке сегодня представлены платформы NGS производителей (Roche, Швейцария; Illumina, США; Life Technologies, США), в которых заложены различные подходы к секвенированию: путем синтеза (Sequencing by Synthesis) или лигирования (Sequencing by Oligonucleotide Ligation and Detection).
На платформах NGS производства Illumina (США) применяют подход к секвенированию путем синтеза, используя флуоресцентную технологию. Данная технология основана на получении сигнала, поступающего от ДНК-полимеразы в момент присоединения одного из четырех нуклеотидов, имеющих флуорофоры. Присутствие флуоресцентной метки позволяет идентифицировать нуклеотид, после чего происходит его обработка реактивами для удаления этих меток и запускается новый цикл считывания.
Кластерная амплификация реализуется «методом моста» («bridge amplification»), используя универсальный праймер, специфичный последовательности адаптера. В результате на поверхности чипа образуются кластеры из фрагментов ДНК (рис.2).
Среди приборов, позволяющих реализовать данную технологию, можно отметить секвенаторы HiSeq и MiSeq, которые были выпущены на рынок компанией Illumina (США) в 2011 г. На платформе HiSeq можно секвенировать фрагменты длиной 100 нуклеотидов и получать до 600 Гб информации за пробег. MiSeq позволяет считывать фрагменты длиной до 250 нуклеотидов и получать данные 8,5 Гб/день.
Рисунок 2 – Схема флуоресцентной технологии секвенирования [2]
Сиквенсы нуклеотидных последовательностей, полученные на платформе NGS, представлены в виде небольших фрагментов, длиной около 100-400 нуклеотидов. Соответственно, очень значимым является степень покрытия (coverage) исследуемой последовательности (гена, генома и т.д.). Покрытие – это количество считывания одного нуклеотида в этой последовательности, при этом обязательным условием является то, чтобы фрагменты перекрывались друг с другом. Например, 50-кратное покрытие гена GH означает, что каждый нуклеотид был считан минимум 50 раз и представлен в различных перекрывающихся фрагментах [2].
Для анализа полученных результатов (ассоциативных, филогенетических и т.д.) необходимо провести обработку полученных данных. Этапами первичной обработки сиквенсов являются:
1) фильтрация последовательностей;
2) корректировка ошибок;
3) выравнивание последовательностей;
4) объединение фрагментов в единую последовательность.
Фильтрация позволяет определить и убрать последовательности, имеющие низкое качество прочтения. Для оценки качества используют показатель Phred Quality Scores (Q) (табл.1). Например, значение Q50 означает, что возможность некорректного определения нуклеотида составляет один из 100 000, а достоверность полученных результатов 99,999%.
Таблица 1 – Значения показателя Q для оценки качества секвенирования
| Phred Quality Scores (Q) | Возможность некорректного определения нуклеотида | Достоверность полученных сиквенсов (%) |
| 10 | 1 из 10 | 90,000 |
| 20 | 1 из 100 | 99,000 |
| 30 | 1 из 1 000 | 99,900 |
| 40 | 1 из 10 000 | 99,990 |
| 50 | 1 из 100 000 | 99,999 |
Следует отметить, что для различных платформ NGS свойственны определенные ошибки. Так, для платформ Illumina характерны ошибки в виде замен.
Последующий этап, выравнивание, позволяет собрать короткие нуклеотидные фрагменты в одну или несколько общих последовательностей. Для этого этапа могут быть использованы различные программы, большинство из которых основаны на алгоритмах перекрытия (overlap-layout-consensus) или на графах де Брейна (de Bruijn Graph). Для выравнивания и объединения могут быть использованы референсные нуклеотидные последовательности. Для коротких прочтений хорошие результаты показывают такие программы, как Clustal, MAQ (Mapping and Assembly with Quality), ELAND (Efficient Large-Scale Alignment of Nucleotide Databases) (www. illumina.com), BLAST (Basic Local Alignment Search Tool), SOAP (Short Oligonucleotide Alignment Program), SeqMap, MUSCLE, MAFFT и др.
По сравнению со сборкой коротких последовательностей на основе референса, выравнивание de novo является более сложным процессом, которое представляет собой реконструкцию в чистой форме, без сопоставления с ранее полученными последовательностями геномов или транскриптов. Для объединения нуклеотидных последовательностей de novo используются такие программные обеспечения, как SOAP de novo, Velvet, Euler и др.
Третий этап обработки данных секвенирования – непосредственный анализ набора последовательностей ДНК, полученного после объединения. В основе анализа набора полученных нуклеотидных последовательностей лежит определение филогенетических связей с уже известными нуклеотидными последовательностями целого генома или участками генома, относящимися к различным таксонам. Для этих целей используется программное обеспечение RITA, UniFrac, Naïve Bayes, BLAST, CARMA. TreePhyler, MetaDomain, Markov model, MEGAN, TOCOA и др. Источниками нуклеотидных последовательностей служат базы данных: GenBank (www.ncbi.nlm.nih.gov/genbank/), DNA Data Bank of Japan (www.ddbj.nig. ac.jp), GreenGens (www.greengenes.secondgenome.com), Genomes Online Database (www.genomesonline.org), Ribosomal Database Project (www.rdp.cme.msu.edu), SILVA (www.arb-silva.de) и др [87, 91, 95-96, 103].
При анализе результатов секвенирования проводятся множественные сравнения полученных нуклеотидных последовательностей с целью выявления изменчивости в виде однонуклеотидных полиморфизмов (single nucleotide polymorphism – SNP), инсерций, делеций и т. д. Для проведения исследований на основе генотипирования большого числа SNP, расположенных в изучаемом геноме, более доступными решениями являются SNP-панели (биочипы). SNP-панели были разработаны для включения высокого (HD), среднего (MD) или низкого (LD) распределения маркеров по геному. В отличие от данных секвенирования, панели генотипирования SNP обладают более точными результатами. В качестве недостатков отмечают наличие жесткой структуры, что позволяет анализировать только то, что уже предопределено конструкцией чипа, и в связи с этим можно пропустить потенциально важную информацию. Маркеры не имеют одинаковой плотности на всех хромосомах и не в полной мере отслеживают структурные генетические вариации, например вставки и делеции. Однако, несмотря на эти недостатки, на сегодняшний день SNP-панели завоевали большую популярность в исследованиях генетической архитектуры количественных признаков сельскохозяйственных животных [2].
llumina BeadChip System предлагает современную технологию микрочипов, способную как к геному, так и к индивидуальному генотипированию и экспрессии генов для человека, мыши и крысы, основных видов сельскохозяйственных животных и других организмов. С самого начала развития молекулярно-генетических исследований генотипирование было сильной стороной, что нашло отражение в многочисленных публикациях. В настоящее время фиксированный набор геномных SNP-маркеров остается привлекательным для многих исследовательских направлений. Illumina, как компания, впервые начала коммерческую деятельность в 2002 году и была основана на современной технологии BeadArray. Первые предложения, которые компания предоставила, были в качестве «нестандартной» платформы генотипирования с использованием теста GoldenGate ® . Хотя этот анализ способен мультиплексировать 1536 SNP в одном анализе, он не способен охватить весь геном. Несмотря на это, технология оценивается как обеспечивающая примерно 70% данных HapMap фазы 1. Сопоставление этих данных показало, что эта технология не только популярна у генетиков, но и является надежной, скорость вызовов и воспроизводимость которой превышают 99%. Впоследствии Illumina разработала анализ Infinium TM, позволяющий осуществлять типирование SNP по всему геному. В 2003 году она расширила возможности системы за счет экспрессии генов с теми же форматами Sentrix BeadChips и Array Matrix. В 2006 году Illumina добавила Rat в Human and Mouse в качестве продуктов с широким ассортиментом генома. Сейчас Illumina входит в состав Affymetrix и является одним из мировых лидеров в области коммерческих микрочипов.
Технология BeadArray от Illumina состоит в том, что для анализа используют гранулы размером 2 или 3 микрона (в зависимости от того, являются ли чипы стандартными или в формате HD), которые покрыты специфическими ДНК — репортерами. Одной из уникальных особенностей этой технологии является случайная сборка пула шариков. (рис.3).
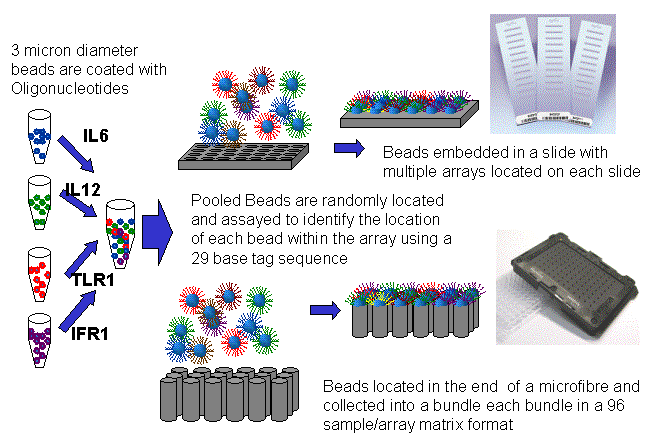
Рисунок 3 – Технология BeadArray от Illumina
Illumina разработала собственное программное обеспечение для генотипирования SNP в массиве BeadChip. Программное обеспечение называется GenCall. Алгоритм GenCall был реализован в приложении BeadStudio (последняя версия v3.2.2), но теперь он является частью приложения GenomeStudio (текущая версия 1.1.0). Он основан на определенной нормализации, которая происходит автоматически в программном обеспечении Illumina GenomeStudio и состоит из нескольких этапов (включая удаление выбросов и оценку фона) [111].
Вызов выполняется с использованием файла кластера, предоставленного Illumina, на основе эталонного набора образцов. Нужно ли использовать эталонный набор для вызова генотипа, зависит от количества выбранных массивов, качества ДНК и минимальной частоты аллелей (MAF), представляющих интерес.
Для SNP с менее чем тремя кластерами генотипов местоположение и вариации отсутствующих генотипов оценивают с использованием искусственных нейронных сетей. Также можно вручную изменить вызов любого SNP, используя инструмент визуализации Illumina. Для анализа CNV компания Illumina разработала серию инструментов, которые доступны в виде плагинов для модуля генотипирования GenomeStudio. Также доступно программное обеспечение для оценки количества копий (CNV), обнаружения и аннотации гомозиготности в отдельных образцах (Homozygosity Detector) обнаружения и аннотирования хромосомных аберраций в отдельных образцах (ChromoZone) и для расчета показателя вероятности для силы потери гетерозиготность (показатель LOH) [97].

Рисунок 4 – OvineSNP50 BeadChip
Для исследования ядерного генома овец хорошим инструментом является OvineSNP50 BeadChip (рис. 4), который был разработан Illumina в сотрудничестве с Международным консорциумом генома овец (ISGC), в состав которого входят ведущие исследователи из AgResearch, Baylor, UCSC и Австралийской государственной организации научных и прикладных исследований (CSIRO). На чипе расположено 54 000 SNP, которые равномерно охватывают весь геном овцы.
Для выбора SNP были применены такие критерии как частота минорного аллеля (MAF), количество аллелей, показатели качества, хромосомный интервал и местоположение. Эти требования привели к разработке массива высокой плотности, содержащий 54 241 SNP со средним размером 42,5 — 50,9 Кб (рис. 5). Этот уровень охвата обеспечивает превосходную плотность SNP для мощных исследований ассоциаций генома и обнаружения CNV у овец. Содержимое BeadChip включает покрытие аутосомных, митохондриальных и связанных с полом (X / Y) регионов.
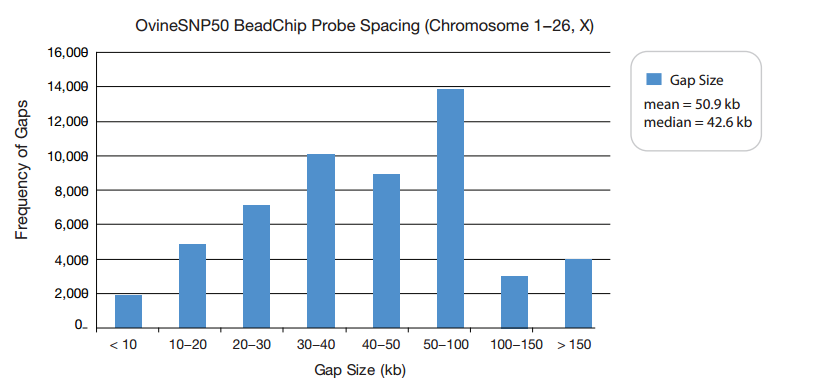
Рисунок 5 – Расстояние между SNP в OvineSNP50 BeadChip
1 Полногеномные исследования овец волгоградской породы
Выбор и обоснование направления исследования
Для исследования генетической архитектуры воспроизводительных признаков овец большие надежды возлагают на полногеномный поиск ассоциаций (GWAS, genome-wide association studies) [97]. Результатом GWAS может быть информация об обнаруженных ассоциациях с различными генетическими аберрациями – хромосомными мутациями (целые хромосомы или их фрагменты), крупными вставками или делециями (100 – 100 000 нуклеотидов), небольшими вставками или делециями (от 1 до 100), единичными нуклеотидными заменами (SNP, single nucleotide polymorphism) [100]. Каждый SNP представлен как минимум двумя аллелями – минорным (более редким) и мажорным (с частотой более 50% в популяции). Те генетические вариации, у которых частота минорного аллеля более чем 0,01%, пронумерованы и им присвоен индекс rs.
Воспроизводительная продуктивность обусловлена сложным комплексом признаков. В овцеводстве плодовитость овцематки (количество рожденных ягнят за окот) является главными показателями воспроизводительной продуктивности. Эти признаки отражают уровень всех физиологических процессов, связанных с оплодотворением, внутриутробным развитием плодов и родовой деятельностью, а также достаточно просто поддаются учету.
Цели и задачи исследований
Целью работы является проведение полногеномного генотипирования овец волгоградской породы на платформе OvineSNP50 Genotyping BeadChip и поиск SNP, ассоциированных с плодовитостью.
Для реализации цели были поставлены следующие задачи:
1. Проведение генотипирования овец на платформе OvineSNP50 Genotyping BeadChip
2. Обработка первичных данных
3. Фильтрация генотипов по показателю Call Rate (качество генотипирования)
4. Формирование lgen (генотипов) и fam (половая принадлежность исследуемых животных) файлов
5. Формирование ped (родословная) и map (карта хромосом) файлов
6. Фильтрация данных по уравнению Харди-Вайнберга и отсутствующих аллелей
7. Фильтрация вариантов с незначительной частотой аллеля (MAF) ниже заданного порога
8. Проведение ассоциативного анализа генотипов с фенотипами (GWAS) по классическим методам qt-means (с указанием средств описания фенотипов и стандартных отклонений, распределенных по генотипу) и linear (линейная регрессия)
9. Визуализация ассоциативного анализа построением Manhattan (шкалирование SNP по хромосомам) и qq- plot (график квантилей)
10. Отсечение значимых SNP в геноме по критерию Бонферрони в обратном логарифмическом масштабе
11. Корректировка ассоциативного анализа генотипов с фенотипами более точными математическими методами (LASSO, ridge regression, Bayes)
12. Идентификация генов, которые соответствуют дислокациям значимых SNP на базе программного обеспечения Ensembl
13. Проведение генной онтологии (GO) с целью функционального анализа идентифицированных генов на базе программного обеспечения PANTHER
Материалы и методы исследований
Для работы были выбраны 48 гол. овец волгоградской породы, ранжированные по второму окоту (24 гол. имели одного ягненка, а 24 гол. имели двух ягнят). Для проведения генотипирования овец на платформе OvineSNP50 Genotyping BeadChip были отобраны образцы ткани (ушные выщипы) у овец. Для выделения ДНК использовали набор «К-Сорб-100» (ООО «Синтол») в соответствии с инструкцией производителя.
В соответствии с поставленными задачами проведены предварительная обработка полученных генотипированных данных 48 голов овец волгоградской породы и ассоциативный анализ полиморфизма генов овец с количеством ягнят за два окота.
Результаты исследований
Фильтрация генотипов по показателю Call Rate. С помощью программного пакета Genome Studio 2 с параметрами Sample Name, Sample Group, Sample Index, Sample ID, SNP, SNP Name, SNP Index, SNP Aux, Allele1-Top, Allele2-Top, Allele1-Forward, Allele2-Forward, Allele1-Design, Allele2-Design, Allele1-AB, Allele2-AB, Allele1-Plus, Allele2-Plus, Chr, Position, GT Score, GC Score, Theta, R, X, Y, X-Raw, Y-Raw, B Allele Frequence, Log R Ratio, ILMN Strand, Customer Strand и с частотой вызова (Call Rate) равной 0,95, которая соответствует качеству генотипирования, были отобраны 54241 Single-nucleotide polymorphism (SNP).
Формирование lgen (генотипов) и fam (половая принадлежность исследуемых животных) файлов. Далее, на основе первичной обработки полученных данных на базе программного обеспечения Plink и R-Studio сформированы файлы lgen и fam. Файл lgen содержит информацию о связанном списке аллелей и является файлом длинного формата Plink для загрузки, а также текстовым файлом без заголовка и обычно со следующими пятью полями: индивидуальный номер (ID) животного, внутри-семейный ID, первого и второго аллелей (‘0’ если отсутствует), файл fam является текстовым файлом без заголовка со следующими шестью полями: принадлежность к породе («FID»), индивидуальный номер («ID»), внутрисемейный идентификатор отца («0», если отца нет в наборе данных), внутрисемейный идентификатор матери (‘0’, если мать отсутствует в наборе данных), пол («1» = самец, «2» = самка, «0» = неизвестно), значение фенотипа («1» = контроль, «2» = регистр, «-9» / «0» / не числовой = пропущенные данные, если регистр / контроль). Если имеются какие-либо числовые значения фенотипа, отличные от {-9, 0, 1, 2}, фенотип интерпретируется как количественный признак, а не как случай / контрольный статус. В этом случае -9 обычно обозначает отсутствующий фенотип.
Формирование ped (родословная) и map (карта хромосом) файлов. На следующем этапе были сформированы ped и map файлы. Первый файл является оригинальным стандартным текстовым форматом, содержащий информацию о родословной и генотипов. Обычно должен сопровождаться файлом map. Первые шесть полей такие же, как в файле fam. Седьмое и восьмое поля содержат аллели для первого варианта в файле .map (‘0’ = нет вызова); 9 и 10 — аллели ко второму варианту и так далее. Файл map является текстовым файлом без заголовка со следующими 3-4 полями: код хромосомы, идентификационный номер, позиция хромосомы в морганах или сантиморганах и координаты базовой пары.
Фильтрация данных по уравнению Харди-Вайнберга, отсутствующих аллелей и вариантов с незначительной частотой аллеля. Для дальнейшей обработки данных произведена их фильтрация с помощью параметров —hwe, —maf, —geno с соответствующими значениями. Фильтр —hwe позволяет удалить варианты, не удовлетворяющие условию точного теста равновесия Харди-Вайнберга согласно установленному порогу, —maf — отфильтровывает все варианты с меньшей частотой минорного аллеля ниже установленного порога (по умолчанию 0,01), geno — отфильтровывает все варианты, в которых количество пропущенных вызовов превышает заданное значение (по умолчанию 0.1), подлежащие удалению. В наших исследованиях были установлены пороги —hwe —maf 0,5; —geno 0,1.
В результате проведенных процедур с учетом вышеуказанных фильтров и удаление пустых ячеек, из 54241 остались 46249 SNP, причем 1680 удалены фильтром —geno, 2652 удалены фильтром –maf.
Проведение ассоциативного анализа генотипов с фенотипами. Из формата ped данные были переформатированы в бинарный файл bed, далее была применена команда —indep-pairwise 50 5 0.5. Эта команда создает обрезанное подмножество маркеров, которые находятся в приблизительном равновесии связей друг с другом и они основаны на корреляции между количеством генотипических аллелей. Для параметра «indep» требуется три параметра: размер окна в единицах подсчета вариантов или килобазы (если модификатор ‘kb’ присутствует), подсчет вариантов для перемещения окна в конце каждого шага и порог инфляции коэффициента дисперсии (VIF). На каждом шагу удаляются все варианты в текущем окне с превышением порога VIF. В качестве третьего параметра используется парный порог r2: на каждом шагу отмечаются пары вариантов в текущем окне с квадратной корреляцией больше порога, и из окна вырезаются варианты до тех пор, пока такие пары не исчезнут. Так как нет необходимости хранить всю матрицу корреляции <размер окна> x <размер окна> в памяти, она обычно способна обрабатывать 6-значные размеры окна вне досягаемости —indep.
Визуализация ассоциативного анализа построением Manhattan- (шкалирование SNP по хромосомам) и qq- plot (график квантилей). На рисунке 6 графически методом Manhattan plot представлен результат ассоциативного анализа связи SNP с количеством ягнят для двух окотов. Каждая точка Манхэттен графика представляет собой SNP, выложенную по хромосомам животного, представленным на оси X слева направо, в то время как высоты по оси Y соответствуют силе ассоциации с изучаемым фактором, выраженной логарифмически.
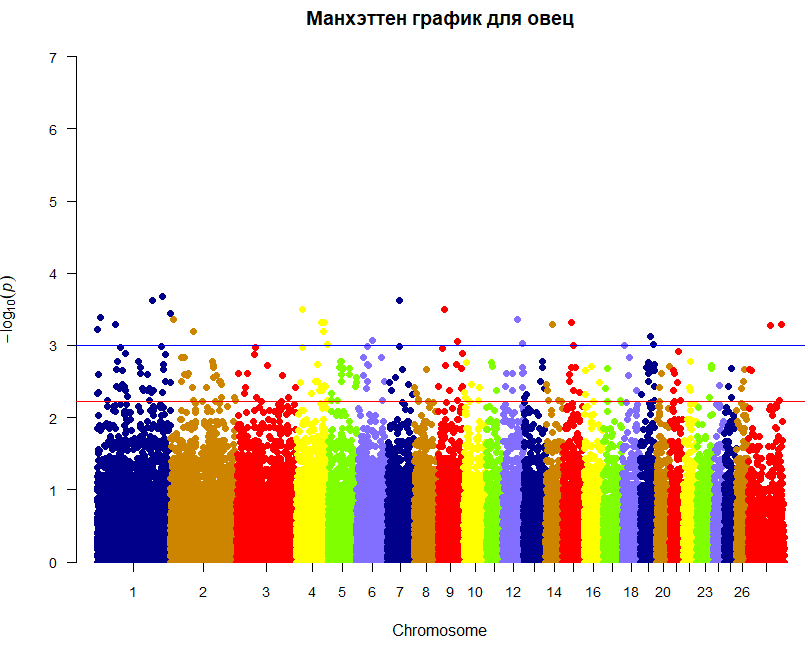
Рисунок 6 – Manhattan plot по результатам ассоциативных исследований
Самые сильные ассоциации образуют аккуратные пики, где близлежащие коррелированные SNP показывают один и тот же сигнал. Из графика видно, что наиболее достоверными являются SNP расположенные в хромосомах 1, 4,7 и 9.
Другим не менее важным визуальным результатом ассоциативного анализа является график квантилей QQ-график (рис.7). QQ-график p-значений показывает, отличается ли очень небольшое p-значение от ожидаемого распределения p-значений (равномерного распределения). Если значение p отклоняется от ожидаемого распределения, то его можно считать статистически значимым. На рисунке 7 видно, что р-значения, отклоняющиеся от ожидаемых значений, для определенного количества SNP являются статистически значимыми.
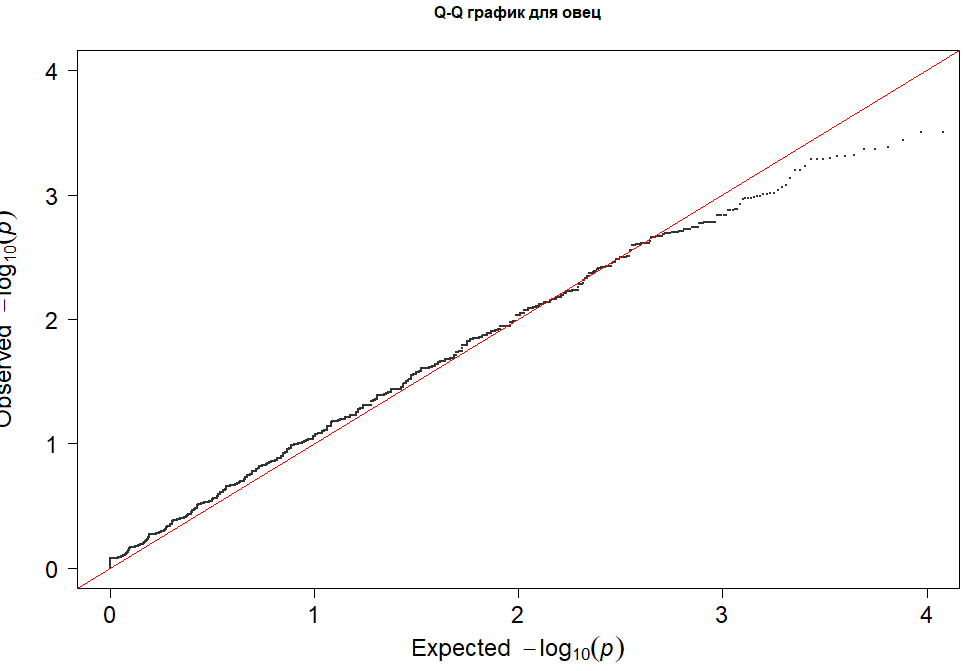
Рисунок 7 – QQ график. На оси Х расположены ожидаемые значения, на оси У- наблюдаемые.
На графике QQ показано ожидаемое распределение статистики тестирования ассоциации (ось X) по тысячам SNP по сравнению с наблюдаемыми значениями (ось Y). Любое отклонение от линии X = Y подразумевает постоянную разницу между случаями и контролем во всем геноме. С другой стороны, чистый график QQ должен показывать сплошную линию, соответствующую X = Y, до тех пор, пока она не будет резко кривыми в конце (что представляет собой небольшое число истинных ассоциаций среди тысяч непривязанных SNP).
Корректировка ассоциативного анализа генотипов с фенотипами более точными математическими методами (LASSO, ridge regression, Bayes). Для обеспечения надежности и эффективности оценок параметров регрессии, можно использовать методы регуляризации, которые позволяют «корректировку» отклонений от нормального распределения остатков. При нахождении оценок параметров модели методом МНК к функции потерь: 
подлежащей минимизации функции следует добавить дополнительный член, зависящий от этих параметров. Т.е. вместо RSS минимизируется выражение:

или

где ![]() коеффициенты уравнений.
коеффициенты уравнений.
Гребневая регрессия (ridge regression) реализует классическую L2-регуляризацию Тихонова, при которой оценки параметров модели ![]() находят из условия минимизации (3). При
находят из условия минимизации (3). При ![]() регуляризованное решение стремится к МНК-решению, т.е. обычной линейной модели. При увеличении
регуляризованное решение стремится к МНК-решению, т.е. обычной линейной модели. При увеличении ![]() вектор коэффициент
вектор коэффициент ![]() становится всё более устойчивым и жёстко определённым (фактически, происходит понижение эффективной размерности решения). При → ∞ чрезмерная регуляризация приводит к вырожденному решению:
становится всё более устойчивым и жёстко определённым (фактически, происходит понижение эффективной размерности решения). При → ∞ чрезмерная регуляризация приводит к вырожденному решению: ![]() → 0. Оба крайних случая нежелательны, поэтому оптимальным является некоторое промежуточное значение
→ 0. Оба крайних случая нежелательны, поэтому оптимальным является некоторое промежуточное значение ![]() .
.
Метод регрессии «лассо» (LASSO, от англ. Least Absolute Shrinkage and Selection Operator) внешне похож на гребневую регрессию, т.е. оценки параметров модели ![]() также находят из условия минимизации функции (2). В таблице 2 сгруппированы наиболее значимые 40 SNP с соответствующими хромосомами и их позициями.
также находят из условия минимизации функции (2). В таблице 2 сгруппированы наиболее значимые 40 SNP с соответствующими хромосомами и их позициями.
Таблица 2 – Значимые SNP, ассоциированные с плодовитостью овец
| CHR | SNP | BP | A1 | F_A | F_U | A2 | CHISQ | P | OR |
| 1 | s47846.1 | 243201901 | A | 0.375 | 0.0625 | G | 13.71 | 0.000213 | 9 |
| 1 | OAR1_224337515.1 | 207635268 | G | 0.6667 | 0.2917 | A | 13.52 | 0.000236 | 4.857 |
| 7 | s58843.1 | 45816558 | A | 0.2917 | 0.6667 | G | 13.52 | 0.000236 | 0.2059 |
| 9 | OAR9_25927499.1 | 24882070 | G | 0.5417 | 0.1875 | A | 12.99 | 0.000312 | 5.121 |
| 4 | OAR4_24841202.1 | 23675881 | G | 0.06522 | 0.375 | A | 12.99 | 0.000313 | 0.1163 |
| 1 | OAR1_297010151.1 | 274211714 | G | 0.5625 | 0.2083 | A | 12.71 | 0.000364 | 4.886 |
| 1 | s44128.1 | 11136653 | G | 0.2292 | 0.5833 | A | 12.48 | 0.000411 | 0.2124 |
| 2 | OAR2_12233770.1 | 12893239 | A | 0.0625 | 0.3542 | G | 12.38 | 0.000434 | 0.1216 |
| 12 | s00442.1 | 54910458 | A | 0.0625 | 0.3542 | G | 12.38 | 0.000434 | 0.1216 |
| 4 | OAR4_98736801.1 | 93098952 | G | 0.625 | 0.2708 | A | 12.17 | 0.000485 | 4.487 |
| 4 | s09521.1 | 105511711 | G | 0.1042 | 0.4167 | A | 12.17 | 0.000486 | 0.1628 |
| 15 | s21666.1 | 33494554 | A | 0.4167 | 0.1042 | G | 12.17 | 0.000486 | 6.143 |
| 27 | s34139.1 | 123804584 | C | 0.3125 | 0.04167 | A | 12.08 | 0.00051 | 10.45 |
| 1 | s72471.1 | 69968097 | C | 0.6667 | 0.3125 | G | 12.05 | 0.000519 | 4.4 |
| 14 | s37599.1 | 25825863 | A | 0.3125 | 0.6667 | G | 12.05 | 0.000519 | 0.2273 |
| 27 | OARX_102576796.1 | 82685690 | A | 0.02083 | 0.2708 | G | 12.04 | 0.00052 | 0.05728 |
Продолжение таблицы 2
| CHR | SNP | BP | A1 | F_A | F_U | A2 | CHISQ | P | OR |
| 1 | s68493.1 | 775181 | G | 0.4565 | 0.1304 | A | 11.79 | 0.000594 | 5.6 |
| 2 | s52970.1 | 86438687 | G | 0.1875 | 0.5208 | A | 11.66 | 0.000639 | 0.2123 |
| 4 | s16058.1 | 100806043 | A | 0.1875 | 0.5208 | C | 11.66 | 0.000639 | 0.2123 |
| 19 | OAR19_39947329_X.1 | 38107817 | G | 0.5417 | 0.2083 | A | 11.38 | 0.000743 | 4.491 |
| 6 | OAR6_67964178.1 | 61757917 | G | 0.5625 | 0.2292 | A | 11.15 | 0.00084 | 4.325 |
| 9 | OAR9_77977112.1 | 73410452 | G | 0.0625 | 0.3333 | A | 11.09 | 0.000868 | 0.1333 |
| 12 | OAR12_81266260.1 | 73679900 | A | 0.5833 | 0.25 | G | 10.97 | 0.000925 | 4.2 |
| 4 | OAR4_124681944.1 | 116783114 | A | 0.1042 | 0.3958 | G | 10.89 | 0.000967 | 0.1775 |
| 19 | s45366.1 | 48104614 | G | 0.3958 | 0.1042 | A | 10.89 | 0.000967 | 5.634 |
| 15 | s65298.1 | 39953524 | C | 0.6042 | 0.2708 | A | 10.84 | 0.000996 | 4.109 |
| 18 | s74170.1 | 7227477 | A | 0.2708 | 0.6042 | G | 10.84 | 0.000996 | 0.2433 |
| 1 | OAR1_260711291.1 | 241022280 | A | 0.02083 | 0.25 | G | 10.77 | 0.001034 | 0.06383 |
| 7 | OAR7_48227587.1 | 43512379 | G | 0.02083 | 0.25 | A | 10.77 | 0.001034 | 0.06383 |
| 6 | OAR6_45997407.1 | 41220008 | A | 0.625 | 0.2917 | G | 10.74 | 0.001048 | 4.048 |
| 1 | OAR1_90720314.1 | 85318570 | G | 0.1667 | 0.4792 | A | 10.72 | 0.00106 | 0.2174 |
Продолжение таблицы 2
| 3 | OAR3_74471615.1 | 70316645 | G | 0.4792 | 0.1667 | A | 10.72 | 0.00106 | 4.6 |
| 4 | OAR4_24779248.1 | 23621163 | A | 0.1667 | 0.4792 | G | 10.72 | 0.00106 | 0.2174 |
| 9 | OAR9_16955020.1 | 16440142 | G | 0.3333 | 0.6667 | A | 10.67 | 0.001091 | 0.25 |
| 21 | OAR21_36019220.1 | 32346421 | A | 0.5227 | 0.1957 | G | 10.5 | 0.001194 | 4.503 |
| 1 | s21713.1 | 104807920 | G | 0.125 | 0.4167 | A | 10.34 | 0.001303 | 0.2 |
| 9 | OAR9_97113607.1 | 91173830 | G | 0.4167 | 0.125 | A | 10.34 | 0.001303 | 5 |
| 1 | OAR1_277749768.1 | 256797131 | G | 0.08333 | 0.3542 | A | 10.3 | 0.00133 | 0.1658 |
| 1 | OAR1_277830695.1 | 256880139 | G | 0.08333 | 0.3542 | A | 10.3 | 0.00133 | 0.1658 |
Идентификация генов, которые соответствуют дислокациям значимых SNP на базе программного обеспечения Ensembl. Дислокация значимых SNP была осуществлена с использованием программных продуктов базы Ensembl (https://www.ensembl.org/index.html). Ensembl — это браузер генома геномов позвоночных, который поддерживает исследования в области сравнительной геномики, эволюции, изменения последовательности и регуляции транскрипции. Ансамбль аннотирует гены, вычисляет множественные выравнивания, прогнозирует регуляторную функцию и собирает данные о заболеваниях. Инструменты Ensembl включают BLAST, BLAT, BioMart и Variant Effect Predictor (VEP) для всех поддерживаемых видов, в том числе овец.
На диаграмме представлено распределение идентифицированных значимых SNP овец относительно областей геном (рис. 8). Более 50% SNP локализованы в генах, остальные расположены в меженных регионах, а также в областях промоутеров и сплайсинга.
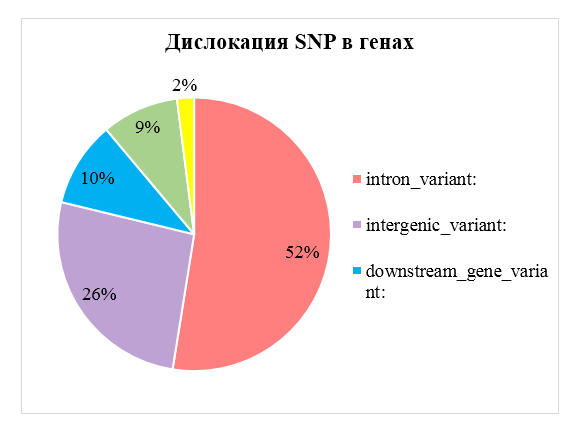
Рисунок 8. Распределение SNP по геному
Гены, в которых были локализованы выбранные SNP, были определены с использованием базы Ensembl и представлены в таблице 3.
Таблица 3 – SNP, локализованные в генах
| Номер хромосомы | Позиция | SNP | Ген |
| 1 | 70044230 | s72471.1 | BCAR3 |
| 1 | 85430810 | OAR1_90720314.1 | GPSM2 |
| 1 | 104919388 | s21713.1 | RHBG |
| 1 | 243372009 | s47846.1 | SLC9A9 |
| 1 | 257046798 | OAR1_277830695.1 | ATP2C1 |
| 15 | 40040411 | s65298.1 | MICAL2 |
| 18 | 7233159 | s74170.1 | PGPEP1L |
| 19 | 38093997 | OAR19_39947329_X.1 | SYNPR |
| 19 | 48091346 | s45366.1 | ITIH1 |
| 2 | 12846994 | OAR2_12233770.1 | SVEP1 |
| 4 | 23648876 | OAR4_24779248.1 | AGMO |
| 4 | 100824042 | s16058.1 | DGKI |
| 4 | 116829611 | OAR4_124681944.1 | RBM33 |
| 7 | 43534926 | OAR7_48227587.1 | USP3 |
В результате были идентифицированы 14 генов, которые были подвергнуты тщательному анализу с помощью базы Panther (http://www.pantherdb.org/). PANTHER (Protein ANalysis THrough Evolutionary Relationships) – этот система классификации белков (и их генов), которая обладает высокой пропускной способностью и позволяет определять:
— семейства и подсемейство белков: семейства — это группы эволюционно родственных белков; подсемейства — родственные белки, имеющие сходные функции);
— молекулярные функции: функции отдельных белков на биохимическом уровне, а так с учетом их взаимодействий с другими белками;
— биологический процесс: функция белка в контексте более широкой сети белков, которые взаимодействуют, чтобы выполнить процесс на уровне клетки или организма;
— путь: в некотором смысле это тот же биологический процесс, но с более четкой классификацией между взаимодействующими молекулами.
Результаты генной онтологии генов, в которых локализованы значимые SNP, ассоциированные с плодовитостью овец, представлены на рисунке 9. Согласно полученным результатам исследований большинство из этих генов принимают участие в клеточных процессах и биологической регуляции.
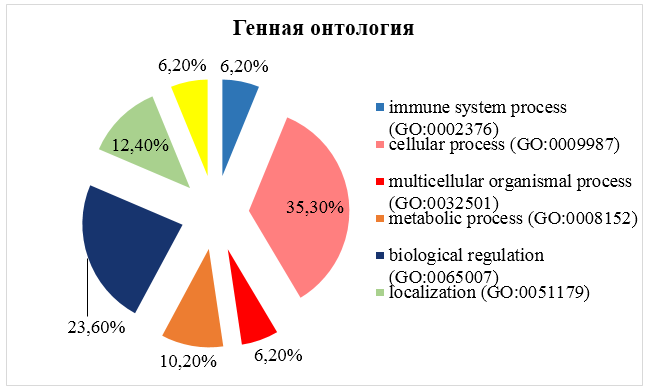
Рисунок 9. Результаты генной онтологии
Выводы
В результате проведенного полногеномного генотипирования овец волгоградской породы с использованием OvineSNP50 Genotyping BeadChip получены данные, характеризующие 54241 SNP, расположенные в геноме овец. По результатам анализа были определены 40 SNP, ассоциированных с плодовитостью овец, а также определены 14 генов, которые могут быть ответственны за признаки, связанные с воспроизводительной функцией.
Дальнейшие исследования (2020 г.) будут направлены на увеличение выборки овец для генотипирования (96 гол.). На основе полученных данных будут установлены гены, связанные с продуктивными качествами овец.
2 Изучение полиморфизма ДНК-маркеров и их влияния на показатели продуктивности овец отечественных пород
2.1 Воспроизводительные качества овец волгоградской породы различных генотипов гена GDF9
Выбор и обоснование направления исследования
В настоящее время возрастает интерес к технологиям, основанным на использовании ДНК-маркеров, которые находят широкое применение в национальных селекционных программах ряда стран с развитым овцеводством и оказывают значительное воздействие на улучшение состава туши, качество мяса и эффективность производства баранины. Значимым направлением практической генетики применительно к отрасли овцеводства, является маркерная селекция (Marker Assisted Selection — MAS), предусматривающая использование ДНК-маркеров, ассоциированных с уровнем проявления признаков продуктивности [101]. Эти методы позволяют значительно ускорить совершенствование мясной и воспроизводительной продуктивности овец, в том числе и мериносовых пород, что делает отрасль более конкурентоспособной [4, 26, 44, 47, 51].
Тонкорунная волгоградская порода овец мясо-шёрстного направления продуктивности, начала создаваться в — 1932 году, в Волгоградской области, на первом этапе методом сложного воспроизводительного скрещивания грубошерстных курдючных маток выбранных в качестве материнской линии с тонкорунными баранами пород новокавказской и прекос (тип суассоне), полученных в результате помесных маток в желательном типе в основном второго поколения, разводили в себе. Потомство от этих скрещиваний, в первую очередь не удовлетворяло по шерстной продуктивности. Поэтому одновременно с улучшением мясных качеств и скороспелости для улучшения показателей по настригу и качеству шерсти, полученных помесных маток с — 1948 года, начали скрещивать с баранами кавказской и в меньшей степени грозненской пород [15-16, 101].
Одной из главных задач, которая ставилась при создании Волгоградской породы овец, это получение породы мясного направления. В результате более чем сорокалетней селекции, в результате отбора и целенаправленного подбора к семидесятым годам был создан однородный массив овец нового типа, который в — 1978 году, был опробован и официально признан как новая, самостоятельная Волгоградская порода овец.
Тонкорунная волгоградская порода овец больше всего подходит для разведения в средней полосе России, наряду с такими уже традиционными породами как, романовская и куйбышевская. Волгоградские овцы получились очень правильных пропорций, крепкие, плотные с хорошими мясными формами с очень хорошим сочетанием мясных и шерстных показателей. Эта порода и по сей день остается очень выгодной и прибыльной отечественной породой мясошерстной направленности, несмотря на существенное снижениях спроса на шерсть после развала овцеводства в России, в постсоветское время [6, 12, 27, 30].
Представители данной породы имеют высоту в холке порядка — 68-70 см. при косой длине туловища — 70-73 см. Живая масса маток не менее — 58-65 кг, баранов — 110-125 кг. Волгоградские овцы достаточно скороспелые, живая масса ягнят к отбивке достигает — 30-35 кг, а к годовому возрасту ярки достигают до — 80% от массы взрослых овец. Баранчики в возрасте — 7-9 месяцев имеют чистый вес тушки в — 20-24 кг. Волгоградские овцы, как и большая часть баранов комолые, то есть безрогие. Овцы этой породы бесскладчатые, имеется лишь небольшой запас кожи на шее в виде фартука или бурды. Туловище овец компактное, длина туловища — 70-75см. Голова у Волгоградских овец крупная либо средней величины. Шея довольно массивная, заметно расширяющаяся к туловищу. Холка, спина, поясница широкие, спина ровная. Ноги крепкие, правильно поставленные, ляжки хорошо выполнены [18].
Шерсть у волгоградских овец белая, довольно густая. Уравненность шерсти вполне удовлетворительная. Руно штапельного строения, средней плотности, замкнутое. Наружный штапель преимущественно мелкоквадратной формы. Извитость шерстных волокон достаточно выраженная, равномерная, несколько растянутая. Тонина шерсти — 60 и 64 качества. Длина шерсти у овец — 8-9 см, у баранов — 9,5-10,5 см. Жиропот светлых тонов, в основном, светло-кремовый. Оброслость головы рунной шерсти вплоть до линии глаз, ног, до запястного и скакательного суставов.
Настриг шерсти у овец — 5,5-6,0 кг, у баранов не менее — 13-15 кг. Выход чистого волокна порядка — 48-50 %. Плодовитость у овец довольно хорошая — 130-160 %. Молочность составляет — 95-105 кг у маток с одним ягненком и — 146-150 кг у маток с двойнями [23, 25].
В настоящее время наиболее выгодно в экономическом плане разводить мясные и молочные породы овец, потому что баранина и ягнятина более востребована, чем шерсть. По этой причине волгоградская порода овец, как нельзя лучше подходит для тех овцеводов, которые специализируются на производстве мяса. Но несмотря на то, что порода больше ориентирована на мясную продукцию, шерстная продуктивность у волгоградских овец тоже находится на достаточно высоком уровне, бараны дают от — 13 до — 15 кг шерсти при настриге, а овцы около — 6 кг. Тем не менее у данной породы недостаточно высокое качество шерсти, она несколько суховата и не уравнена по тонине.
Помимо высоких мясных и молочных качеств, а также скороспелости, выделяют прекрасную адаптивность овец волгоградской породы к различным условиям содержания. Лучшие племенные стада сохранились именно в Волгоградской области, однако волгоградскую породу можно встретить по всей средней полосе России, например, на Урале и Поволжье [31, 36]. Для интенсификации овцеводства, обеспечения экономической эффективности и рентабельности отрасли, необходимо использовать современные молекулярно-генетические методы в селекции.
Стремительное развитие молекулярной генетики в последние два десятилетия, связанное, в первую очередь, с технологическим прорывом в области секвенирования геномов высших организмов, позволило уже сейчас применять на практике такие методы работы с биологическими объектами, о которых совсем недавно еще не приходилось и мечтать. Для большинства важнейших сельскохозяйственных видов созданы подробные генетические карты, на которые нанесены сотни молекулярно-генетических маркеров. Многие из этих данных находятся в свободном, публичном доступе, еще больше данных по генетическим маркерам можно получить на специально оговоренных условиях и для коммерческого использования. Этот последний факт свидетельствует сам за себя: генетические маркеры начинают все шире использовать в практике и, в первую очередь, с целью применения их как нового и многообещающего инструмента в селекционных программах [50. 56-57, 59].
Селекция на улучшение интересующего признака, базируется изначально на выборе генов, детерминирующих биохимические процессы, связанные с формированием признака. Затем исследуется полиморфизм этих генов и продуктивные особенности животных, несущих в своем генотипе разные аллели этого гена. Следовательно, гены-кандидаты – это гены, кодирующие ключевые белки, принимающие участие в формировании признака. Особенностью таких генов является то, что изначально неизвестно наличие аллельных вариантов гена и их влияние на величину признака. Существуют различные механизмы влияния аллельных генов на признаки. В то же время, по мнению многих авторов, маркерная селекция оказывается эффективной даже при отсутствии некоторых из них (например, плейотропии и сцепления). В любом случае, даже временные связи маркерных генов с хозяйственно полезными признаками могут быть использованы в племенной работе с конкретными популяциями животных и получен экономический эффект [53, 60, 62, 68].
Геном овцы был секвенирован в 2012 г (The International Sheep Genomics Consortium, 2012). Сочетание расшифрованного генома с высокоплотным SNP-чипом дает возможность значительно ускорить поиск значимого генетического полиморфизма, ассоциированного с продуктивными признаками овец [76, 88, 106, 116].
Ведение прямой селекции на плодовитость характеризуется относительно низкой эффективностью, что связано, с одной стороны, с недостаточно высокой наследуемостью признаков, с другой стороны, с ограниченным полом их проявления [61]. В связи с этим, исследования, направление на поиск и идентификацию ДНК-маркеров, ответственных за развитие этих признаков, является актуальными и востребованными [64].
У овец выделяют три главных гена, связанных с плодовитостью:
Ген дифференциального фактора роста (GDF9) расположен в 6 хромосоме. Белковый продукт гена играет важную роль для поддержания нормального яичникового фолликулогенеза у овец [88, 94, 104, 108].
Ген костных морфогенетических протеинов 15 (BMP15), известный как на FecX и расположенный на Х-хромосоме [114].
Ген BMPR1B — рецептор костных морфогенетических протеинов типа 1Б (также известный как ALK6, активин-подобные киназы 6), расположенный на 6 хромосоме овец. Его название в научной литературе часто встречается как FecB— Бурула ген (Booroola) [92].
Бурула — одна из небольшого числа мериносовых пород овец, с высокой генетической обусловленной способностью к высокой плодовитости. Порода представляет большую ценность в качестве генетического улучшателя (путем скрещивания) овец других мериносовых пород в отношении увеличения (на 40— 60%) их плодовитости. При традиционной внутрипородной селекции по этому признаку такой эффективности можно достичь примерно в течение 30 лет [54, 92].
Высокая плодовитость овец бурула — вероятно, следствие влияния одного гена (F), который воздействует на число овулируемых яйцеклеток из яичника за каждый цикл течки. Этот факт возродил интерес к изучению роли основных генов в развитии количественных признаков животных и к развитию методов их распознавания и использования.
Цель и задачи исследований
Целью работы стало исследование продуктивных особенностей овец волгоградской породы на основе вариабельности ядерной ДНК с целью повышения племенных и продуктивных качеств.
В связи с этим были поставлены следующие задачи:
- Провести молекулярно-генетический мониторинг овец волгоградской породы по гену GDF9
- выявить желательные генотипы, связанные с воспроизводительными качествами овец.
Материалы и методы исследования
Объектом исследования были овцы волгоградской породы (n=117) СПК ПЗ «Ромашковский», с. Ромашки, Волгоградской области. Для оценки продуктивных качеств использовали данные зоотехнического учета хозяйства животных 2012-2016 года рождения. Живую массу определяли путем взвешивания животных весной (возраст 8 мес.) и осенью (возраст 12 мес.). Воспроизводительную продуктивность маток оценивали по количеству и массе ягнят при рождении. Учитывали результаты по первому и второму окоту.
Молекулярно-генетические исследования, а также обработку полученных результатов, проводили в лаборатории молекулярной диагностики и биотехнологии с.-х. животных Донского ГАУ, пос. Персиановский. Для проведения исследований у овец волгоградской породы были отобраны образцы ткани с ушной раковины площадью 1 см². ДНК выделяли с применением набора реагентов DIAtom DNA Prep 100 (ООО «НПФ Генлаб»). Анализ проводили методом ПЦР-ПДРФ (полимеразной цепной реакции — полиморфизм длин рестрикционных фрагментов).
Для амплификации гена GDF9 использовали специальные олигонуклеотидные праймеры, предложенные Chu с соавторами (2011):
5´ — GAAGACTGGTATGGGGAAATG -3´
5´ — CCAATCTGCTCCTACACACCT-3´
Условия ПЦР: первоначальная денатурация – 2 мин при 940С; денатурация 940С — 30с, отжиг 630С — 40с, элонгация 720С – 30 с (35 циклов), завершающая элонгация при 720С 4 мин. Рестрикцию фрагмента гена GDF9 длиной 462 н.п. проводили с использованием эндонуклеазы BstHHl. Рестрикционные фрагменты разделяли в 2% агорозном геле, с добавлением бромистого этидия.
Результаты исследований
Процесс фолликулогенеза и оогенеза зависит от взаимосвязи между ооцитом и окружающими его соматическими клетками. В процессе фолликулогенеза происходит дифференциация соматических клеток, в результате чего образуются муральные клетки, выстилающие полость фолликула, и кумулюсные клетки, окружающие ооцит.
Формирование зрелого ооцит-кумулюсного комплекса происходит под действием лютеинизирующего гормона (LG), в этом блоке ооцит готов к овуляции и последующему оплодотворению. Окружающие ооцит соматические клетки и активация различных сигнальных путей влияет на рост и развитие фолликула, транскрипционную активность, регулируя таким образом ядерное и цитоплазматическое созревание ооцита.
Ооциты в процессе фолликулогенеза секретируют факторы, необходимые для роста окружающих фолликулярных клеток и регулируют дифференцировку, пролиферацию, апоптоз и лютеинизацию гранулезных и кумулюсных клеток. Одним из таких факторов является дифференциальный фактор роста -9 (GDF9), продуцируемый ооцитом в течение всего времени от формирования первичного фолликула до овуляции. На ряду с GDF9 в ооцитах секретируется костный морфогенетический белок-15 (BMP15). GDF9 и BMP15 относятся к суперсемейству трансформирующего фактора роста B (TGF-B) , являются синергистами и играют важную роль в развитии фолликуллогенеза у млекопитающих, т.к. являются необходимыми для нормального созревания ооцита и дальнейшего развития эмбриона, а при отсутствии данных факторов эмбрионы останавливаются в развитии до достижения ими стадии бластоцисты. GDF9 регулируют экспрессию ряда генов, участвующих в экспансии клеток кумулюса, в том числе гиалуронан-синтетазы 2 (HAS2), простагландин-синтетазы 2 (PTGS2) и гена гремлина (GREM1) [94, 100].
Впервые экспрессию гена GDF9 в ооцитах овец установил Bodensteiner et al. (2000) [79]. Позже, рядом ученых были проведены исследования in vivo экспрессии гена GDF9 и BMP15 в ооцитах и кумулюсных клетках на разных стадиях развития фолликулов. В результате была установлена экспрессия генов GDF9 и BMP15 в ооцитах и кумулюсных клетках на всех стадиях. Самая высокая экспрессия генов GDF и BMP15 (3,38 ± 0,02 и 2,69 ± 0,06 соответственно)установлена в примордиальных фолликулах, по сравнению с преантральными, антральными и преовуляторными фолликулами. Аналогично, экспрессия GDF9 и BMP15 в кумулюсных клетках (0 ± 0,16 и 0 ± 0,07) и ооциты (1,47 ± 0,07 и 1,32 ± 0,03) была самая низкая в антральных фолликулах [114].
Роль гена GDF9 в процессах фолликулогенеза, нормального созревания ооцита и развития эмбриона послужила поводом для исследования его полиморфизма и тестирования в качестве генетического маркера репродуктивных показателей с.-х. животных. У овец последовательность гена GDF9 (Gene ID: 100217402) cоставляет 2500 пар оснований и содержит 2 экзона и один интрон. Белковый продукт гена представлен 456 аминкислотами. Первый экзон составляют 397 пар оснований, кодирующих 134 аминокислоты, второй экзон – 968 п.о. кодирует 322 аминокислоты. В исследованиях Hanrahan с соавторами (2004) были представлены 8 полиморфных точек гена GDF9. В нашей работе мы изучали полиморфизм гена GDF9 у овец волгоградской породы, обусловленный нуклеотидной заменой во втором экзоне в точке G1(G260A) [77].
Результаты молекулярно-генетических исследований показали, что полиморфизм гена GDF9 в исследуемой популяции представлен двумя аллелями Aи Gи двумя генотипами AGи GG с частотами 8,55 и 91,45%. На электрофореграмме (рис.10) генотип АА представлен двумя фрагментами, размеры которых составляют 254- и 117 п.н., генотип AG – тремя фрагментами: 410-, 254 – и 117 п.н.
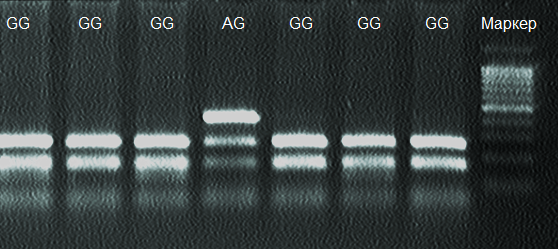
Рисунок 10 – Электрофореграмма результатов ПЦР-ПДРФ гена GDF9/G1 в 2% агарозном геле
Частоты аллелей A и G в исследуемой популяции составили 0,04 и 0,96. Гомозиготный генотип АА в изучаемой выборке отсутствовал, гетерозиготный генотип AG имел очень низкую частоту (8,55%), а частота генотипа GG составила 91,45%.
Исследования, проведенные на овцах сальской породы, также показали отсутствие генотипа АА, а частоты генотипов AG и GG составили 10 и 90% соответственно [110]. Eghbalsaied с соавторами (2017) изучали полиморфизм гена GDF9 (G1) у овец Shal, Ghezel, Afshari, Lori-Bakhtyari, результаты которых показали высокую частоту генотипа GG, которая составила 85,7; 73,7; 85,4 и 88,2% соответственно. Однако в породах Shal, Ghezel и Afshari был установлен генотип АА с частотой 6,1; 1,7 и 3,6%. Интересно отметить, что в этих породах генотип АА был определен только у овец, у баранов генотип АА отсутствовал [94].
Анализ воспроизводительных качеств овец волгоградской породы показал, что все матки генотипа AG по первому окоту (2016 г.) имели по 1 ягненку, но уже по второму окоту (2017 г.) средняя плодовитость маток составила 1,50 гол. (табл.4). Многоплодие маток генотипа GG по первому окоту составило – 1,03 гол. и по второму – 1,43 гол. Достоверных различий по плодовитости между матками генотипов AG и GG в исследуемой группе не установлено. Можно отметить небольшое преимущество маток генотипа AG по второму окоту, которое составило 0,07 гол, но в среднем по двум окотам различия составили 0,03 гол.
Таблица 4 – Плодовитость овец различных генотипов гена GDF9
| Номер окота | Первый | Второй | В среднем по двум | |||
| Генотипы/ Показатели | AG | GG | AG | GG | AG | GG |
| Среднее многоплодие | 1,00 | 1,03 | 1,50 | 1,43 | 1,26 | 1,23 |
| Стандартная ошибка | 0,00 | 0,02 | 0,17 | 0,05 | 0,10 | 0,03 |
Несмотря на отсутствие различий по количеству ягнят при рождении у овец, мы обратили внимание на массу ягнят при рождении. У овец генотипа AG средняя масса ягненка по первому окоту составила 3,62 кг (табл.5). В помете было 55,6% ярочек, средний вес который составил 3,54 кг и 44,6% баранчиков, со средним весом составил 3,73 кг. По второму окоту вес ягнят в одинцовых пометах в среднем составил 3,32 кг, а в двойнях – 2,71 кг. Среди одинцовых пометов было 60% ярочек и 40% баранчиков со средним весом 3,2 и 3,5 кг соответственно. Среди двоен было по 50% ярочек и баранчиков, средний вес которых составил 2,56 и 2,86 кг соответственно.
У маток генотипа GG по первому окоту в одинцовых пометах было 52% ярочек и 48% баранчиков, средний вес которых был равен 3,37 и 3,59 кг соответственно (табл.6). Среди двоен по первому помету ярочек было 62,5%, вес которых был около 1,68 кг и 37,5% баранчиков с весом 1,93 кг.
Во втором окоте от маток было получено в одинцовых пометах 43,1% ярочек и 56,9% баранчиков с весом 3,24 и 3,48 кг. В двойнях прослеживается подобное соотношение ярочек и баранчиков (44,8 и 55,2%) вес которых составил 2,35 и 2,67 кг соответственно. В целом, у маток генотипа GGпервого окота вес ягнят в одинцовых пометах был 3,48 кг, в двойнях – 1,79 кг, второго окота 3,38 и 2,53 кг соответственно.
Полученные результаты показали, что генотип AG гена GDF9 связан с лучшей массой ягнят при рождении.
Таблица 5 — Вес ягнят при рождении у овцематок генотипа AG гена GDF9
| Стат. показатель | Одинцы | Двойни | ||||
| Ярочки | Баранчики | Все | Ярочки | Баранчики | Все | |
| Первый окот | ||||||
| Среднее | 3,54 | 3,73 | 3,62 | |||
| Стандартная ошибка | 0,07 | 0,09 | 0,06 | |||
| Станд. отклонение | 0,17 | 0,19 | 0,19 | |||
| Дисперсия выборки | 0,03 | 0,04 | 0,04 | |||
| Минимум | 3,30 | 3,60 | 3,30 | |||
| Максимум | 3,70 | 4,00 | 4,00 | |||
| Счет | 5,00 | 4,00 | 9,00 | |||
| Второй окот | ||||||
| Среднее | 3,20 | 3,50 | 3,32 | 2,56 | 2,86 | 2,71 |
| Стандартная ошибка | 0,15 | 0,20 | 0,13 | 0,20 | 0,17 | 0,14 |
| Станд. отклонение | 0,26 | 0,28 | 0,29 | 0,45 | 0,39 | 0,43 |
| Дисперсия выборки | 0,07 | 0,08 | 0,08 | 0,20 | 0,15 | 0,18 |
| Минимум | 3,00 | 3,30 | 3,00 | 1,90 | 2,40 | 1,90 |
| Максимум | 3,50 | 3,70 | 3,70 | 3,00 | 3,30 | 3,30 |
| Счет | 3,00 | 2,00 | 5,00 | 5,00 | 5,00 | 10,00 |
Таблица 6 — Вес ягнят при рождении у овцематок генотипа GG гена GDF9
| Стат. показатель | Одинцы | Двойни | ||||
| Ярочки | Баранчики | Все | Ярочки | Баранчики | Все | |
| Первый окот | ||||||
| Среднее | 3,37 | 3,59 | 3,48 | 1,68 | 1,93 | 1,79 |
| Стандартная ошибка | 0,03 | 0,03 | 0,03 | 0,12 | 0,12 | 0,09 |
| Станд. отклонение | 0,23 | 0,24 | 0,26 | 0,24 | 0,21 | 0,25 |
| Дисперсия выборки | 0,05 | 0,06 | 0,07 | 0,06 | 0,04 | 0,06 |
| Минимум | 2,80 | 3,10 | 2,80 | 1,50 | 1,70 | 1,50 |
| Максимум | 4,00 | 4,00 | 4,00 | 2,00 | 2,10 | 2,10 |
| Счет | 51,00 | 47,00 | 98,00 | 4,00 | 3,00 | 7,00 |
| Второй окот | ||||||
| Среднее | 3,24 | 3,48 | 3,38 | 2,35 | 2,67 | 2,53 |
| Стандартная ошибка | 0,07 | 0,04 | 0,04 | 0,07 | 0,06 | 0,05 |
| Станд. отклонение | 0,33 | 0,23 | 0,30 | 0,42 | 0,43 | 0,45 |
| Дисперсия выборки | 0,11 | 0,05 | 0,09 | 0,17 | 0,18 | 0,20 |
| Минимум | 2,40 | 3,00 | 2,40 | 1,70 | 2,00 | 1,70 |
| Максимум | 3,90 | 4,00 | 4,00 | 3,30 | 3,50 | 3,50 |
| Счет | 25,00 | 33,00 | 58,00 | 39,00 | 48,00 | 87,00 |
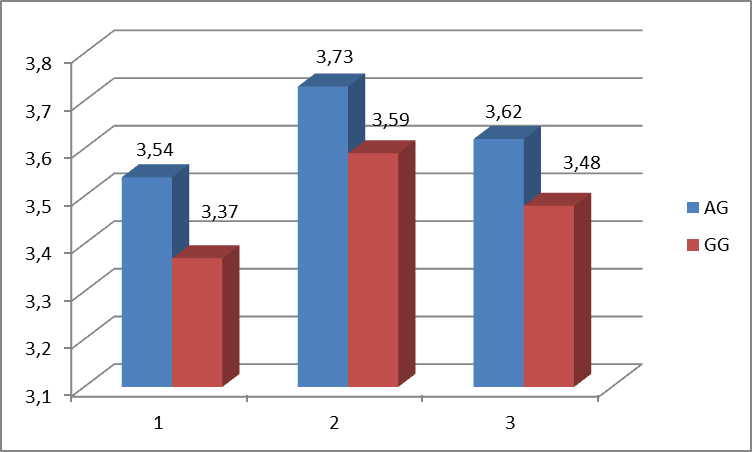
Рисунок 11 – Масса ягнят при рождении у овец различных генотипов гена GDF9 (результаты первого окота одинцовых пометов)
(На рисунке обозначения: 1 – ярочки, 2- баранчики, 3- без учета пола ягненка)
У маток генотипа AG вес ягненка при рождении в первом окоте был выше на 0,14 кг (4,02%, р=0,09) по сравнению с матками генотипа GG (рис.11). Результаты первого окота также показали, что у маток генотипа AG вес ярочек был выше на 0,17 кг (5,04%, р=0,1) и баранчиков на 0,14 кг (3,90%, р=0,12) по сравнению с матками другого генотипа.
Результаты второго окота в одинцовых пометах не показали влияния полиморфизма гена GDF9 на вес ягнят при рождении (рис.12).
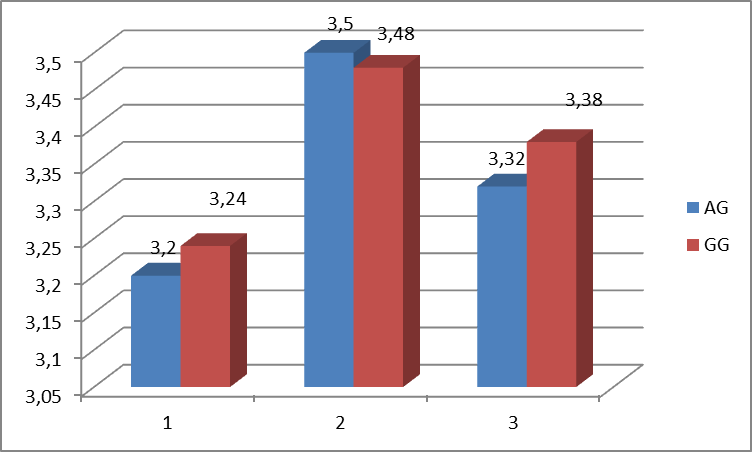
Рисунок 12 – Масса ягнят при рождении у овец различных генотипов гена GDF9(результаты второго окота одинцовых пометов)
(На рисунке обозначения: 1 – ярочки, 2- баранчики, 3- без учета пола ягненка)
Однако при анализе двоен, во втором окоте ягнята, полученные от маток генотипа AG, весили больше, по сравнению с ягнятами, полученными от маток генотипа GG. В среднем, вес ягнят, полученных от маток генотипа AG был выше на 0,18 кг (7, 11%, р=0,2). Различия между ярочками составили 0,21 кг (8,92%, р=0,2) и баранчиками 0,19 кг (7,11%.р=0,2) (рис.13).
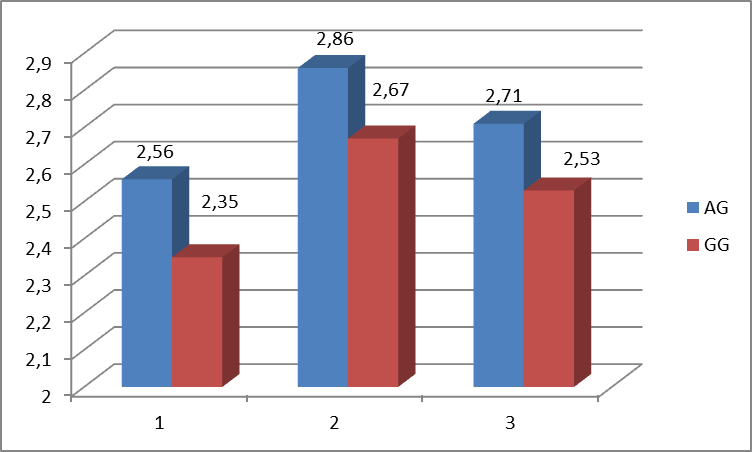
Рисунок 13 – Масса ягнят при рождении у овец различных генотипов гена GDF9 (результаты второго окота, двойни)
(На рисунке обозначения: 1 – ярочки, 2- баранчики, 3- без учета пола ягненка)
Следует отметить, что в связи с небольшим количеством маток генотипа AG, уровень достоверности влияния генотипа AG на массу ягнят при рождении находился в пределах 0,09-0,2. Однако, основываясь на результатах других исследований, мы считаем, что наличие аллельного варианта A гена GDF9 положительно влияет на воспроизводительные качества овец. Для исследуемых нами овец волгоградской породы желательным является аллельный вариант A гена GDF9, который имеет положительный эффект на массу ягнят при рождении.
Выводы
1. Результаты молекулярно-генетических исследований показали, что полиморфизм гена GDF9 у овец волгоградской породы представлен двумя аллелями A и G с частотами 0,04 и 0,96 и двумя генотипами AG и GG с частотами 8,55 и 91,45%.
2.Установлено влияние полиморфизма гена GDF9 на массу ягнят при рождении. У маток генотипа AG вес ягненка при рождении в одинцовых пометах (первый окот) был выше на 0,14 кг (4,02%, р=0,09) по сравнению с матками генотипа GG, при этом вес ярочек был выше на 0,17 кг (5,04%, р=0,1) и баранчиков на 0,14 кг (3,90%, р=0,12).
Во втором окоте вес ягнят из двоен, полученных от маток генотипа AG был выше на 0,18 кг (7, 11%, р=0,2). Различия между ярочками составили 0,21 кг (8,92%, р=0,2) и баранчиками 0,19 кг (7,11%.р=0,2).
Достоверных различий по плодовитости между матками генотипов AG и GG гена GDF9 в исследуемой группе не установлено.
2.2 Изучение полиморфизма гена гормона роста (GH) у овец карачаевской породы в контексте формирования продуктивных качеств
Выбор и обоснование направления исследования
Овцеводство исторически всегда было неотъемлемой частью народного хозяйства, особенно на территории Северо-Кавказского и Южного федерального округов, обеспечивая потребности промышленности в специфических видах сырья (шерсть, смушки, овчины), а население — в продуктах питания (баранина и молоко). В относительно недавнем прошлом экономика овцеводства в нашей стране базировалась в основном на производстве шерсти, доля которой в общей стоимости продукции этой отрасли составляла 60-80%, а реализационная цена 1 кг шерсти была эквивалентна 20 кг баранины в живой массе. В тоже время соотношение цен на шерсть и баранину на мировом рынке составляло 1:3 [30].
Мясо овец — баранина относится к наиболее ценным видам мясной продукции и пользуется повышенным спросом на мировом рынке. В связи с повышением экономической значимости мясной продуктивности важное значение приобретает использование грубошерстных пород овец, обладающих универсальной продуктивностью с акцентом на мясную [8-9, 21, 70].
Уникальность овец грубошерстных пород заключается в том, что они обладают свойствами и признаками, которые не характерны животным других направлений продуктивности: высокая резистентность к инфекционным болезням, приспособленность к резким перепадам температур, способность преодолевать большие расстояния при перегоне на пастбища [33].
История развития животноводства в целом и овцеводства в частности знает множество примеров внедрения в отрасли методов молекулярной биологии. С развитием молекулярной генетики решение вопросов прогнозирования уровня продуктивности и особенностей проявления хозяйственно-полезных признаков животных, в целях совершенствования протекающих в отраслях процессов производства, ускорения селекционного процесса, рационального использования и сохранение пород животных, стало возможно благодаря использованию в практике животноводства современных технологий молекулярно-генетического маркирования. Учеными и практиками сельскохозяйственного производства на регулярной основе прорабатывается вопрос применения достижений молекулярной генетики для решения тех или иных задач [71, 81].
Генетические маркеры чаще всего рассматривают как последовательности ДНК, используемые для идентификации некоторого участка (локуса) определенной хромосомы. Важной основой генетического маркера является полиморфизм ДНК, который может быть легко установлен при молекулярном или фенотипическом анализе [117, 22].
Использование ДНК-маркеров для повышения эффективности селекционной работы, является основой маркерной селекции (MAS).
Гены с известной функцией или известной локализацией, определяющие конкретный фенотипический признак, или тесно с ним связанные, используемые при маркерной селекции или в генетических исследованиях разного рода, носят название маркерных [7, 10-11, 92]. Современная зоотехническая наука требует в своей практике умение рационально использовать огромное количество групп животных, искусственно созданных человеком. В связи с этим в настоящее время особенно актуальна эффективная идентификация видов животных, правильная и точная дифференциация пород и внутрипородных типов. Все имеющиеся в стране породы овец, в том числе и местные аборигенные, заслуживают пристального внимания к своей генетической структуре. В практике зарубежного животноводства уже давно широко используется ДНК-маркирование поголовья [80, 84, 112].
Созданы и успешно функционируют специализированные лаборатории, оказывающие соответствующие услуги хозяйствам разных типов, а также выполняющие работы по оптимизации существующих тест-систем и поиску новых перспективных маркеров [45].
Зачастую данные лаборатории создаются на базе крупных научных организаций, или на базе высших учебных заведений соответствующего профиля. Разработка методов более эффективного использования генофонда грубошерстных пород овец с целью повышения уровня и качества мясной продуктивности, снижения затрат кормов на единицу производимой продукции, генетического контроля и управления селекционным процессом, изыскание дополнительных резервов, улучшающих экономические показатели отрасли, являются важнейшими задачами на современном этапе развития овцеводства [28, 43, 72, 83].
Постепенное развитие молекулярной биологии определило возможность создания тест-систем, обеспечивающих проведение анализа генетического полиморфизма на уровне продуктов экспрессии генов. Так называемый белковый или биохимический полиморфизм стал основой для первых молекулярных маркеров [55, 58, 67].
Однако и тест-системы данного вида не лишены недостатков. Выявление полиморфизма возможно только в последовательностях ДНК, кодирующих белок, причем исключительно у экспрессирующихся генов. Параллельно с этим существуют ограничения в выборе биологического материала для исследования, трудности при организации его отбора
Не смотря на имеющиеся ограничения и особенности реализации применения данного вида маркеров, практика использования биохимических маркеров в исследовании различных пород овец находит место и в современном овцеводстве.
Изобретение метода полимеразной цепной реакции и создание на его основе маркеров ДНК, открыло новые возможности в решении проблемы маркирования практически любого участка ДНК.
Основными свойствами, определяющими удобство использования на практике генетических маркеров являются: доступность фенотипических проявлений аллельных вариантов генов для идентификации, отличимость аллельных замещений в разных локусах, доступность для идентификации большей части аллельных замещений в каждом изучаемом локусе, малозатратность применяемой методики и ее быстрая воспроизводимость, возможность организации поточных исследований, а также возможностью автоматизации процесса исследования [11, 89, 98, 115].
Ген гормона роста (GH) рассматривается в качестве одного из перспективных маркеров по общему росту и развитию тела овец, набору живой массы, молочной продуктивности [85-86].
Известно, что гормон роста обладает широким спектром биологического действия, усиливает биосинтез белка, ДНК, РНК и гликогена, способствует распаду высших жирных кислот и глюкозы в тканях. Данный гормон не только активирует биологические процессы, сопровождающиеся увеличением размеров тела, стимуляцией роста скелета, но и координирует и регулирует скорость протекания обменных процессов [86].
Гормон роста представляет собой белок массой около 22 кДа, полипептидная цепь которого включает в себя 191 аминокислотный остаток. Одним из способов выявления полиморфизма гена GH у овец является метод PCR-SSCP, сущность которого была рассмотрена ранее.
Однако, метод ПЦР-ПДРФ считается одним из самых удобных в исполнении и дешевых по экономическим затратам для применения в животноводстве [109]. Первоначально полиморфизм гена GH у овец с помощью метода ПЦРПДРФ был изучен A. D. Malewa с коллегами у овец пород Donggala и East Java. Полиморфизм выявлялся в экзоне 3 гена гормона роста с помощью эндонуклеазы рестрикции HaeIII. Основываясь на опыте A.D. Malewa описанную тест-систему для обнаружения полиморфизма гена GH применили при исследовании овец породы Awassi [114].
В отечественном овцеводстве отмечено применение данной методики для получения сведений о наличии полиморфных вариантов гена гормона роста у овец сальской породы [110].
Связь аллельных вариантов гена гормона роста с откормочными и мясными качествами овец сальской породы изучалась в ООО «Белозёрное». Влияние генотипов гена GН на скорость роста учитывали у баранчиков по следующим показателям: вес при рождении, при отъеме и в 9 месяцев (кг); среднесуточный прирост (от отъема до 9 мес.) (г). Мясные качества учитывали по результатам контрольного убоя баранчиков в возрасте 9 мес.
Результаты тестирования полиморфизма гена GH показали, что лучшую живую массу при отъеме и в 9 мес., среднесуточный прирост, а также мясную продуктивность имели овцы генотипа АВ/GH.
Результаты исследований ученых по изучению полиморфизма гена гормона роста GН/HaeIII, проведенных на овцах породы Donggala и East Java также показали наличие полиморфизма гена с динамикой роста молодняка овец.
Карачаевская порода овец — одна из старейших пород грубошерстного типа. Родиной овец является высокогорный Карачай у устьев реки Кубань. В официальном издании МСХ РФ «Государственный реестр селекционных достижений, допущенных к использованию», карачаевская порода овец значится под номером 9354024, (оригинаторы № 296, 5670, 14214) [18].
Традиционные районы разведения овец карачаевской породы — Северная Осетия, Кабардино-Балкария, Карачаево-Черкесия. Порода мясо-шерстно-молочного направления. Создана она народной селекцией в условиях круглогодового пастбищного содержания. Это некрупная порода. Бараны весят 60-70 кг, матки — 40-50 кг. Конституция крепкая. Глубокая грудь, хорошо развитые ноги, прочный копытный рог, благодаря которому они легко пасутся в горной местности Северного Кавказа. Характерный хвост, с изогнутым в виде буквы Z кончиком, длинный, до 44 сантиметров, может содержать до 5-6 кг жира. Карачаевская порода овец рогата, причем у баранов большие спиралеобразные рога, встречаются даже трех- и четырёхрогие животные. Голова у животных этой горной породы небольшая, узкая и горбоносая. Окрас в основном черный (до 80 % от общего числа в стаде), однако есть серые, рыжие и белые особи. Карачаевские овцы были выведены в результате многолетних усилий карачаевского народа, живущего на Северном Кавказе, и в процессе выведения не была задействована ни одна из существующих пород [29].
Карачаевская порода считается универсальной, у животных ценится мясо, молоко, и шерсть, поэтому овцы высоко ценятся среди животноводов, благодаря своим свойствам сваливаться в очень плотный войлок, руно карачаевских овец идеально подходит для изготовления бурок и теплых изделий. Племенная работа направлена на увеличение маточного поголовья, поднятия живой массы овец, и выхода молодняка на 100 овцематок.
Мясо (баранина) карачаевских овец отличается высокими вкусовыми качествами. Эта особенность у карачаевской породы в значительной степени обусловлена еще и тем, что в разведении всегда придавалось большое значение их мясности, поскольку сложился большой спрос на баранину, благодаря близости курортных зон Северного Кавказа [32].
С баранов карачаевской породы, настригают в год в среднем до 3 кг шерсти, с маток настригают до 2,6 кг. Это скороспелая порода, уже в три месяца ягнята достигают 40 % живой массы тела взрослых животных. Молочность карачаевских маток довольно высокая, так, 30-50 кг составляет выход товарного молока с одной особи с жирностью до 9,6 %. Плодовитость составляет 105-110 ягнят на сто маток. Убойный выход с овечьей туши 47-56 %.
Преимущества карачаевской породы состоят в том, что она имеет отличные вкусовые качества мяса при большом убойном выходе. По показателю живой массы карачаевскую породу превосходят многие породы. Однако вкусовые качества мяса этих пород намного уступают аромату, сочности и нежности мяса карачаевской овцы. Порода характеризуется высоким выходом чистой шерсти. Овцы имеют густой покров, который образуется за счет выращивания в естественных климатических условиях. И, наконец, безусловным козырем породы является высококачественное молоко. Благодаря высокой молочности обеспечивается высокая скорость роста молодняка, что определяет высокую скороспелость и возможность получать высококачественную тушку уже в 6-месячном возрасте. И, наконец, в зависимости от спроса и конъюнктуры рынка ягнятину от овец карачаевской породы можно получать и в более раннем возрасте [35].
Сочетание инновационных и традиционных методов селекции в работе с карачаевской породой находится в стадии формирования, так как имеющихся исследований пока недостаточно для широкой интеграции различных приёмов селекции. Информация о взаимосвязи мясной продуктивности овец с полиморфными системами гормона роста в карачаевской породе – это один из элементов создания информационной базы, призванной оптимизировать селекционный процесс с породой. Исследований по выявлению взаимосвязи гена гормона роста с хозяйственно-полезными качествами у овец карачаевской породы не проводилось. Поэтому заявленная тема является актуальной с научной точки зрения и имеет безусловную практическую значимость.
Цель и задачи исследований
Целью выполненной нами научно-исследовательской работы было выявить способы и сформировать основные принципы по совершенствованию селекционно-племенной работы в племенных хозяйствах по разведению овец карачаевской породы на основе изучения полиморфизма гена гормона роста.
В связи с этим были поставлены следующие задачи:
1. Провести анализ уровня продуктивности овец карачаевской породы;
2. Изучить полиморфизм гена гормона роста овец карачаевской породы и выявить желательные генотипы, связанные с продуктивными качествами овец;
3. Оценить возможности использования полиморфизма гена гормона роста по отбору овец грубошерстных пород, генетически предрасположенных к высокой мясной продуктивности.
Материал и методы исследования
Объектом исследования были овцы карачаевской породы (n=106) ООО «Дарган» Кабардино-Балкарской республики в 2018-2019 гг. Для оценки продуктивных качеств родительских особей использовали данные зоотехнического учета хозяйства животных 2015-2016 года рождения. Животные содержались по традиционной технологии отгонно-пастбищного содержания. Подготовку овец к перегону начинают за 1-1,5 месяца. В это время проводят ветеринарно-санитарную обработку животных против инфекционных и инвазионных заболеваний. Сформированные отары закрепляют за чабанскими бригадами. Затем осуществляют ветеринарно-зоотехнический осмотр всех овец. Больных, слабых, истощенных овец к перегону не допускают. Зоотехник и ветеринарный врач проверяют трассу прогона, организуют на ней в определенных местах площадки для подкормки и водопоя овец. При перегоне строго соблюдают утвержденный маршрут и график движения. За день овцы должны проходить не более 10-15 км с периодическими остановками для отдыха (через 3 ч пути — 1-1,5 ч отдыха, через сутки— 10-12 ч отдыха и через каждые 10-15 дней перегона — 2-3 дня отдыха). Места остановок предусматривают там, где хорошие пастбища и запас концентрированных кормов. Для подвозки в пути кормов, медикаментов отары сопровождают машина или повозка. Поят овец в пути два раза в сутки. Наиболее сложно перегон переносят ягнята текущего года рождения. Перегон обычно негативно сказывается на сохранности молодняка.
Для проведения молекулярно-генетических исследований у овец (n=106) были отобраны образцы ткани с ушной раковины площадью 1 см². ДНК выделяли с использованием колонок Nexttec («Nexttec GmbH», Германия) в соответствии с рекомендациями производителя. Анализ проводили методом ПЦР-ПДРФ (полимеразной цепной реакции — полиморфизм длин рестрикционных фрагментов). Для амплификации фрагмента гена гормона роста длиной 934 п.н. использовали олигонуклеотидные праймеры:
5′- GGAGGCAGGAAGGGATGAA -3′,
5′- CCAAGGGAGGGAGAGACAGA-3′ [74].
Режим амплификации: предварительная денатурация при 95°С — 5 мин. и далее 33 цикла: 95°С — 45 с, 60°С — 45 с, 72°С — 45с; заключительный синтез при 72°С — 10 мин. Рестрикцию амплифицированного фрагмента проводили эндонуклеазой HaeIII. Наличие 10 сайтов рестрикции соответствовало аллелю А, наличие 11 сайта аллелю В. Размер полученных рестрикционных фрагментов определяли методом электрофореза в 4 %-ном агарозном геле в присутствии бромистого этидия. По результатам молекулярно — генетического анализа устанавливали наличие и частоту аллелей и генотипов. Мясные качества учитывали по результатам контрольного убоя баранчиков в возрасте 8 мес. по следующим показателям: предубойная масса (кг), масса мякоти (полутуши) (кг), убойная масса (кг), убойный выход (%). Все исследуемые животные были одного года рождения, с минимальными различиями в возрасте, содержались в одинаковых условиях кормления и распорядка дня, обслуживались одними и теми же работниками. Материалы исследований подвергались биометрической обработке, которые согласуются с алгоритмами, предложенными для животноводства Плохинским и Меркурьевой.
Результаты исследований
Карачаевская порода является наиболее многочисленной среди грубошерстных пород Российской Федерации. Общее поголовье данной породы составляет в нашей стране около 287 тыс. голов. По грубошерстным породам насчитывается 98 племенных организаций (в том числе 1 селекционно-генетический центр и 10 племенных заводов).
Лучшие показатели по реализации племенных овец в расчете на 100 маток по грубошерстным породам варьируют в интервале от 22 до 56 голов. По карачаевской породе этот показатель составляет 29,2 головы. На этом фоне ООО «Дарган» Кабардино-Балкарской Республики выделяется хорошими показателями: 37 голов на 100 овцематок – таков этот показатель был в 2018 году. Живая масса животных карачаевской породы различных половозрастных групп находится в пределах: баранов-производителей – 79 кг, ремонтных баранчиков-годовиков 62 кг, овцематки 46-47 кг и ярки-годовики в среднем 41-42 кг. Общее поголовье овец в ООО «Дарган» составляет около 9,4 тыс. голов, в том числе более 50 % (4700) овцематок. Средний настриг на остриженную голову составил 1,4 кг и выход чистой шерсти 68 %. На 100 овцематок получено 95 ягнят и сохранено к отъёму от овцематок 93 %. Средняя живая масса по группе баранов-производителей -79 кг и по группе овцематок 54 кг, что почти на 8 % выше, чем средние данные по племенным хозяйствам.
На первом этапе в результате проведения молекулярно-генетических исследований у овец карачаевской породы были определены аллельные варианты гена GН (рис.14) и установлены генотипы, представленные фрагментами: 277-, 202-, 110-, 100-, 94-, 68-, 49-, 22- , 8- и 4 н.п. генотип АА; 256-, 202-, 110-, 100-, 94-, 68-, 49-, 22-, 21-, 8- и 4 н.п. генотип ВВ и 277-, 256-, 202-, 110-, 100-, 94-, 68-, 49-, 22- ,21-, 8- и 4 генотип АВ, 9- маркер.
.
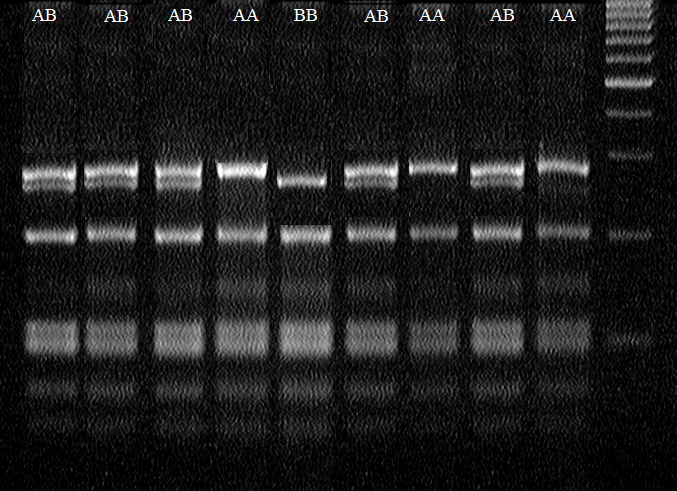
Рисунок 14 – Электрофореграмма результата ПЦР-ПДРФ гена GН в 4% агарозном геле
Частота встречаемости трех генотипов АА, АВ и ВВ установлена в ходе эксперимента в соотношении 51, 40 и 15%, соответственно (табл.7). В целом у карачаевской породы овец наибольшую частоту имел аллель А и гомозиготный генотип АА.
Таблица 7 – Частота аллелей и генотипов гена GН овец карачаевской породы, n=106
| Ген | Аллели | Генотипы | |||
| А | В | AA | AВ | ВВ | |
| GН|HaeIII | 0.67 | 0.33 | 51 | 40 | 15 |
Проведение дальнейших исследований по изучению связи аллельных вариантов гена GН с показателями мясной продуктивности карачаевской породы овец показало, что наилучшую мясную продуктивность имели баранчики генотипа АВ/GH, которые достоверно превосходили аналогов генотипа АА/GH практически по всем анализируемым признакам (табл.8).
Таблица 8 – Результаты контрольного убоя баранчиков различных генотипов гена GH
| Генотип | Предубойная живая масса, кг | Масса туши (парной), кг | Масса туши (охлажденной), кг | Убойная масса, кг | Убойный выход, с курдюком % |
| АА | 38.0± 1.78 | 16.4± 0.81 | 16.0± 0.73* | 19.3± 0.72 | 50.7 |
| АВ | 41.7± 1.24** | 18.9± 0.40 | 18.4± 0.54 | 21.5± 0.69 | 51.6 |
| ВВ | 39.1± 2.85** | 16.9± 0.34* | 16.3± 0.58 | 20.1± 0.51 | 51.1 |
*р≤0.05 **р≤0.01
Анализ материалов, полученных в результате контрольного убоя, свидетельствует, что баранчики карачаевской породы генотипа АВ/GH превосходили по предубойной массе своих сверстников с генотипами АА/GH и ВВ/GH на 3.7 и 2.6 кг.
Убойная масса и убойный выход у баранчиков генотипа АВ/GH также превышали данные показатели баранчиков генотипа АА/GH на 2.2 кг и 0.9% соответственно.
Аналогичная закономерность наблюдалась и при сравнении гетерозигот с баранчиками генотипа ВВ/GH. Убойная масса и убойный выход у баранчиков генотипа АВ/GH были больше на 1.2 кг и 0.5% соответственно.
Полученные данные полиморфизма гена гормона роста GH согласуются с результатами исследований других пород овец [75]. Так, согласно данным у породы Donggala частота встречаемости генотипов составляла АА-0,357, АВ-0,357 и ВВ-0,286. У породы East Java АА-0,464, АВ-0,250 и ВВ-0,286 [113]. В результате работы отечественных исследователей по обнаружению полиморфных вариантов гена гормона роста для овец сальской породы были получены следующие данные частоты встречаемости генотипов: АА-0,57, АВ-0,36 и ВВ-0,07 [100].
Подводя итог данной работы, нужно отметить, что впервые получены результаты полиморфизма гена GН у овец карачаевской породы и выявлены достоверные ассоциации между генотипами гена GН и селекционно-ценными признаками овец карачаевской породы.
Полученные результаты показали перспективность гена в качестве маркера откормочной и мясной продуктивности овец и дальнейшие исследования в данном направлении позволят разработать селекционные программы по совершенствованию карачаевской породы овец с учетом полиморфизма гена GН.
Все это очередной раз подтверждает необходимость поиска и изучения ДНК-маркеров, ассоциированных с продуктивными качествами овец для большей эффективности селекционных работ и повышения рентабельности отрасли овцеводства.
В процессе выполнения работы нами была решена задача оценки состояния исследуемой популяций овец карачаевской породы с точки зрения достоверности различий значений наблюдаемой и ожидаемой гетерозиготности. Частота гетерозигот является важным показателем, т.к. каждая гетерозиготная особь несет разные аллели и тем самым иллюстрирует наличие изменчивости. Значения наблюдаемой и ожидаемой гетерозиготности в исследованной популяции овец карачаевской породы представлены в таблице 9.
Таблица 9 — Наблюдаемая и ожидаемая гетерозиготность гена гормона роста в популяции овец карачаевской породы
| Порода | Количество
животных (n) |
Наблюдаемая гетерозиготность (HO) | Ожидаемая гетерозиготность (HE) | χ² |
| Карачаевская порода | 106 | 0,377 | 0,442 | 2,311 |
В нашей работе во всех случаях не было обнаружено достоверной разности значений наблюдаемой и ожидаемой гетерозиготности. Наблюдаемые распределения гетерозиготных генотипов достоверно соответствовали ожидаемым по закону о равновесии Харди-Вайнберга, согласно полученному значению χ² — это показывает, что изученные популяции находились в генном равновесии.
Выводы
Повышение экономического значения мясного направления в овцеводстве требует концентрации внимания руководства сельскохозяйственных предприятий на проявлении максимального уровня мясной продуктивности, скороспелости овец, качестве получаемой баранины, ее органолептических характеристиках.
На основании проведённых нами исследований можно найти подтверждение о возможности внедрения практики использования молекулярно-генетических маркеров мясной продуктивности овец в селекционной работе с овцами карачаевской породы наряду с традиционными методами племенной работы.
Проведен анализ уровня продуктивности овец карачаевской породы, изучен полиморфизм гена GH. Это позволяет дополнить имеющиеся базы данных по использованию их в практической селекции.
Установлено влияние полиморфизма генотипов гена гормона роста на мясные показатели и определены желательные генотипы для дальнейшей селекционной работы по повышению их удельного веса в структуре популяции. Результаты проведенных исследований свидетельствуют о целесообразности использования генетических маркеров для оптимизации селекционных программ в стадах овец грубошерстных пород с целью повышения уровня мясной продуктивности.
3 Анализ мировых информационных ресурсов, предоставляющих доступ к результатам генетических исследований
Выбор и обоснование направления исследования
Современная молекулярная биология к настоящему времени накопила беспрецедентно огромные объемы экспериментальных данных. Эти данные могут быть эффективно использованы на различных этапах молекулярно-генетических исследований. В настоящее время в сети Интернет существуют сотни баз данных, содержащие материалы по молекулярной биологии и другим смежным дисциплинам. Каждая из них имеет свой формат хранения данных, различную степень избыточности, взаимосвязи с родственными или аналогичными базами данных. Понимание их назначения, владение способами работы с ними является дополнительным инструментом в руках исследователя. Каждая база данных имеет также свои средства доступа к информации, различные поисковые программы, инструменты визуализации, пополнения базы. Важную роль при этом играют специальные методики и подходы, позволяющие осмыслить эти данные и интерпретировать их в контексте собственных исследований. Данная сфера знаний носит название информационной биологии и биоинформатики [52].
Биоинформационные подходы в геномике включают в себя: базы данных, в которых хранится биологическая информация; набор инструментов для анализа тех данных, которые лежат в таких базах; правильное применение компьютерных методов для правильного решения биологических и медицинских задач, поиск геномных маркеров, ассоциированных с различными заболеваниями.
Цель исследований
Целью работы было изучение наиболее популярных сетевых информационных ресурсов, предоставляющих доступ к результатам генетических исследований, в контексте их информационного потенциала и технологии работы с ними.
Материалы и методы исследований
Материалом исследований являлись Информационная система Национального центра биотехнологической информации США (NCBI), база данных локусов количественных признаков сельскохозяйственных животных AnimalQTLdb, база данных нуклеотидных последовательностей GenBank, Bovine Genome Database, «SheepGenomesDatabase» (SGD), комплекс информационных ресурсов по геному свиней.
Методами исследований являлись изучение методических материалов и самостоятельная работа с ресурсами.
3.1 Способы классификации геномных баз данных
Современные банки и базы данных по биологии можно классифицировать по содержащейся в них информации следующим образом:
- Библиографические;
- Первичных последовательностей ДНК, РНК, белков;
- Пространственной структуры молекул;
- Геномные;
- Прочие тематические.
В мире существует лишь несколько крупных центров, поддерживающих банки данных первых четырех типов, и большое количество организаций, содержащих базы данных по определенным темам (например, база данных по рибосомальным генам или классификации ферментов, по мутациям гена p53 или белкам Saccharomycescerevisiae и т.д.).
В зависимости от организации информации базы данных подразделяются на архивные, производные и курируемые базы данных.
Архивные базы данных:
- GeneBank& EMBL – здесь хранятся первичные последовательности всех расшифрованных геномов;
- PDB – пространственные структуры белков.
Курируемые базы данных:
- Swiss-Prot – наиболее качественная база данных, содержащая аминокислотные последовательности белков;
- KEGG – информация о метаболизме;
- FlyBase – информация о Drosophila;
- COG – информация об ортологичных генах в алфавитном списке напротив названия каждого.
Производные базы данных
Такие базы получаются в результате обработки данных из архивных и курируемых баз данных:
- SCOP – База данных структурной классификации белков
- (описывается структура белков);
- PFAM – База данных по семействам белков;
- GO (GeneOntology) – Классификация генов (попытка создания набора терминов, упорядочивания терминологии, чтобы один ген не назывался по-разному, и чтобы разным генам не давали одинаковые названия);
- ProDom– белковые домены.
3.2 Информационная система Национального центра биотехнологической информации США (NCBI)
Особую роль в молекулярно-генетических исследованиях играет информационная система NCBI (Национальный центр биотехнологической информации США) https://www.ncbi.nlm.nih.gov/. NCBI организован в 1988 как отделение National Library of Medicine (NLM) в National Institutes of Health (NIH).
В задачи базы данных NCBI входит:
- Создание автоматизированных систем для хранения и анализа данных по молекулярной биологии, биомедицине и генетике.
- Компьютерная обработка данных полученных в исследованиях структуры и значения биологически активных молекул и веществ.
- Содействие широкому использованию баз данных и программного обеспечения для исследователей в области биотехнологий и медицинского персонала.
- Координирование усилий по накоплению биотехнологической информации по всему миру.
В настоящее время информационная система NCBI предоставляет крупнейшую биологическую база данных (молекулярная биология, биохимия и генетика), а также мощные системы обработки и представления этих данных. С ее помощью можно получить необходимую информацию о полной последовательности ядерного или митохондриального генома исследуемого вида животных, которая может использоваться в качестве референсной популяции для сравнительных исследований с собственными полученными данными. NCBI также обеспечивает доступ к таким информационным ресурсам, как:
- база данных белковых доменов;
- база данных аннотированных последовательностей ДНК и РНК (GenBank);
- базы данных научной литературы (PubMed) и таксономичной информации (TaxBrowser), обеспечивает поиск данных о конкретном биологическом виде (Taxonomy).
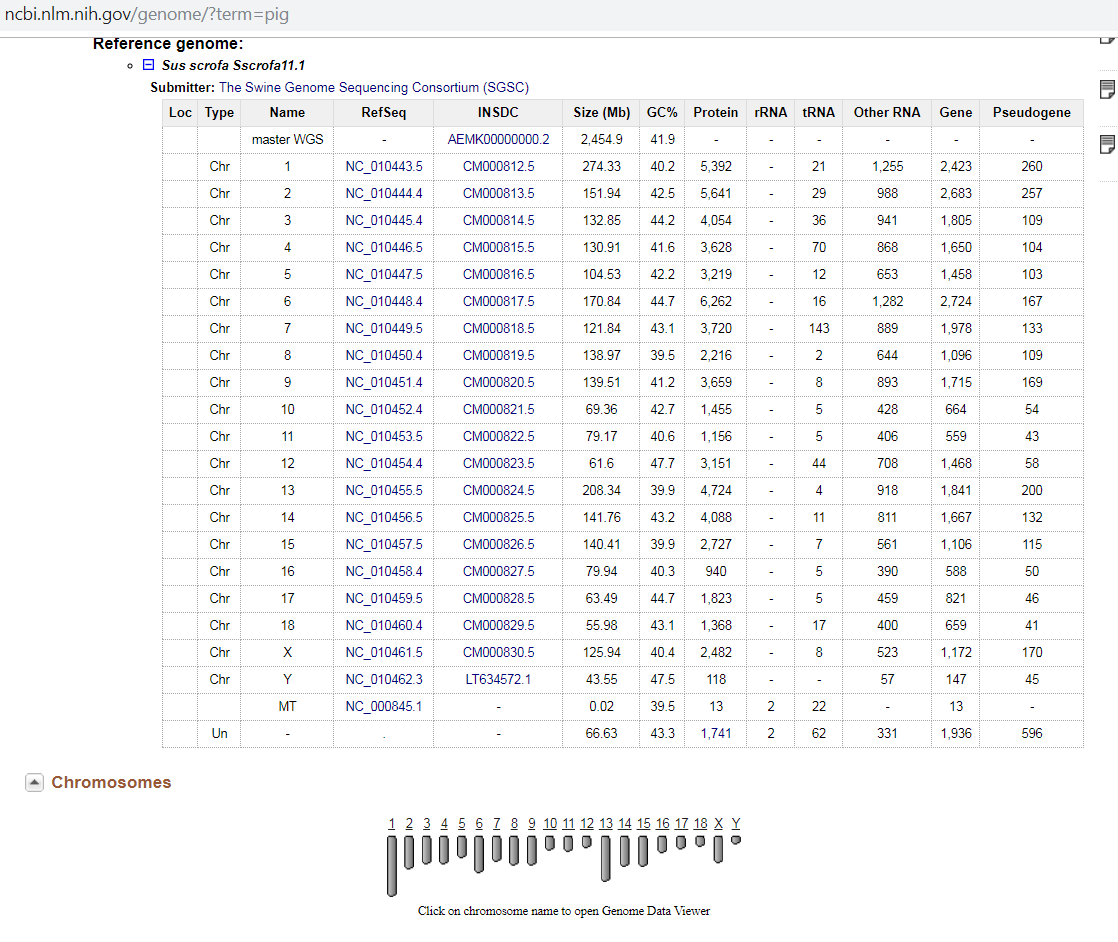
Рисунок 15 – Представление информации по геному свиней в NCBI
В настоящее время список секвенированных геномов живых организмов в базе NCBI Genome представлен: Eukaryotes (9184); Prokaryotes (209746); Viruses (32428); Plasmids (18162); Organelles (14566).
В качестве примера представления информация информационной системой NCBI на рисунке 1 представлены данные из области исследования генома свиней (SusScrofa).
Наряду с информационными ресурсами NCBI предоставляет специализированное ПО, предназначенное для решения задач обработки и анализа данных в области биоинформатики (BLAST).
3.3 База данных локусов количественных признаков сельскохозяйственных животных AnimalQTLdb
В базе данных NCBI для генетических исследований большой интерес представляет информационный ресурс AnimalQTLdb, который был разработан для размещения общедоступных данных количественных локусов признаков (QTL) для крупного рогатого скота, кур и свиней, чтобы предоставить инструменты для согласования различных характеристик генома с QTL и для сравнения результатов QTL. В 2007 году были разработаны инструменты, позволяющие сопоставлять карты QTL с различными признаками генома, такими как концевые последовательности бактериальной искусственной хромосомы (BAC), однонуклеотидные полиморфизмы (SNP), (RH) отметки привязки карты и т.д. В сочетании с AnimalQTLdb сравнение QTL между видами стало возможным благодаря виртуальной сравнительной карте (VCmap), инструменту, совместно разработанному Университетом штата Айова, Медицинским колледжем штата Висконсин и Университетом Айовы (http://www.animalgenome.org/VCmap). Эти усилия позволили мировому сообществу получать и анализировать данные QTL. За последние несколько лет был достигнут значительный прогресс. Во-первых, база данных была расширена, в нее были включены еще два вида: овцы и радужная форель ( http://www.animalgenome.org/QTLdb/notes.php). Между тем, новые данные QTL активно курируются в базе данных. С 2007 года количество QTL в базе данных увеличилось в 5,5 раза, достигнув 17 566 QTL (5920 голов крупного рогатого скота, 3442 кур, 7451 свиней, 753 овец и 88 радужной форели). Одно из преимуществ AnimalQTLdb перед аналогичными инструментами базы данных состоит в том, что одни и те же инструменты, разработанные для одного вида, могут быть легко применены к другим видам и могут эффективно использоваться повторно, что позволяет максимизировать ценность средств, вложенных в развитие. В 2013 году вышел QTLdbHorse.
3.4 База данных нуклеотидных последовательностей GenBank
GenBank включает более чем 70,000 организмов (для кодирующих последовательностей приведена трансляция). GenBank – участник (вместе с EMBL и DDBJ) консорциума баз данных (базы данных обмениваются данными ежедневно и потому эквивалентны; описания и номера всех последовательностей одинаковы).
Доступ (получение последовательностей) осуществляется через Entrez (поиск по названию, номеру, организму, автору и т.п.). Поиск по подобию – с помощью BLAST.
Для представления последовательностей в GenBank предложено два инструмента (перед отправкой полезно провести поиск векторных последовательностей с помощью VecScreen– инструмента, позволяющего идентифицировать в предложенной последовательности компоненты векторов, линкеров или адаптеров. Разработан для предотвращения загрязнения публичных баз данных векторами и т.п):
1. BankIt – WWW представление одной или нескольких последовательностей.
2. Sequin – WWW представление для длинных последовательностей, полных геномов, результатов популяционных и филогенетических исследований.
Разделы GenBank:
• ESTs – expressed sequence tags; короткие (300-500), просеквенированные один раз cDNA последовательности. Также включают последовательности cDNA из RACE и differential display экспериментов.
• GSSs — genomesurveysequences; короткие, просиквенированные один раз геномные последовательности, exontrapped последовательности, cosmid/BAC/YAC концы и т.п.
• HTGs — highthroughputgenomesequences от крупных сиквенсовых центров. Незавершенные и завершенные последовательности.
• STSs — sequencetaggedsites; короткие (200-500) последовательности, уникальные для данного генома. Используются для картирования геномов.
Reference Sequences база данных. Неповторяющиеся последовательности геномной DNA, mRNA и известных белков (в будущем — хромосом).
База данных single nucleotide polymorphisms, небольших делеций/инсерций, полиморфных повторяющихся элементов и microsatellite variation (как клинические, так и нейтральные). Содержит популяционные частоты распространённости.
В этой базе данных ESTs и полноразмерные mRNA последовательности организованы в уникальные кластеры, представляющие известные или предполагаемые гены. Для кластеров представлена информация по картированию, экспрессии и другие ресурсы.
В настоящее время для четырёх организмов: Homo sapiens, Mus musculus, Rattus norvegicus, Danio rerio.
Online Mendelian Inheritance in Man – база данных человеческих генов и генетических заболеваний под редакцией Dr. Victor A. McKusick. Имеются ссылки на Entrez базу данных.
The OMIM MorbidMap – алфавитный каталог генетических заболеваний и их цитологической локализации.
Представлены геномные карты (с Entrez связями) для Fruitfly, Human, Malaria parasite, Mouse, Rat, Retroviruses, Zebrafish. По возможности суммированы данные UniGene, сиквенс, имеющиеся мутанты, гомологи из других организмов.
Обеспечивает доступ к нуклеотидным и белковым последовательностям (GenBank, EMBL, DDBJ, PIR-International, PRF, Swiss-Prot и PDB, GenPept, RPF), 3-х мерным структурам и популяционным данным. Практически для каждой последовательности можно затребовать подобные последовательности или структуры (заранее вычисленные) и MEDLINE ссылки, относящиеся к последовательности. Относительно большие запросы можно организовывать с помощью BatchEntrez. Запрос можно организовывать с помощью разного рода текстовой информации: имя автора, организм, название гена или белка, уникальный идентификатор (accession number, sequence ID, PubMed ID, MEDLINE UID).
Обеспечивает доступ к полным секвенированным геномам (>800 эукариот, >500 вирусов, >25 бактерий, Saccharomyces cerevisiae, Drosophila melanogaster; включает незаконченные геномы: human, mouse, Caenorhabditis elegans, Plasmodium falciparum, Leishmania major, rice, and corn). Графическое представление геномов. Интегрированные хромосомные карты дрозофилы и человека.
3.5 Специализированные информационные ресурсы по геномам конкретных видов животных
3.5.1 Bovine Genome Database – крупнейшая база данных в области исследования генома крупного рогатого скота
Геном коровы герефордской породы был секвенирован Консорциумом по секвенированию и анализу генома крупного рогатого скота (Bovine Genome Sequencing and Analysis Consortium (BGSAC)), группой исследователей во главе с Национальными институтами здравоохранения (National Institutes of Health) и Министерством сельского хозяйства США (U.S.Department of Agriculture) в 2009 году. Это один из самых крупных из когда-либо секвенированных геномов [87].

Рисунок 16 – Корова L1 Dominette 01449, породы Герефорд, выбранная в качестве объекта исследований в проекте BovineGenomeProject
Результаты, опубликованные в журнале Science 24 апреля 2009 г., до сих пор оказывают существенное влияние на животноводство. Они были получены благодаря объединенным усилиям более чем 300 ученыхиз 25 стран мира и работе, продолжавшейся 6 лет. Размер генома коровы составляет 3 Гб (3 миллиарда пар оснований), он содержит приблизительно 22 000 генов, из которых 14 000 являются общими для всех видов млекопитающих. Коровы делят 80 процентов своих генов с людьми (рис. 17). Также геном коров включает около 1000 генов, общих с собаками и грызунами, но не идентифицированных у людей.
Рисунок 17 – Сравнительная хромосомная карта Homo sapiens и Bos taurus
Дальнейшие исследования и уточнения, а также работа с полученными данными потребовали разработки специальной базы данных с разветвленной системой запросов, в которой бы хранилась и структурировалась информация о геноме коров. Таким решением стала база данных Bovine Genome Database (BGD), разработанная в 2010 году коллективом авторов из США (рисунок 18).
База данных генома крупного рогатого скота Bovine Genome Database(BGD) поддерживает работу исследователей генома крупного рогатого скота, предоставляя инструменты анализа данных, навигации по геному и аннотирования [87].
Целью BGD изначально было оказание поддержки Консорциуму по секвенированию и анализу генома крупного рогатого скота (BGSAC). При разработке базы данных исследователи столкнулись с рядом проблем:
— поддержание постоянноговысокого качества базы данных, несмотря на разнообразие запросов исследовательского сообщества,
— поддержание согласованных форматов данных,
— минимизация возможного дублирования при пополнении базы данных.
BGD включает обозреватели генома GBrowse, редактор аннотирования Apollo, средство просмотра локусов количественных признаков (QTL), базы данных последовательностей BLAST и страницы генов. Браузеры генома отображают Официальный набор генов крупного рогатого скота версия 2 (OGSv2), модели генов RefSeq и Ensembl, некодирующую РНК, повторы, псевдогены, однонуклеотидные полиморфизмы SNP, маркеры, QTL и выравнивания для комплементарной ДНК, ESTs (expressed sequence tags) и белковые гомологи. Браузер локусов количественных признаков QTL коров подключен к BGD ChromosomeGBrowse, что позволяет идентифицировать гены-кандидаты, лежащие в основе QTL. Редактор аннотаций Apollo напрямую подключается к базе данных BGD Chado, чтобы предоставить исследователям удаленный доступ к генетическим данным в графическом интерфейсе, который позволяет редактировать и создавать новые модели генов. Исследователи могут загружать свои аннотации на сервер BGD для просмотра и интеграции в последующую версию OGS. Страницы, посвященные генам, отображают информацию для отдельных моделей генов OGS, включая их структуру, варианты транскриптов, функциональные описания, символы гена, термины генной онтологии, комментарии аннотатора и ссылки на базы данных NCBI и Ensembl [95].
Исследования генома крупного рогатого скота помогают в решении фундаментальных вопросов биологии и эволюции жвачных животных и выявления генов, связанных с признаками, важными для человека, как, например, признаки мясной и молочной продуктивности, а также влияние производства на экологию и здоровье животных [96].
Рисунок 18 – Общий вид домашней страницы базы BovineGenomeDatabase
Просмотр генома и аннотации к руководству сообщества были основными областями внимания BGD в его оригинальной версии, опубликованной в NucleicAcidsResearch в 2011 году. С тех пор основные усилия разработчиков были сосредоточены на инструментах для множественных версий сборки генома, новых инструментах аннотации и включении новых данных об экспрессии генов. Позднее с развитием GWAS исследований появилась необходимость работы с большими наборами геномных данных. Чтобы помочь в интерпретации этих данных, аннотирование крупного рогатого скота должны быть доступным для широкого круга исследователей, а информация должна быть представлена в форматах, удобных для статистической обработки. Эта проблема была решена с разработкой BovineMine, нового инструмента интеллектуального анализа данных, которое позволяет пользователям создавать настраиваемые наборы данных с функциональной информацией, которая может быть интегрирована с результатами их исследований. Сборка референсного генома коровы производилась несколько раз. Новейшая версия BovineMine (BovineMine v1.6) включает сборки генома ARS-UCD1.2 и UMD3.1, однако версия BovineMine v1.4 для сборки UMD3.1 по-прежнему доступна. JBrowse работает со сборками ARS-UCD1.2 и UMD3.1.
BovineMine объединяет сборки генома с различными данными, включая гены, белки, ортологи, онтологию генов, экспрессию генов, их взаимодействия, QTL и публикации, посвященные исследованию генома коров. BovineMine позволяет ускорить геномные исследования, давая возможность исследователям без навыков программирования создавать и экспортировать настраиваемые массивы данных аннотирования, объединять их с результатами собственных исследований, а также использовать в последующих анализах. BovineMine позволяет исследователям отслеживать пути генов в модельных организмах (например, человека, мыши и крысы) на основе ортологии и особенно полезен для анализа GO (GeneOntology) и исследований в сочетании с исследованиями GWAS (полногеномных исследований) и QTL (локусов количественных признаков). BovineMine также содержит референсные геномы овец и коз, позволяя исследователямсравнивать данные нескольких видов жвачных животных.
BovineMine может быть полезен для изучения функций генов, связанных с признаками в исследованиях GWAS и QTL. Такие методы, как анализ избыточной презентативности после GWAS или GSEA в сочетании с анализом SNP, позволяют понять генетические механизмы и выявить гены-кандидаты с умеренным эффектом. Для выбора данных о функциях генов и онтологиях, пользователи могут загрузить список SNP с помощью инструмента «List», выполнить поиск местоположения с помощью шаблона «SNP → Хромосома и координаты», а затем загрузить местоположения в поиск регионов, чтобы получить гены, расположенные вблизиисследуемого SNP. Затем этот список генов может быть использован в QueryBuilder для получения таблиц, содержащих информацию о функциях или с шаблонными запросами, такими как «Gene → Gene Ontology» и «Gene → Pathway». Использование ортологов человека или мыши с шаблоном «Gene → Orthologue → Pathway» может дать дополнительную информацию о функции гена (рис. 19).
Рисунок 19 – Пример выполнения запроса на поиск в геноме КРС гомологов гена по его идентификатору и выполняемых ими функций
Преимущество BovineMine по сравнению с другими информационными ресурсами для исследований генома крупного рогатого скота заключается в интеграции данных о специфической тканевой экспрессиис данными о геномной вариации. Для удобства пользователя сформированы и доступны для работы шаблоны «QTL Id → Транскрипт и его экспрессия» и «QTL Признак → Транскрипт и его экспрессия». Если пользователь вошел в систему, идентификаторы транскрипта, предоставленные в ходе выполнения запроса, могут быть сохранены в виде списка и затем использованы в качестве входных данных для других шаблонов, таких как «Транскрипт → Ген → Онтология гена» [103]. Для очень сложных запросов, объединяющих несколько больших наборов данных, пользователь может разбить задачу на несколько отдельных запросов, используя результаты выполнения одного запроса в качестве входных данных для последующих.
Совместная работа BovineMine с другими информационными ресурсами
BovineMine дополняет другие информационные ресурсы, посвященные исследованию генома КРС, такие как AnimalQTLdb, CattleBioMarts (http://AnimalGenome.org) и SNPchiMp.
На веб-сайте AnimalGenome.org также есть доступ к базе данных локусов количественных признаков (CattleQTLdb), который регулярно пополняется из новых опубликованных данных и содержит информацию о QTL и GWAS. Интерфейс поиска позволяет просматривать информацию о QTL, структурированную по хромосомам и по иерархическим классам признаков. BovineMine поддерживает идентификаторы QTL из CattleQTLdb, что позволяет получать данные из этой базы. Исследователи могут выполнить поиск в CattleQTLdb, чтобы найти определенный QTL, а затем ввести идентификатор QTL в BovineMine, чтобы определить связанные с ним гены, их функции и тканеспецифичную экспрессию [118].
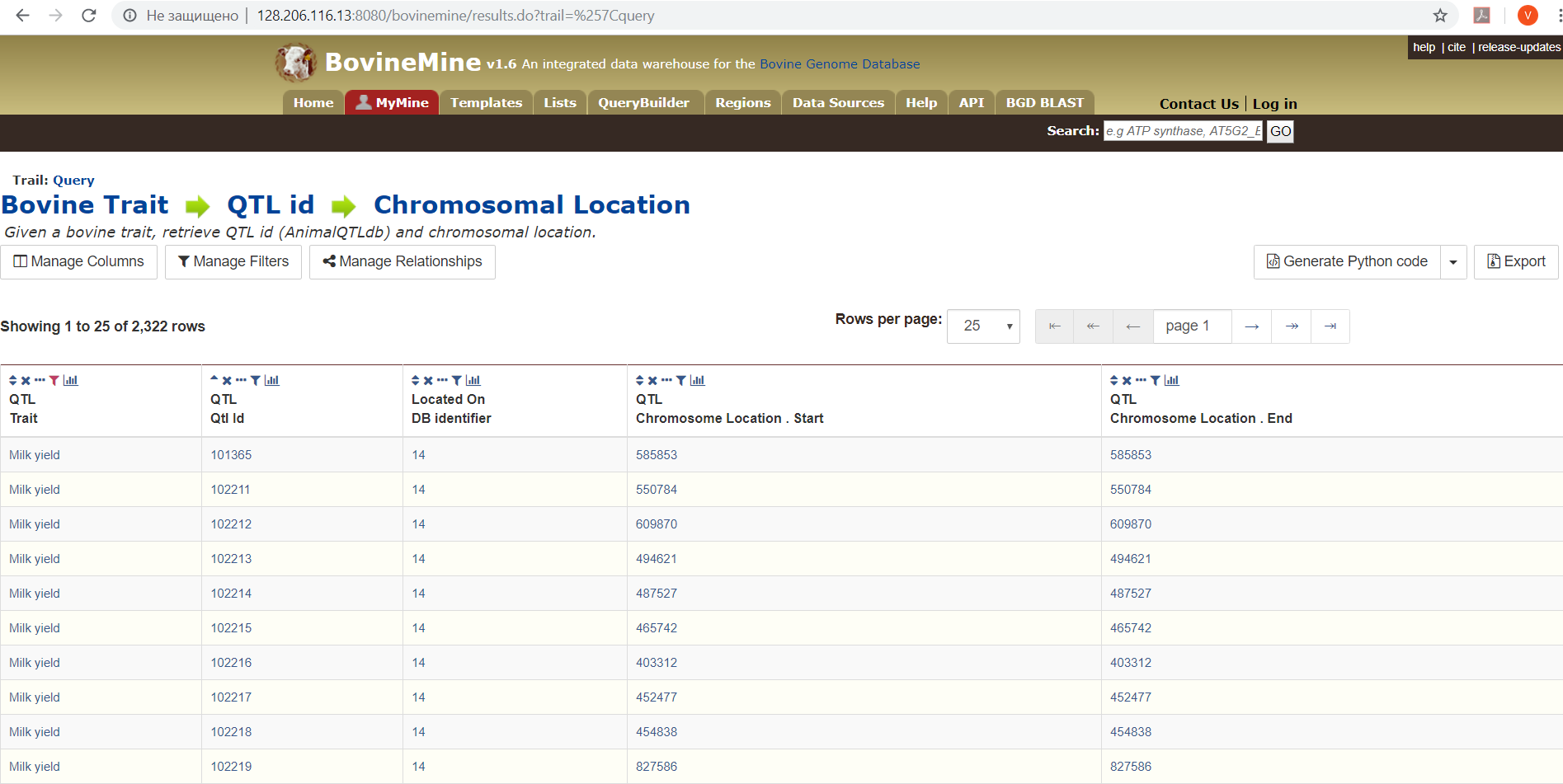
Рисунок 20 – Пример выполнения запроса на поиск в геноме КРС генов, отвечающих за признак «молочная продуктивность» (QTL milk yield)
3.5.2 Исследование генома овец
Овцы стали одними из первых одомашненных человеком животных. По данным многочисленных исследований это событие произошло в период от 11,000 до 9,000 лет до н.э. в Месопотамии. Наиболее распространена версия происхождения домашней овцы (Ovis aries) от азиатского муфлона (O. orientalis). Выращивание овец и формирование пород началось либо в Юго-Западной Азии, либо в Западной Европе. Скульптуры, найденные при археологических раскопках в Иране, позволяют предположить, что отбор овец шерстного направления начался около 6000 до н. э. Овцы, относящиеся к жвачным животных, обладающим специализированным пищеварительным органом – рубцом, осуществляющим первоначальную переработку растительной пищи, являются важным источником мяса, молока и шерсти. На сегодняшний день, овцы также являются важной биомедицинской моделью, особенно в Австралии.Детальное исследование сходств и различий между овцами и человеком на молекулярном уровне позволит использовать новые более эффективные методы лечения ряда заболеваний, таких как сепсис и астма [116].
В 2014 году в журнале Science была опубликована статья, посвященная результатам секвенирования генома овцы. Этой публикации предшествовали 8 лет кропотливой работы, в которой участвовали ученые из 26 институтов, расположенных в 8 странах мира (CSIRO Livestock Industries, AgResearch, Utah State University, BGI-Shenzhen, The Roslin Institute, HGSC – BCM, INRA Toulouse). Ученые из CSIRO (Государственное объединение научных и прикладных исследований Австралии – Commonwealth Scientific and Industrial Research Organisation) возглавляли коллектив исследователей, задачей которого было секвенировать геном овцы с целью поиска более эффективных стратегий разведения и разработки новых подходов к содержанию овец в Австралии и по всему миру [91].
С помощью технологий Illumina были секвенированы геномы овец породы Texel (рис. 21). Сборка v1.0 (ACIV000000000) была основана на результатах секвенирования генома овцы. Затем для ее уточнения и заполнения обнаруженных пробелов, был расшифрован геном барана этой же породы. Размер овечьего генома составляет около 2.7 миллиарда пар оснований, расположенных на 26 парах аутосом и паре половых хромосом.
На сегодняшний день разработана уже четвертая версия сборки генома овцы Oar_rambouillet_v1.0, датированная февралем 2017 года. Каждая последующая сборка позволяет устранить неточности и заполнить пробелы впредыдущей. Однако, в качестве референсной до сих пор используется сборка Oar_v3.1 (GCA_000298735.1).

Рисунок 21 – Овцы голландской породы Texel
Международный консорциум по геномике овец (InternationalSheepGenomicsConsortium)
ISGC была создана при участии Meat&Livestock Australia и Australian Wool Innovation Limited, а также 11 австралийских и новозеландских исследовательских организаций. Люди, стоявшие у истоков создания ISGC,так описывали его общую задачу — «найти полезные гены и заставить их работать». Неудивительно, что инициаторами создания консорциума по исследованию генома овец стали именно Австралия и Новая Зеландия, на долю которых приходится порядка 70% от общего объема экспорта баранины в мире.
Долгосрочной целью работы консорциума является разработка ресурсов для международного научного сообщества, занимающегося геномикой овец, что позволит снизить временные и финансовые затраты. Первоначальной задачей было создание виртуального генома овец (рис. 22). Для этого использовались технологии WGS и Roche 454 FLX вместе с дополнительным секвенированием с помощью технологий Illumina GA.
Рисунок 22 – Домашняя страница ISGC
Эта стратегия позволила создать 50К SNP чип для овец. Затем чип SNP использовался для создания карты гаплотипов низкого разрешения с использованием образцов, полученных от представителей разных пород. Чип 50k SNP доступен для использования исследовательскими группами для выявления маркеров, связанных с такими важными признаками, как устойчивость к болезням, а также улучшение мясных, молочных и шерстных показателей.
Следующей задачей проекта была сборка генома овцы denovo и выявление новых SNP. Что обеспечит возможность создания SNP чипов более высокой плотности и массивов экспрессии покрытия всего генома [91].
В сотрудничестве с Illumina, консорциум запустил в начале 2009 года SNP50BeadChip, который широко использовался для исследования геномных ассоциаций (GWAS) и внедрения геномный отбор. Эта панель очень хорошо подходит для точного картирования моногенных признаков, и ряд их уже успешно охарактеризован. Например, микрофтальмия у представителей породы тексель, карликовость/ахондроплазия у породы шевиот и тексель, эпидермолиз соединительной ткани у немецких черноголовых овец, рогатость/комолость и т.д.
Интернет-ресурсы, осуществляющие хранение и обработку генетической информации по овцам
База данных «SheepGenomesDatabase» (SGD) была создана благодаря финансовой поддержке Министерства сельского хозяйства США (USDA AFRI).Проект SGD был запущен совместными усилиями (James Kijas), Utah State University (Noelle Cockett), Baylor College of Medicine Human Genome Sequencing Centre (Kim Worley), AgResearch (RudigerBrauning) и the Department of Economic Development, Jobs, Transport and Resources Victoria (Hans Daetwyler) [116].
Создание базы было инициировано Международным консорциумом по овцеводству (International Sheep Genomics Consortium) (рис. 23). Она дает сообществу исследователей в области геномики овец достоверную информацию и постоянно расширяется, благодаря интеграции последних достижений по уточнению референсной сборки генома овец v3.1.
Ключевыми задачами SGD являются:
1) сбор и обеспечение широкого доступа к данным по геному овец;
2) обеспечение пользователю доступа к ранее загруженным сиквенсам для поиска аналогий с вновь полученными;
3) загрузка данных по SNP и CNV из постоянно пополняющейся коллекции геномов.
SheepGenomesDB — это электронное хранилище, содержащее варианты последовательностей изпостоянно пополняющейся коллекции геномов овец. Благодаря согласованному контролю качества чтения, картирования, обнаружения вариантов и аннотаций, SheepGenomesDB делает доступными коллекции вариантов, полученные стандартным способом.
Эти варианты в коллекции содержат SNP, CNV и инсерции/делеции, расположенные на референсной сборке генома версии 3.1. В будущем эти варианты будут переноситься в последующие версии референсного генома. Важно отметить, что в SheepGenomesDB осуществляется поиск вариантов по расположению на хромосоме, по результатам аннотирования SNP или по породам, представляющим интерес.
Рисунок 23 – Общий вид базы данных геномов овец Sheep Genomes DB
Создатели SheepGenomesDB рассматривают эту возможность как значительное удобство для исследователей, заинтересованных в обнаружении генных мутаций, и полногеномных исследованиях, а также для специалистов, занимающихся исследованиями эволюции генома, одомашнивания и последствий отбора.
Существует возможность пополнения базы данных. Подробные инструкции для загрузки новых последовательностей представлены на сайте.
3.5.3 Информационное обеспечение исследований генома свиней
В настоящее время молекулярные методы анализа генотипа очень широко используются в характеристике диких видов животных и практическом животноводстве многих стран. Используя их, можно решать проблемы генетической дифференциации пород и подвидов кабанов, выявлять или уточнять таксономические отношения между различными подвидами диких свиней, а также устанавливать степень родства между дикими и домашними свиньями. Для характеристики генотипа животных используют как микросателлитные последовательности, так и селекционно значимые SNPs (single nucleotide polymorphism). Микросателлиты представляют собой короткие (от 100 до 350 п.н.) тандемно расположенные участки ДНК, обладающие высокой степенью полиморфизма. Эти маркеры известны под несколькими названиями: микросателлиты, STMS (sequence tagged microsattelite site), STR (short tandem repeat), SSR (simple sequence repeat). Их применяют для определения степени гетерозиготности малочисленных популяций, пород, консолидированности линий и групп, раннего прогнозирования потенциала продуктивности животных. Из-за высокой степени полиморфизма микросателлитные маркеры часто используют при оценке генетических расстояний между различными популяциями домашних свиней и их дикими предками. В научной литературе представлены результаты целого ряда исследований, посвященных молекулярно-генетической характеристике популяций кабанов, обитающих в Азии, Америке, Западной Европе, Австралии и Папуа Новой Гвинее. Для оценки генетического потенциала свиней непосредственно на уровне генотипа и для характеристики различных пород вида Sus scrofa проводятся эксперименты по выявлению информативных SNPs и разработке систем ДНК-анализа генов, влияющих на проявление селекционно и экономически значимых признаков [106].
Sus scrofa представляет собой наиболее распространенный вид сельскохозяйственных животных с наиболее широким ареалом. По данным ФАО, в 2016 году в мире насчитывалось более 730 пород и линий свиней, из которых две трети разводятся в Китае и Европе, а более 270 считаются редкими. В то же время 58 пород (включая 25 региональных и 33 международных) зарегистрированы, как распространенные, то есть встречающиеся более чем в одной стране. На сегодняшний день в мире наиболее распространенными являются пять международных пород: крупная белая (117 стран), дюрок (93 страны), ландрас (91 страна), гемпшир (54 страны) и пьетрен (35 стран). Дикие свиньи, или кабаны, благодаря крепкой конституции, высокой устойчивости к болезням, прекрасной приспособленности к условиям существования, долголетию и другим ценным признакам, служат постоянным источником генетических ресурсов при выведении новых пород.
Консорциум по исследованию генома свиньи
В 2003 году был создан консорциум по исследованию генома свиньи, а также начат глобальный проект по его секвенированию (SGSC) (http://www.piggenome.org/). Стратегия SGSC была основана на иерархическом секвенировании по Сэнгеру, которое позднее было дополнено данными NGS секвенирования, реализованного Illumina. Результатом этих усилий стала сборка и публикация проекта эталонной последовательности генома SusScrofa в 2012 году. В дополнение к этой эталонной последовательности генома, которая была получена от самки свиньи Дюрок, SGSC описаны геномы еще 48 свиней различных пород и диких кабанов. Одновременно с этим была опубликована, независимая сборка генома китайской мини-свиньи Wuzhishan, основанная на кратких чтениях (ридах) Illumina. За ней последовали дополнительные сборки de novo генотипов мини-свиньи Göttingen и тибетского дикого кабана в 2013 году. С тех пор геномы сотен отдельных свиней были повторно секвенированы для изучения вариаций, эволюции генома у этого вида, и в настоящее время доступно около 350 полных геномов https://gsejournal.biomedcentral.com/articles/10.1186/s12711-016-0204-2
В дополнение к получаемой важной информации об эволюционном развитии вида Sus Scrofa этот уникальной проект позволяет идентифицировать гены или геномные области локусов количественных признаков (QTLs), что позволит понять молекулярные механизмы, управляющие фенотипами, поможет объединить традиционные и биоинформационные методы для решения вопросов повышения устойчивости животных к заболеваниям и оперативного реагирования на изменяющиеся требования рынка [107]. На рисунке 24 изображена свиноматка китайской породы Erhualian — самая плодовитая порода свиней в мире.

Рисунок 24 – Erhualian, самая плодовитая порода свиней в мире
База данных PigVar
Свиньи являются хорошими моделями животных для проведения медицинских исследований и перспективным источником доноров органов для трансплантации. Технология секвенирования следующего поколения (NGS) привела к резкому увеличению объема геномных данных по свиньям. Однако ограниченный объем данных о геномных вариациях, предоставляемых dbSNP, и неконгруэнтные критерии, используемые для поиска вариаций, создают значительные препятствия для использования этих данных [119].
Статистические тесты различных эволюционных признаков SNP можно проводить с использованием http://www.ibiomedical.net/pigvar/), который является веб-ресурсом открытого доступа (рис. 25).
Рисунок 25 – Общий вид базы данных PigVar
PigVar собирает параметры вариаций, включая сводные списки местоположений вариаций в генах, кодирующих белок, и длинных межгенных некодирующих РНК (lincRNA), независимо от того, являются ли SNP синонимичными или несинонимичными, их наследственные и производные состояния, географические местоположения выборки, а также информацию о породе. База данных PigVar поддерживается и постоянно обновляется для использования в медицинских исследованиях с использованием свиней в качестве модели и в сельскохозяйственных исследованиях по свиноводству. С помощью PigVar пользователи могут искать информацию о SNP и структурных вариациях для конкретных образцов [119].
Структура и содержание базы данных PigVar
База данных PigVar включает SNP, структурные изменения и информацию о положительном отборе для свиней. Для каждого образца предоставляется информация о локации. В таблице 1 представлены статистические условия для положительного отбора в базе данных PigVar
Таблица 10 – Статистические условия для положительного отбора в базе данных PigVar
| Название статистического метода | Аббревиатура | Популяция 1 | Популяция 2 |
|---|---|---|---|
| Нуклеотидное разнообразие | PI | Chinese domestic pig | – |
| European domestic pig | – | ||
| Tibetan pig | – | ||
| Tajima’s D | TD | Chinese domestic pig | – |
| European domestic pig | – | ||
| Tibetan pig | – | ||
| Cockerham&Weir FST | FST FST_POP1_POP2 | Chinese domestic pig | Chinese wild boar |
| European domestic pig | European wild boar | ||
| Tibetan pig | Chinese domestic pig |
Продолжение таблицы 10
| Название статистического метода | Аббревиатура | Популяция 1 | Популяция 2 |
|---|---|---|---|
| Составной коэффициент вероятности для разных популяционных групп | XP-CLR XP_CLR_POP1_POP2 | Chinese domestic pig | Chinese wild boar |
| European domestic pig | European wild boar | ||
| Tibetan pig | Chinese domestic pig | ||
| Cross-population extended haplotype homozygosity | XP-EHH XP_EHH_POP1_POP2 | Chinese domestic pig | Chinese wild boar |
| European domestic pig | European wild boar | ||
| Tibetan pig | Chinese domestic pig |
PigVar предоставляет сигнатуры для выбора китайских домашних свиней, тибетских свиней и европейских домашних свиней.
На рисунке 26 показан алгоритм формирования базы данных PigVar.
Рисунок 26 – Источники данных и анализ конвейера, использованного для построения PigVar
Инструменты работы с базой данных PigVar
Высококачественные SNP, структурные изменения, а также их аннотации и оценки выбора могут быть обработаны скриптами Python и помещены в базу данных MySQL (5.1.66) (http://www.mysql.com). Для визуализации, поиска и загрузки данных можно использовать Java Server Pages (JSP, Java v1.6). Для визуализации данных на основе хромосом можно использовать Gbrowse (v2.55).
PigVar использует серию удобных интерфейсов для отображения результатов. Чтобы облегчить доступ к базе данных, предоставляется загружаемое руководство пользователя. Вкратце, PigVar имеет пять основных функций: поиск данных, просмотр, поиск SNP и структурных изменений и поиск хозяйственно ценных показателей [119].
Для SNP пользователи могут просматривать отдельные образцы SNP, как в виде таблиц в текстовом формате, так и на основе хромосом в GBrowse. Таблицы содержат идентификатор dbSNP.
Удобные функциональные возможности PigVar должны позволять исследователям получать подробную информацию о конкретных SNP, включая местоположение в генах, кодирующих белки или lincRNA, синонимические или несинонимичные замены, наследственное или производное состояние, а также связанные с ними положительные оценки отбора. Использование базы данных PigVar может интенсифицировать медицинские исследования, используя свиней в качестве модели, и усилить геномные исследования в свиноводстве.
Выводы
В настоящее время активно развивается множество информационных ресурсов для молекулярно-генетических исследований, в первую очередь это базы данных, связанные с описанием отдельных элементов молекулярно-генетических систем и процессов (гены, белки, их функции и структура и т. д.). Развитие баз данных позволяет облегчить решение поставленных задач. Для создания информационного пространства, обеспечивающего сравнительно полное покрытие рассматриваемой области, необходимо интегрировать разноплановые базы, количество которых измеряется двумя-тремя десятками.
Молекулярно-генетических баз данных большое количество, многие имеют свой формат хранения данных и средства доступа к содержащейся в ней информации. Возникает задача создания стандартов и программных средств, которые дадут возможность пользователю быстро находить информацию на основе компьютерного анализа многих баз данных.
Заключение
В результате проведенных исследований получены новые данные по результатам молекулярно-генетических исследований овец отечественных пород (волгоградская, карачаевская).
На основе полногеномного генотипирования с использованием OvineSNP50 Genotyping BeadChip получены данные, характеризующие 54241 SNP, расположенные в геноме овец волгоградской породы. По результатам анализа были определены 40 SNP, ассоциированных с плодовитостью овец, а также определены 14 генов, которые могут быть ответственны за признаки, связанные с воспроизводительной функцией.
Следует отметить, что, несмотря на развитие геномных технологий, на сегодняшний день только через аннотацию генов мы можем предположить некий биологический смысл выявленных ассоциаций. Соответственно, наряду с растущей популярностью полногеномного генотипирования не теряют свою значимость и гены-маркеры продуктивности животных (гены-кандидаты QTL). На основе исследование полиморфизмов генов GDF9 (волгоградская порода) и GH (карачаевская порода) были определены эффекты аллельных вариантов этих генов на формирование продуктивных показателей овец.
Полученные результаты имеют высокую научную и практическую значимость с точки зрения сохранения биоразнообразия и совершенствования локальных пород. И могут внести вклад в разработку рациональных программ улучшения, сохранения, а также создания пород, способствующих с одной стороны экономической эффективности производства за счет высоких продуктивных показателей, а с другой стороны удовлетворяющих спрос на высококачественную продукцию.
Учитывая значимость исследований, можно говорить о необходимости их продолжения. В первую очередь следует расширить базу полногеномных исследований, а также разработать методологию и инструменты интеграции получаемых результатов с мировыми информационными ресурсами.
Список использованных источников
- Абенова, Ж.М. Продуктивные и биологические особенности местных коз Республики Калмыкия: дис. канд. с.-х. наук: 06.02.10 / Ж.М. Абенова. — Москва, 2017 — 106 с.
- Алексеева А.Е., Бруснигина Н.Ф. Возможности и перспективы применения методов массивного параллельного секвенирования в диагностике и эпидемиологическом надзоре за инфекционными заболеваниями // Москва, 2014. – №2.- С.3-4
- Амерханов, Х.А. Из истории российского овцеводства / Х.А. Амерханов, В.И. Трухачев, М.И. Селионова. — Ставрополь: ИП Мокринский Н.С., 2017. — 408 с.
- Астафьева, Е.Е. Характеристика овец романовской породы по микроядерному тесту / Е.Е. Астафьева, С.Н. Марзанова, Е.А. Комкова и др// Вестник РАСХН. – 2015. – № 1. – С. 33-35.
- Бальмонт, В.А. Проявление гетерозиса и возможности закрепления его при создании кроссбредного овцеводства [Текст] /В.А. Бальмонт, К.У. Меде-узбеков, А. Байжуманов //Доклады ВАСХНИЛ. — 1967. — № 7. — с. 28-29.
- Батожаргалов ,Д.Р. Откормочные и мясные качества овец забайкальской породы [Текст] / Д.Р. Батожаргалов, Б.Б. Цибиков, Ф.Д. Высочанский // Овцы. Козы. Шерстяное дело, 2014. — № 1, — с. 22-24.
- Бачевская, Л.Т. Переверзева В.В. Генетическая изменчивость популяций кеты Oncorhynchusketa (Walbaum) р. Ола (материковое побережье Охотского моря) // Чтения памяти академика К.В. Симакова: Тез.докл. Всерос. науч. конф. Магадан, 25–27 ноября 2009. Магадан: СВНЦ РАН, 2009. С. 156–157.
- Бессонов, Н.М. Мясная продуктивность чистопородного и помесного молодняка овец цигайской породы [Текст] / Н.М. Бессонов //Материалы Всероссийского совещания по координации селекционно-племенной работы в породах с.-х. животных. — ВНИИплем, 2013. — Вып. 2. — с. 215-222.
- Билтуев, С.И. Мясная продуктивность молодняка овец забайкальской тонкорунной породы и её помесей с новозеландскими корриделями [Текст] / Билтуев С.И., Матханов А.В., Бальжинимаева С.Е.//Овцы. Козы. Шерстяное дело, 2016. — № 4. — с 55.
- Брыков, В.А., Кирилова О.Н., Кухлевский А.Д. и др. Анализ изменчивости митохондриальной ДНК у кеты Oncorhynchus keta (Walbaum) в популяциях рек Приморья и Сахалина // Генетика. 2000. Т. 36. № 10. С. 1388–1393.
- Брыков, В.А., Полякова Н.Е., Прохорова А.В. Фило- географический анализ кеты Oncorhynchusketa (Walbaum) в азиатской части ареала, основанный на изменчивости митохондриальной ДНК // Генетика. 2003. Т. 39. № 1. С. 75–82.
- Воронцов, Н.Н. Генофонд диких баранов в отдаленной гибридизации / Н.Н. Воронцов, К.М. Гаспарян // Природа. – 1988. – № 4. – С.49- 53.
- Гладырь, Е.А. Изучение генома свиней (Sus scrofa) с использованием ДНК-маркеров / Е.А. Гладырь, Л.К. Эрнст, О.В. Костюнина. – Текст : непосредственный // Сельскохозяйственная биология. – 2009. — № 2. – С. 16-26.
- Глазко, В.И. Генетическая структура украинской горнокарпатской породы овец и её связь с генофондом исходных пород / В.И. Глазко // Доклады РАСХН. – 2000. – № 4. – С.30-31.
- Горбунов, А.Н. Эффективность скрещивания [Текст] /А.Н. Горбунов //Овцеводство, 1990 — №5 — с. 26-28.
- ГОСТ 25955-83 Животные племенные сельскохозяйственные. Методы определения параметров продуктивности овец. — М., 1984.
- Государственная программа развития сельского хозяйства и регулирования рынков сельскохозяйственной продукции, сырья и продовольствия на 2013 — 2020 годы: утв. постановлением Правительства Российской Федерации от 14.07.2012 г. № 717. Режим доступа: http://base.garant.ru/70210644.
- Государственный реестр селекционных достижений, допущенных к использованию. Породы животных: официальное издание / под ред. В.В. Лабинова, В.С. Волощенко – М.: ФГБНУ «Росинформагротех», 2015. – Том 2. – 188 с.
- Гречко, В.В. Молекулярные маркеры ДНК в изучении филогении и систематики / В.В. Гречко // Генетика. – 2002. — Т. 38, №8 – С. 1013-1033
- Григорян, Л.Н. Племенная база овцеводства России / Л.Н. Григорян, С.А. Хататаев // Овцы, козы, шерстяное дело. — 2016. — №1. — С. 2-4
- Давлетова, А.М. Мясная продуктивность молодняка эдилбаевских овец / А.М. Давлетова, Б.Б. Траисов, К.Г. Есенгалиев, Ю.А. Юлдашбаев, К.А.Куликова, Р.И. Кудияров, М.И. Донгак // Овцы, козы, шерстяное дело. — 2018. — № 4. — С. 24-25.
- Дейкин, А.В. Генетические маркеры в мясном овцеводстве / Дейкин А.В., Селионова М.И., Криворучко А.Ю., Коваленко Д.В., Трухачев В.И. // Вавиловский журнал генетики и селекции. 2016. — Т. 20. — № 5. — С. 576-583.
- Дунин, И.М. Порода и породообразование (Теоретические аспекты) / И.М.Дунин, С.К.Охапкин. – Лесные Поляны: Изд-во ВНИИплем,1999. – 48 с.
- Дупал, Т.А. Внутрипопуляционная морфологическая изменчивость узкочерепной полевки (Microtusgregalis, Rodentia, Arvicolinae) / Т.А. Дупал, С.А. Абрамов // Зоологический журнал – 2010. – Т. 89, № 7. – С. 850–861.
- Дюбин, A.M. Продуктивность и некоторые биологические особенности потомства отскрещивания тонкорунных маток с баранами мясной и мясошерстных пород [Текст] /A.M. Дюбин //Автореф. дис. канд. с.-х. наук. Ставрополь, 2014. — 24 с.
- Елькина, М.А. Популяционно-генетическая дифференциация монгольских овец, крупного рогатого скота, яков в условиях хронического действия экологического стресса / М.А. Елькина, Е.Е. Астафьева, Т.В. Карпушкина и др. // Известия ТСХА – 2011. – № 2. – С.134-138.
- Ельсукова, И.А. Сравнительная характеристика мясной продуктивности баранчиков бирликского и суюндукского внутрипородных типов эдильбаевской породы овец / И.А. Ельсукова, Ю.А. Юлдашбаев, И.Н. Сычева // Овцы, козы, шерстное дело. – 2010. – № 4. – С. 44-51.
- Ермаков, О.А. Изучение гибридизации четырех видов сусликов (Spermophilus: Rodentia, Sciuridae) молекулярно-генетическими методами / О.А. Ермаков, В.Л. Сурин, С.В. Титов, А.Ф. Тагиев, А.В. Лукьяненко, Н.А. Формозов // Генетика. –- 2002. – Т. 38, № 7. – С. 950–964.
- Ерохин, А.И. Овцеводство [Текст] / А.И. Ерохин, С.А. Ерохин // Москва-2014 — с – 61
- Ерохин, А.И. Романовская порода овец /А.И. Ерохин, Е.А. Карасев. – М.: Изд-во МГУП, 2001. – 119 с.
- Ерохин, А.И. Эффективность промышленного скрещивания мериносов с мясошерстными баранами [Текст] / А.И. Ерохин, Ю.А. Юлдашбаев // Известие ТСХА – 1999 — вып. 4 — с. 144-153.
- Ефимова, Н.И. Качественная оценка мясной продуктивности молодняка овец разного происхождения / Ефимова Н.И., Завгородняя Г.В., Шумаенко С.Н., Штельмах А.И. // Овцы, козы, шерстяное дело. — 2012. — №2. — С. 45.
- Ефимова, Н.И. Мясная продуктивность потомков от баранов пород советский меринос и австралийский мясной меринос / Ефимова Н.И., Завгородняя Г.В. // Сборник научных трудов Ставропольского научно-исследовательского института животноводства и кормопроизводства. — 2011. Т. 1. — № 4-1. — С. 13-14.
- Информационно-аналитический портал «Союзное государство» [Электронный ресурс] – URL: http://www.soyuz.by/(дата последнего обращения 28.08.2015).
- Исмаилов, И.С. Откормочные качества чистопородных и помесных ярок /И.С. Исмаилов, В.А. Кущенко // Овцы, козы, шерстяное дело. — №3. – 2009. – с. 50-51.
- Исмаилов, И.С. Эффективность использования баранов породы австралийский мясной меринос в типе «Dohnemerino» на матках ставропольской породы /И.С. Исмаилов, П.Х. Амирова // Овцы, козы, шерстяное дело. — №4. – 2012. – с. 25-26.
- Колосов, Ю.А. Биотехнологические методы изучения полиморфизма гена гормона роста / Ю.А. Колосов, П.С. Кобыляцкий, Н.В. Широкова, Л.В. Гетманцева, Н.Ф. Бакоев // Дальневосточный аграрный вестник. — 2017. — №2(42). — С. 82 — 86.
- Колосов, Ю.А. Оптимизация техники проведения ПЦР-ПДРФ для генотипирования овец / Ю.А. Колосов, Н.В. Широкова, Л.В. Гетманцева, А.В. 104 Радюк, Н.Ф. Бакоев // Научный журнал КубГАУ.- 2015. — №113(09). Режим доступа: http://ej.kubagro.ru/2015/09/pdf/102.pdf, — свободный.
- Колосов, Ю.А. Полиморфизм гена (GDF9) у овец сальской породы. / Ю.А. Колосов, Л.В. Гетманцева, Н.В. Широкова // Ветеринарная патология. — № 3-4.- 2015. — С.78-81.
- Колосов, Ю.А., Абонеев В.В. Анализ основных положений выполнения программы «Развитие овцеводства и козоводства В России на 2012-2014 и до 2020 гг.»//Инновационные пути решения актуальных проблем АПК России : материалы всероссйской (национальной) научно-практической конференции- пос. Персиановский — Донской ГАУ, 2018. С. 222-227.
- Ли, Ч. Введение в популяционную генетику / Ч. Ли. – М.: Мир, 1978. – 560 с.
- Лухтанов, В.А. Молекулярно-генетические и цитогенетические подходы к проблемам видовой диагностики, систематики и филогенетики / В.А. Лухтанов, В.Г. Кузнецова // Журнал Общей Биологии. – 2009. – Т. 70, №5. – С. 415–437.
- Лухтанов, В.А. Принципы реконструкции филогенезов: признаки, модели эволюции и методы филогенетического анализа / В.А Лухтанов // Труды Зоологического института РАН. – 2013. – Приложение №2. – С. 39–52.
- Марзанов, Н.С. Аллелофонд тонкорунных пород овец / Н.С. Марзанов, Т.А. Магомадов // Сельскохозяйственная биология. – 1996. – № 4. – C.27- 35.
- Марзанов, Н.С. Аллелофонд у различных пород овец по микросателлитам / Н.С. Марзанов, М.Г. Насибов, М.Ю. Озеров, Ю. Кантанен // Дубровицы: Издво «13-Й ФОРМАТ», 2004. – 119 с.
- Марзанов, Н.С. Генетические маркеры в теории и практике разведения овец / Н.С. Марзанов, М.Г. Насибов, Л.К. Марзанова и др. – М.: ИЦ «Пионер», 2010. – 184 с.
- Марзанов, Н.С. Значение популяционно-генетических исследований в животноводстве / Н.С. Марзанов // Материалы I-й Всеросс. науч.-практ. конф. «Роль науки Южного Федерального Округа в развитии животноводства пореализации приоритетного национального проекта «Развитие АПК». – Черкесск, 2006. – С.115.
- Марзанов, Н.С. Методические рекомендации по использованию генетических маркеров в разведении овец / Н.С. Марзанов, М.Г. Насибов, А.М. Жиряков и др. – Дубровицы, 2004. – 44 с.
- Марзанов, Н.С. Современное определение понятия порода / Н.С. Марзанов, Ф.К. Апишева, Л.К. Марзанова и др. // Сельскохозяйственная биология. – 2007. – № 6. – С.16-23.
- Марзанов, Н.С. Сохранение биоразнообразия. Генетические маркеры и селекция животных /Н.С. Марзанов, Ю.В. Саморуков, Г.В. Ескини др. // Сельскохозяйственная биология. – 2006. – № 4. – С.3-19.
- Марзанов, Н.С. Эволюция и генная технология в тонкорунном овцеводстве / Н.С. Марзанов, Х.А. Амерханов, Л.К. Марзанова и др. – М.: Росинформагротех, 2012. – 176 с.
- Мигинский, Д.С. Технология семантической интеграции баз данных в системной биологии / Д.С. Мигинский, В.В. Лабужский, М.М. Лаврентьев-мл [и др.]. – Текст : непосредственный // Вычислительные технологии. – 2008. – Т. 13, № 6. — С. 102-120.
- Мина, М.В. Рост животных / М.В. Мина, Г.А. Клевезаль // М: Наука. – 1976. – 291 с.
- Моисеева, И.Г. Генофонды сельскохозяйственных животных: генетические ресурсы животноводства России (отв. ред. чл.-корр. РАН, проф. И.А. Захаров) / И.Г. Моисеева, С.В. Уханов, Ю.А. Столповский и др. – М.: Наука, 2006. – 478 с.
- Окулова, H.М. Межвидовая и внутривидова дифференциация лесных полевок рода Clethrionomys (Rodentia, Cricetidae) по данным изменчивости жевательной поверхности зуба МЗ / H.М. Окулова, Т.А. Андреева // Зоологический журнал. – 2008. – Т. 87, №8. – С. 991–1003.
- Петров, С.Н. Использование генной технологии для характеристики биологических особенностей и происхождения пород овец: дисс. канд. биол. наук: 03.00.23 / Петров Сергей Николаевич. – Дубровицы, 2008. – 126 с.
- Попов, Н.А. Аллелофонд пород крупного рогатого скота по ЕАВ – локусу. Справочный каталог. / Н.А. Попов, Г.В. Ескин. – М., 2000. – 299 с.
- Потапов, Е.Г. Явление переноса митохондриального генома красной полевки (Clethrionomysrutilus) крыжей (C. glareolus) на северо-востоке Европы / Е.Г. Потапов, Н.А. Илларионова, Т.А. Андреева, М.И. Баскевич, Н.М. Окулова, Л.А. Лавренченко, В.Н. Орлов // Доклады РАН. – 2007. – Т. 417, № 1. – С. 139–142.
- Рожков, Ю.И. Популяции, виды, эволюция / Ю.И Рожков, А.В. Проняев. М.: КМК, 2012. – 432 с.
- Саморуков, Ю.В. Сохранение и рациональное использование генофонда молочного скота Российской Федерации (Учебное пособие для слушателей повышения квалификации РАМЖ по курсу «Племенное дело в животноводстве») / Ю.В. Саморуков, Н.С. Марзанов. – Быково, 2002. – 49 с.
- Санников, С.Н. Филогеография и генотаксономия популяций вида Pinus sylvestris L. / С. Н. Санников, И. В. Петрова // Экология. – 2012. – №4. – С. 252–260.
- Серебровский, А.С. Генетический анализ / А.С. Серебровский. – М.: Наука, 1970. – 342 с.
- Степанов, В.А. Методы статистического анализа в популяционной и эволюционной генетике человека / В.А. Степанов, В.Н. Харьков, Е.А. Трифонова, А.В. Марусин. – Томск. 2014. – 99с.
- Столповский, Ю.А. Консервация генетических ресурсов сельскохозяйственных животных: проблемы и принципы их решения / Ю.А. Столповский. М.: Эребус, 1997. – 112 с.
- Столповский, Ю.А. Популяционно-генетические основы сохранения ресурсов генофондов доместицированных видов животных: дисс. докт. биол. наук: 03.02.07 / Столповский Юрий Анатольевич. – М.: 2010. – 339 с.
- Сулимова , Г.Е., Столповский Ю.А., Рузина М.Н., Захаров-Гезехус И.А. Мониторинг генофондов популяций животных в связи с задачами селекции и изучения филогении // Биоразнообразия и Динамика генофондов, Москва. 2008. С.211-214.
- Сулимова ,Г.Е. 2004. ДНК-маркеры в генетических исследованиях: типы маркеров, их свойства и области применения. Успехи современной биологии. 2004. Т. 124. № 3. С. 260-271.
- Сулимова, Г.Е. Полиморфизм длин рестрикционных фрагментов ДНК сельскохозяйственных животных: Методология, результаты и перспективы // Успехи соврем. Генетики. 1993. Т. 18. С.3-35.
- Сулимова, Г.Е., Федюнин А.А., Климов Е.А., Столповский Ю.А. Оценка генетического потенциала отечественного скота по признакам высокого качества мяса на основе ДНК-маркерных систем. Проблемы биологии продуктивных животных. 2011. № 1. С. 62-64.
- Трапезов, О.В. Доместикация, как одно из самых ранних интеллектуальных достижений человечества. К 140 — летию выхода на русском языке произведения Чарльза Дарвина «Прирученные животные и возделанные растения» / О.В. Трапезов // Философия науки. – 2007. – № 4. – С.104-129.
- Фоминых, М.А. Внутрипопуляционная изменчивость одонтометрических признаков рыжей полевки Myodesglareolus Schreber, 1780 на Среднем Урале / М.А. Фоминых, Е.А. Маркова, А.В. Бородин, Ю.А. Давыдова // Экология. – 2010 – Vol. 6. – С. 468–471.
- Хитринская, И.Ю. Генетическое разнообразие и взаимоотношения популяций Северной Евразии по полиморфным инсерциям ALU-элемента / И.Ю. Хитринская, В.Н. Харьков, М.И. Воевода [и др.] // Молекулярная биология. — 2014. — Т. 48. — № 1. — С. 69-80
- Хлесткина, Е.К. Молекулярные маркеры в генетических исследованиях и в селекции / Е.К. Хлесткина // Вавиловский журнал генетики и селекции. – 2013. – Т.17. – № 4/2. – С.1044-1054.
- Afifi, M. Sequencing and Polymorphism detection in growth hormone gene in Najdi Sheep and their association with milk production / M. Afifi, A. Alkaladi, N. M. Kadasa, S. Saddick, M. Baeshen, A. Al-Farga, F.M. Aqlan, Y.Q. Almulaiky, M. Alkhaled, A.A.M. Alayafi, H.A. Ali, M.K. Warsi, A.S. Allogmani, A. Abdelazim // Indian J. Anim. Res. – 2008. — Open access: https://arccjournals.com/journal/indian-journal-of-animal-research/B-920.
- Anthony, R.V., Liang R., Kayl E.P., Pratt S.L. The growth hormone/ prolactin gene family in ruminant placentae. J. Reprod. Fertil. Suppl. 1995;49:83-95.
- Asadi, N., Nanekarani Sh. and Khederzadeh S. 2014. Genotypic frequency of calpastatin gene in lori sheep by polymerase chain reaction-restriction fragment length polymorphism (PSR-RFLP) metod. African Journal of Biotechnology. 13(19): 1952-54.
- Barzegari, A, S. Atashpaz, K. Ghabili, Z. Nemati, M. Rustaei, R. Azarbaijani, 2010. Polymorphism in GDF9 and BMP15 associated with fertility and ovulation rate in Moghani and Ghezel sheep in Iran. Reprod Domest., 45: 666-669.
- Bi, P., Kuang S. Meat science and muscle biology symposium: Stemcell niche and postnatal muscle growth. J. Anim. Sci. 2012;90(3):924-935.
- Bodensteiner, K.J., C.M. Clay, C.L. Moeller, H.R. Sawyer, 1999. Molecular cloning of the ovine growth/differentiation factor-9 gene and expression of growth/differentiation factor-9 in ovine and bovine ovaries. Biol. Reprod., 60: 381-386.
- Boman, I.A., Klemetsdal G., Nafstad O., Blichfeldt T., Våge D.I. Impact of two myostatin (MSTN) mutations on weight gain and lamb carcass classification in Norwegian White Sheep (Ovis aries). Genet. Sel. Evol. 2010;42:4.
- Busboom, J.R., Wahl T.I., Snowder G.D. Economics of callipyge lamb production. J. Anim. Sci. 1999; 77(Suppl. 2):243-248.
- Byrne, K., Vuocolo T., Gondro C., White J.D., Cockett N.E., Hadfield T., Bidwell C.A., Waddell J.N., Tellam R.L. A gene network switch enhances the oxidative capacity of ovine skeletal muscle during late fetal development. BMC Genomics. 2010;11(1):378.
- Caiment, F., Charlier C., Hadfield T., Cockett N., Georges M., Baurain D. Assessing the effect of the CLPG mutation on the microRNA catalog of skeletal muscle using high-throughput sequencing. Genome Res. 2010;20(12):1651-1662.
- Carpenter, C.E., Cockett N.E. Histology of longissimus muscle from 2-week-old and 8-week-old normal and callipyge lambs. Can. J. Anim. Sci. 2000;80(3):511-514.
- Carpenter, C.E., Rice O.D., Cockett N.E., Snowder G.D. Histology and composition of muscles from normal and callipyge lambs. J. Anim. Sci. 1996; 74(2):388-393.
- Chen, E.Y., Liao Y.C., Smith D.H., Barrera-Saldaña H.A., Gelinas R.E.,Seeburg P.H. The human growth hormone locus: nucleotide sequence, biology, and evolution. Genomics. 1989; 4(4):479-497.
- Childers, C.P. Bovine Genome Database: integrated tools for genome annotation and discovery / C. P. Childers [et al.]. — Text : electronic // Nucleic Acids Res. – 2011. — Vol. 39. — Database issue: D830–D834. — doi:10.1093/nar/gkq1235. — URL: https://www.ncbi.nlm.nih.gov/pubmed/21123190
- Chu, MX, et al. GDF9 as a candidate gene for prolificacy of Small Tail Han sheep. Mol Biol Rep, 2011 Nov. PMID 21184179.
- Chung, H.Y., Davis M.E., Hines H.C. A DNA polymorphism of the bovine calpastatin gene detected by SSCP analysis. Anim. Genet.1999; 30(1):80.
- Chung, H.Y., Davis M.E., Hines H.C. Genetic variants detected by PCR-RFLP in intron 6 of the bovine calpastatin gene. Anim. Genet. 2001; 32(1):53
- Cockett, N.E. Analysis of the sheep genome / N.E/ Cockett, T.L. Shay, M. Smit. — Text : electronic // Physiol Genomics. – 2001. – Vol. 7. – P. 69–78. — URL: https://www.semanticscholar.org/paper /Analysis-of-the-sheep-genome.-Cockett-Shay/baf83ab632c93021720661bc0b90884ef4ed2548
- Davis, G.H., G.W. Montgomery, A.J. Allison, R.W. Kelly, A.R. Bray, 1982. Segregation of major gene influencing fecundity in progeny of Booroola sheep. New Zealand Journal of Agricultural Research, 25: 525-529.
- Deykin, A.V., Selionova M.I., Krivoruchko A.Yu. Kovalenko D.V., Truhachev V.I. Genetic markers in sheep meat breeding Вавиловский журнал генетики и селекции (20 ) 5, 2016.
- Eghbalsaied, S., F.R. Khorasgani, H.-R. Amini, M. Farahi, M. Davari, A. Pirali, S. Pourali, M. Vatankhah, M. Rostami, H. Atashi, 2017. Variant GDF9 mRNA is likely not the main cause of larger litter size in Iranian Lori-Bakhtyari, Shal, Ghezel, and Afshari sheep breeds. Arch. Anim. Breed., 60: 119-129.
- Elsik, C.G. Bovine Genome Database: new tools for gleaning function from the Bos taurus genome / C.G. Elsik, D.R. Unni, C.M. Diesh [et al.]. — Text : electronic // Nucleic Acids Res. – 2016. Vol. 4. — Issue 44. –P. 834-839. — doi:10.1093/nar/gkv1077. — URL: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4702796/
- Elsik, С.G. The genome sequence of taurine cattle: a window to ruminant biology and evolution.» Bovine Genome Sequencing and Analysis Consortium. / C.G. Elsik [et al.]. — Text : electronic //Science. – 2009. – Vol. 24. – Issue 324(5926). – P. 522–528. — doi :10.1126/science.1169588. — URL: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2943200/
- Frank J Steemers et al. (2006) Whole-genome genotyping with the single-base extension assay. Nature Methods 3, 31 – 33
- Georgieva, S., Hristova D., Dimitrova I., Stancheva N., Bozhilova-Sakov M.. 2015. Molecular analysis of ovine calpastatin (CAST) and myostatin (MSTN) genes in Synthetic Population Bulgarian Milk sheep using PCR-RFLP. J. BioSci. Biotechnol. 4(1). p. 95-99.
- Getmantseva, L., A. Kolosov, M. Leonova, S. Bakoev, A. Klimenko, A. Usatov, A. Radyuk, V. Vaselenko, M. Makarenko, N. Bakoev, 2017. Polymorphisms in obesity-related leptin gene and its association with reproductive traits of sows. Bulg. j. Agric. Sci., 23: 843–850.
- Gorlov, I.F., Association of the growth hormone gene polymorphism with growth traits in Salsk sheep breed / I.F. Gorlov, Yu.A. Kolosov, N.V. Shirokova, L.V. Getmantseva, M.I. Slozhenkina, N.I. Mosolova, N.F. Bakoev, M.A. Leonova, A.Yu. Kolosov, E.Yu. Zlobina // Small Ruminant Research. — 2017. — Vol. 150. — P. 11–14.
- Gorlov, I.F., Yu.A. Kolosov, N.V. Shirokova, L.V. Getmantseva, M.I. Slozhenkina, N.I. Mosolova, N.F. Bakoev, M.A. Leonova, A.Yu. Kolosov, E.Yu. Zlobina, 2018. GDF9 gene polymorphism and its association with litter size in two Russian sheep breeds. Rendiconti Lincei. Scienze Fisiche e Naturali, 29: 61–66.
- Groenen, M.A.M. A decade of pig genome sequencing: a window on pig domestication and evolution / Martien A.M. Groenen.- Text : electronic // Genet Sel Evol. — 2016. – Vol. 48. – Nu. 23. — doi:10.1186/s12711-016-0204-2. — URL: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4812630/
- Hagen, D.E. Bovine Genome Database: Tools for Mining the Bostaurus Genome / D.E. Hagen, D.R. Unni, A. Tayal, G.W. Burns G., C.G. Elsik. — Text : electronic // In book: Kollmar M. (eds) Eukaryotic Genomic Databases. Methods in Molecular Biology. Vol. 1757. – P. 211-249. — Humana Press :New York : NY, 2018. — doi: 10.1007/978-1-4939-7737-6_9. — URL: https://www.ncbi.nlm.nih.gov/pubmed/29761461
- Hanrahan, J.P., S.M. Gergan, P. Mulsant, M. Mullen, G.H. Davis, R. Powell, S.M. Galloway, 2004. Mutations in the genes for oocyte derived growth factors GDF9 and BMP15 are associated with both Increased ovulation rate and sterility in Cambridge and Belclare Sheep (Ovis aries). Biol. Reprod., 70: 900-909.
- Hiendleder, S, Kaupe B, Wassmuth R, Janke A. 2002. Molecular analysis of wild and domestic sheep questions current nomenclature and provides evidence for domestication from two different subspecies. Proc R Soc Lond B; 269: 893-904.
- Hu, Z-L. Animal QTLdb: an improved database tool for livestock animal QTL/association data dissemination in the post-genome era / Z-L. Hu, C.A. Park, X-L. Wu, J.M. Reccy. — Text : electronic // Nucleic Acids Res. -2013. — Vol. 41. — P. 871–879. — doi: 10.1093/nar/gks1150. — URL: https://manualzz.com/doc/32021784/animal-qtldb—an-improved-database-tool-for-livestock
- Hu, Z-L. Developmental progress and current status of the Animal QTLdb / Z-L. Hu, C.A. Park, J.M. Reccy. — Text : electronic // Nucleic Acids Research. – 2016. — Vol. 44. – P. 827–833. — doi: 10.1093/nar/gkv1233. — URL: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4702873/
- Juengel, J.L., G.H. Davis, K.P. McNatty, 2013. Using sheep lines with mutations in single genes to better understand ovarian function. Reproduction, 146: 111-123.
- Khan, S.-ul-H., Riaz M.N., Ghaffar A. and Ullah M.F. 2012. Calpastatin (CAST) Gene Polymorphism and its Association with Average Daily Weight Gain in Balkhi and Kajli Sheep and Beetal Goat Breeds. Pakistan J. Zool. 44(2): 377-382.
- Kolosov, Yu., Getmantseva L., Shirockova N. 2013. Sheep Breeding Resources in Rostov Region .World Applied Sciences Journal. 23(10): 1322-1324.
- Kevin, L Gunderson, et al. (2005). A genome-wide scalable SNP genotyping assay using microarray technology. Nature Genetics37, 549 — 554
- Lacroix, M.C., Devinoy E., Cassy S., Servely J.L., Vidaud M., Kann G. Expression of growth hormone and its receptor in the placental and feto-maternal environment during early pregnancy in sheep. Endocrinology. 1999; 140(12):5587-5597
- Malewa A 2009 Sifat kualitatif eksternal domba Donggala pada tiga lokasi di Sulawesi Tengah. Agrisains 10(2): 101-109.
- Moradband, F., G. Rahimi, M. Gholizadeh, 2011. Association of polymorphisms in fecundity genes of GDF9, BMP15 and BMP15-1B with litter size in Iranian Baluchi sheep. Asian Austral. J. Anim., 24: 1179–1183.\
- Noor, R R, Djajanegara A and SCHÜLER L 2001 Selection to improve birth and weaning weight of Javanese fat tailed sheep. Arch. Tierz. Dummerstorf, 44 (6): 649-659.
- Oddy, H. Sheepgenomics and the international sheep genomics consortium / Hutton Oddy, Brian Dalrymple, John McEwan [et al.]. — Text : electronic / Proc. Assoc. Advmt. Anim. Breed. Genet. – 2007. — Vol. 17. – P. 411-417. — DOI: 10.13140/2.1.2937.2806. — URL: https://www.researchgate.net/publication/265597060_SHEEPGENOMICS_AND_THE_INTERNATIONAL_SHEEP_GENOMICS_CONSORTIUM
- Palmer, B.R., Roberts N., Hickford J.G., and Bickerstaffe R. 1998. Rapid Communication: PCR-RFLP for Mspi And Ncoi in the Ovine Calpastatin Gene. J.Anim. Sci., 76: 1499-1500.
- Reese, J.T. Bovine Genome Database: supporting community annotation and analysis of the Bos taurus genome / J.T. Reese.,C.P. Childers, J.P. Sundaram [et al.]. — Text : electronic // BMC Genomics. – 2010. — Nov 19. — doi: 10.1186/1471-2164-11-645. — URL: https://bmcgenomics.biomedcentral.com/articles/10.1186/1471-2164-11-645
- Zhou ZY, Li A, Otecko NO, et al. PigVar: a database of pig variations and positive selection signatures. Database (Oxford). 2017;2017:bax048. doi:10.1093/database/bax048
