Титульный лист и исполнители
РЕФЕРАТ
Отчет 143 с., 5 табл., 26 рис., 104 источника литературы.
СКОТОВОДСТВО, СТРУКТУРА ГЕНОФОНДА, МОЛЕКУЛЯРНО-ГЕНЕТИЧЕСКИЕ МЕТОДЫ, МУТАЦИИ, МАРКЕР, ГЕН, ИДЕНТИФИКАЦИЯ, ГЕНОТИПИРОВАНИЕ, АЛЛЕЛИ, СЕЛЕКЦИЯ, ОДНОНУКЛЕОТИДНЫЕ ПОЛИМОРФИЗМЫ (SNP), ДОСТОВЕРНОСТЬ ПРОИСХОЖДЕНИЯ, НАСЛЕДУЕМЫЕ ЗАБОЛЕВАНИЯ, ПРОДУКТИВНОСТЬ, ЛАКТАЦИЯ, ПЛОДОВИТОСТЬ, ВОСПРОИЗВОДСТВО, ПАНЕЛЬ TRUSEQ BOVINE PARENTAGE SEQUENCING PANEL ILLUMINA (США), БИОИНФОРМАЦИОННЫЙ АНАЛИЗ, ПРОГРАММНЫЙ ПРОДУКТ, ИНТЕРФЕЙС, ПРЕДСТАВЛЕНИЕ,
2D-ДИАГРАММА («КОВЁР»), ИНФОРМАЦИОННЫЕ СИСТЕМЫ
Целью работы было создание рекомендации для внедрения в хозяйствах края разработок по проведению генетического анализа поголовья на современном уровне в соответствии с рекомендациями Международного общества по изучению генетики животных (ISAG), в том числе для создания устойчивого селекционного ядра; проведение анализа статистически достоверной группы животных молочного направления продуктивности суммарным количеством до 300 гол., а также анализа используемых в хозяйстве лидере отросли генотипов спермодоз, используемых для получения потомства; использовать для анализа результаты таргетного секвенирования с использованием панели TruSeq Bovine Parentage Sequencing Panel фирмы Illumina (США); разработка программного продукта, результатом применения которого будет выстраивание ранжированного списка наиболее предпочтительных быков-производителей, относительно индивидуальных генетических характеристик матери; применить данный программный продукт в тестовом режиме в хозяйстве-лидере отросли для максимально эффективного индивидуального подбора отца-производителя.
За отчетный период методом таргетного секвенирования с использованием панели TruSeq Bovine Parentage Sequencing Panel Illumina (США) проведено генотипирование модельных групп КРС (условно низкопродуктивной, высокопродуктивной) и группы телок в возрасте 4,5 мес с неисследованным уровнем продуктивности до начала первой лактации. Исследовано в общей сложности 300 гол. КРС голштинской породы. Для каждого животного определено значение более 250-ти однонуклеотидных замен (SNP), отвечающих за достоверность происхождения и родства, а также за проявление генетически наследуемых заболеваний и факторов продуктивности.
В соответствии с техническим заданием разработаны две компьютерные программы (продукта). Программные продукты полностью работоспособны
и протестированы на реальных экспериментальных базах данных. Программа «Just Append Cows» предназначена для обработки файлов первичных данных секвенирования на приборе MiSeq (Illumina) и формирования индивидуальных генетических паспортов каждого исследованного животного. Программа «Covers» позволяет на основе первичных данных секвенирования сформировать цветную 2D-диаграмму, позволяющую оперативно оценить генетические характеристики и аллельную структуру модельных групп КРС и стада в целом. Описываемая программа дает возможность также вызвать интерактивные окна с описанием соответствующих болезней или факторов продуктивности.
Кроме того, данный тип графического представления данных секвенирования КРС с использованием панели TruSeq Bovine Parentage Sequencing Panel Illumina (США) предложен нами впервые и не имеет российских и зарубежных аналогов.
За отчетный период все исследованные в рамках проекта животные паспортизированы в полном объеме и построены 2D-диаграммы, графически отражающие аллельные профили всего стада КРС. Использование обеих программных продуктов позволяет произвести индивидуальный подбор спермодоз при искусственном осеменении с учетом более 250-ти SNP и дать рекомендации на основе комплексных генетических исследований поголовья для обоснованного формирования устойчивого племенного ядра хозяйства.
ВВЕДЕНИЕ
Таргетное секвенирование, лежащее в основе конкретной технической реализации, примененной нами, дает возможность остановить внимание на конкретных участках (областях) генома или отдельных генов. Этот метод применяется в том случае, когда у изучаемого животного подозревается наличие какого-либо генетического заболевания, для которого известны гены или конкретные мутации, а также с целью генетической паспортизации конкретных животных и установления представленности различных аллелей в определенном стаде или группе животных. Аналогично, путем сложных вычислений и статистического анализа полностью секвенированных геномов, проводится поиск однонуклеотидных полиморфизмов (Single nucleotide polymorphism, SNP), не связанных непосредственно с проявлением в фенотипе, но используемых для доказательства достоверности происхождения/родства.
В случае таргетного секвенирования нет необходимости исследовать все гены, достаточно установить структуру только одного гена или некоторых его участков, так называемых горячих точек, где наличие мутации наиболее вероятно. Как правило, для достижения большинства текущих задач современного товарного животноводства необходимо ответить на ограниченное число вопросов, наиболее честно возникающих у специалистов хозяйств, специализирующихся на производстве молока или мяса. К этим вопросам, прежде всего, относится степень гетерозиготности/гомозиготности стада (близость родства, которая может привести к увеличению представленности генетических отклонений и уродств), наличие генетически наследуемых заболеваний или генов, повышающих предрасположенность к болезням, наличие вариантов генов, отвечающих за экономически значимые признаки. Причем в этом случае исследуемые варианты аллелей, как правило, не связаны с развитием патологического состояния, являются вариантами нормы, просто те или иные замены в кодируемых такими генами белках, являются более предпочтительными у пород мясного направления селекции, а другие варианты генов более предпочтительны для коров молочного направления селекции.
Из имеющихся на рынке коммерчески доступных решений для осуществления таргетного секвенирования КРС в практических целях наиболее применима, по нашему мнению, панель TruSeq Bovine Parentage Sequencing Panel (Illumina, США).
Вместе с тем, выбором платформы для исследования признаков и нахождения конкретных SNP, позволяющих доказать степень родства, достоверность происхождения, наличие или отсутствие генетических заболеваний, а также установить тип аллелей, отвечающих за реализацию экономически значимых признаков не ограничиваются запросы хозяйств. Необходимо также разработать программные продукты, которые с одной стороны позволили бы оперативно получить генетический паспорт животного с перечислением всех установленных SNP, с другой представить полученный массив экспериментальных данных в виде понятном специалисту хозяйства, визуализированном до степени возможности наглядного и интуитивно понятного использования. Такую задачу решает разработанный нами программный продукт, позволяющий генерировать цветовые 2D-диаграммы, визуализирующий распределение гомозигот по алельным и доменантным признакам, а также гетерозигот в исследуемом стаде
с произвольным количеством голов. Это позволяет специалисту с первого взгляда оценить степень присутствия нежелательных или, наоборот, желательных генетических вариантов и вести мероприятия по осеменению, опираясь на эти данные. Так, сопоставление графических профилей генотипов животных, отраженных в 2D-диаграмме группы телок с графическим представлением по генотипам отдельных образцов спермодоз, позволяет подобрать оптимальное сочетание аллелей предков и исключить или свести к минимуму вероятность закрепления в ядре стада наследственных заболеваний. Этот подход не является чем-то абсолютно новым, более того, в последние годы подобного рода услуги активно рекламируются для человека. Этичность такого «генетически детерминированного» подбора пар в случае людей спорна, однако в ветеринарии она становится, и станет в ближайшем будущем «золотым стандартом». Наша работа, выполненная в продолжении годичного периода, всего лишь позволяет оптимизировать и автоматизировать анализ первичных данных секвенирорвания
с использованием панели TruSeq Bovine Parentage Sequencing Panel, а разработанные программные продукты предназначены для генерации генетических паспортов животных единого образца (содержащих информацию о 265 отдельных SNP) и визуализации данных при помощи построения цветовых 2D-диаграмм, которые могут быть использованы, в том числе и для быстрого подбора оптимальной спермодозы при искусственном осемениеии/оплодотвуорении.
1 ТЕОРЕТИЧЕСКАЯ ЧАСТЬ
1.1 Типы полиморфизма ДНК
В связи с развитием методов молекулярно-генетического анализа за прошедшие десятилетия было доказано, что до трети локусов эукариотического генома представлены аллельными вариантами со средней гетерозиготностью на особь вплоть до 12 % и более (Brookes, A. J., 1999). В природных популяциях эта изменчивость постоянно поддерживается c помощью известных микроэволюционных факторов – мутациями, миграцией генов, случайным генетическим дрейфом и естественным отбором. Генетический полиморфизм может быть выявлен на фенотипическом, биохимическом, хромосомном, молекулярном уровнях.
В случае молекулярно-генетического полиморфизма речь идет об изменениях в структуре ДНК, обусловленных различными типами мутаций: точечными мутациями, делециями и инсерциями от одного до большего числа нуклеотидов. Схема образования гаплотипов в случае нарушения аллельного равновесия на примере единичных точечных мутаций на локусе одной хромосомы показана на рисунке 1.
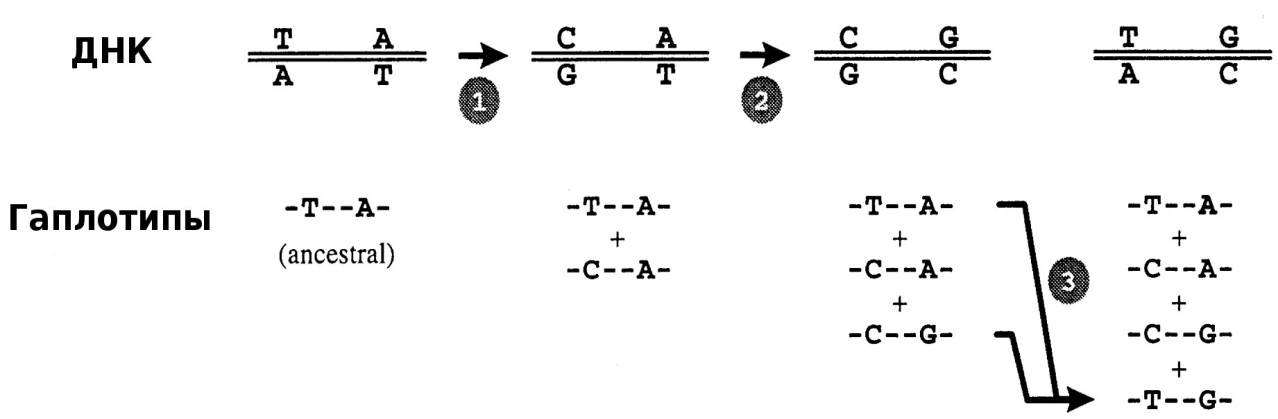
Рисунок 1 – Схема образования гаплотипов:
1 – точечная замена T↔C и образование 2-го возможного гаплотипа; 2 – точечная замена A↔G и образование 3-го возможного гаплотипа; 3 – вариант меойтической
рекомбинации, приводящей к образованию четырех гаплотипов [5]
После открытия явления полиморфизма ДНК в середине 80-х гг. для создания насыщенных генетических карт и маркирования QTL появился новый класс генетических маркеров. Огромную роль в их появлении сыграло развитие методов клонирования и рестрикции генов. Но открытие полимеразной цепной реакции (ПЦР) имело решающее значение. Суть метода состоит в том, что предназначенный для амплификации участок ДНК служит матрицей для синтеза in vitro комплементарной последовательности. Реакция катализируется Taq-полимеразой в присутствии двух олигонуклеотидных праймеров, комплементарных к последовательностям, окружающим анализируемый фрагмент. Число анализируемых фрагментов ДНК в каждом цикле удваивается, и после 30 циклов репликации количество исходной ДНК увеличивается в миллион раз.
Поскольку преобладающая часть эукариотического генома не вовлечена
в какие-либо известные и важные функции, соответствующие участки некодирующей ДНК обнаруживают уровни полиморфизма, многократно превосходящие данные, которые известны о полиморфизме генов. В случае структурных генов, которые колируют белки, изменения в структуре ДНК могут обуславливать изменения в аминокислотных последовательностях белков, и как следствие нарушения действия генов. Так, например, точечные мутации могут приводить
к появлению стоп-кодона в кодирующей последовательности, приводя к остановке трансляции и образованию белков с нарушениями их функциональности. Точечные мутации также могут приводить к замене одного аминокислотного остатка на другой, изменяя тем самым структуру молекулы белка, а, следовательно, и его функцию. Как и в случае обрыва белковой цепи, это может приводить к различным генетическим заболеваниям.
Основные типы полиморфизмов ДНК эукариотических геномов представлены в таблице 1 (Алтухов Ю. П., Салменкова Е. А., 2002).
1.1.1 Рестрикционные фрагменты ДНК
Геномная ДНК эукариот имеет существенные размеры, в частности, геном коровы составляет 3,1 млрд пар оснований (Van Eenennaam A. L., Weigel K. A., Young A. E., Cleveland M. A., J. C. M. Dekkers, 2014). Сайт-специфические ДНК-эндонуклеазы – это бактериальные ферменты, расщепляющие ДНК по определенным последовательностям, называемым сайтами узнавания. Наиболее известными среди этих ферментов являются эндонуклеазы рестрикции II типа, первая из которых (HindII) была обнаружена 45 лет назад, и которые сыграли большую роль в развитии генной инженерии, поскольку их использование позволило специфично проводить фрагментацию протяженных геномных ДНК. А затем клонировать участки генома в различные векторные конструкции (цит. по Ю. П. Алтухову, Е. А. Салменковой, 2002). Эти эндонуклеазы вступают
в реакцию лишь со специфическими состоящими из 4–6 пар оснований, деметилированными участками ДНК – сайтами узнавания, которые в гомологичных бактериальных ДНК защищены метильными группами.
Таблица 1 – Типы полиморфизма ДНК
| Характер изменчивости |
Причина возникновения полиморфизма |
Методы выявления |
| 1 | 2 | 3 |
| Полиморфизм длины рестрикционных фрагментов (RFLP) |
Нуклеотидные различия в сайтах рестрикции |
Разрезание цепи ДНК с помощью рестриктаз, электрофорез продуктов рестрикции, их визуализация |
| Минисателлиты, варьирующее число тандемных повторов (VNTR) |
Варьирующее число тандемно повторенных нуклеотидных последовательностей с размером повтора 10–100 нуклеотидов |
Разрезание цепи ДНК с помощью рестриктаз, электрофорез, Саузерн-блоттинг со специфической пробой к повторяющейся последовательности. Полилокусные пробы комплементарны к часто встречающимся в геноме повторам, однолокусные – к уникальным повторам |
| Микросателлиты, простые тандемные повторы (STR), простые нуклеотидные повторы (SSR) |
Варьирующее число тандемно повторенных коротких нуклеотидных последовательностей с размером повтора 1–6 нуклеотидов |
ПЦР-амплификация с праймерами, комплементарными уникальным последовательностям, фланкирующим семейство повторов, электрофорез продуктов |
| Полиморфизм фрагментов ДНК, амплифицированных с произвольными праймерами (RAPD) |
Нуклеотидные различия в сайтах связывания с праймерами |
ПЦР-амплификация случайных сегментов ДНК с использованием 10–20 членных нуклеотидных праймеров с произвольной нуклеотидной последовательностью, электрофорез продуктов амплификации |
| Продолжение таблицы 1 | ||
| 1 | 2 | 3 |
| Полиморфизм длины амплифицированных фрагментов (AFLP) |
Нуклеотидные различия в сайтах рестрикции и фланкирующих их областях |
Рестрикция с помощью двух рестриктаз, действующих на частые и редкие сайты рестрикции, присоединение олигонуклеотидных адаптеров, ПЦР-амплификация, электрофорез |
| Однонуклеотидный полиморфизм (SNP) |
Замены отдельных нуклеотидов в последовательности ДНК | Гибридизация меченых ПЦР-продуктов с микропанелями ДНК-проб для выявления вариантов, ДНК-чипы, Секвенирование ДНК-проб (фрагментов, полногеномное) |
Несмотря на то, что развитие ПЦР-технологий и методов полногеномного секвенирования несколько снизило интерес к использованию ферментов рестрикции, их значимость для научно-практических исследований остается высокой. Они находят широкое применение в полногеномном поиске молекулярных маркеров для исследований в области популяционной и адаптационной генетики. Геномы эукариот могут различаться по нескольким миллионам нуклеотидов, часть из которых входит в сайты узнавания коммерчески доступных ферментов. Как известно, полиморфизм ДНК вызывается несколькими причинами: либо точечными заменами, либо ошибками при репликации ДНК в виде инсерций или делеций протяженностью от одного до тысяч нуклеотидов. Все изменения в первичной структуре ДНК ведут к изменениям в длине фрагментов, образующихся под действием рестриктаз.
Сравнение секвенированных рестрикционных фрагментов из различных популяций организмов позволяет легко выявлять фрагменты, свойственные лишь одной из исследуемых групп и тем самым обнаружить нуклеотидные различия. При этом фрагменты используются в качестве простейших генетических маркеров. Основанный на этом подходе метод анализа получил название RFLP (Restriction Fragment Length Polymorphism).
Смесь фрагментов ДНК разделяют электрофорезом в агарозном или полиакриламидном геле и идентифицируют с помощью метки с последующей авторадиографией. Метод малоэффективен для разделения множества фрагментов. Для избирательного выявления в геле каких-либо определенных фрагментов ДНК используется гибридизация со специфической пробой методом Саузерна. Такая проба представляет нуклеотидную последовательность, комплементарную исследуемой области и снабженную радиоактивной или флуоресцентной меткой. Благодаря метке нужный фрагмент специфично визуализируется.
Первоначально для гибридизации с фрагментами ДНК, полученными
в результате действия рестриктаз, использовались универсальные мультилокусные пробы, которые содержали структуры, часто встречающиеся в геноме
в виде семейств многочисленных повторов. При использовании таких проб
в геле выявляется одновременно много сходных локусов, находящихся в гетерозиготном состоянии из-за большого числа аллелей. Эти сложные картины фрагментов ДНК, специфичные для отдельных особей, получили название «ДНК-фингерпринтов» или «фингерпринтов».
Несмотря на то, что было проведено несколько исследований на растениях с целью выявления QTL посредством сцепления с RFLP-маркерами (Paterson A. H., Lander E. S., Hewitt J. D., Paterson S., Lincoln S. E., Tanksley S. D., 1988), эти маркеры оказались не очень полиморфными у домашних животных.
1.1.2 Мини- и микросателлиты
С 1969 г. было известно, что ДНК высших организмов содержит много повторяющихся фрагментов. В 1989 г. сразу несколько лабораторий независимо друг от друга обнаружили, что короткие последовательности повторяющихся ДНК были полиморфными в отношении числа повторяющихся ДНК. Наиболее распространенной из этих повторяющихся последовательностей является poly-[TG], которая, как было установлено, очень часто встречается у всех высших видов (цит. по Дж. И. Веллеру, 2018).
Геном сельскохозяйственных животных насыщен (до 30 %) такого рода повторами, называемыми короткими тандемными (Shot Tandem Repeats – STR) или простыми структурными повторами (Single Sequence Repeats – SSR). Помимо выше упомянутого [TG]n, повторяющейся единицей может быть, например, мононуклеотид [A]n или динуклеотид [AC]n (Ellegren H., 2004; Mason, A. S., 2015). Но повторяющейся единицей может быть и тринуклеотид, и тетра, и т. д. Очень часто в одном и том же локусе на разных хромосомах присутствует разное количество таких повторов. Количество аллелей может быть значительно больше за счет изменения числа повторов «n» (см. таблицу 2). Следует отметить, что хотя каждый из таких локусов отличается в своих аллелях числом повторов, нуклеотидные последовательности по краям этих повторов для одинаковых локусов одинаковы. И если такой повтор попадает между двумя соседними сайтами какой-либо рестриктазы, то длины полученных рестрикционных фрагментов для разных аллелей будут различаться (Zane L., Bargelloni L., Patarnello T., 2002).
Особое внимание исследователей в семействах тандемных повторов в качестве генетических маркеров получили минисателлиты, состоящие из повторяющихся копий («мотива») длиной от 9–10 до сотни нуклеотидов каждая,
и микросателлиты, повторяющиеся копии которых обычно имеют длину от 1 до 4, иногда 6 нуклеотидов. Минисателлитный локус может насчитывать от двух до нескольких сотен повторов, микросателлитный локус – от 10 до 100 повторов. Эти маркеры – кодоминантные, т. е. гетерозиготный генотип всегда отличается от любой гомозиготы. Кроме того, микросателлиты почти всегда полиаллельны, т. е. в популяциях всегда присутствует более двух аллелей. Высокополиморфный характер и менделевский тип наследования делает микросателлиты идеальными ДНК-маркерами сельскохозяйственных животных.
Высокая достоверность и специфичность ДНК анализа может быть достигнута при использовании любых видов биологического материала: кровь, слюна, волосы, сперма, образцы мягких тканей. Для анализа микросателлитов требуется очень малое количество крови и какой-либо ткани организма, поэтому возможно прижизненное взятие образцов. Микросателлиты одинаковы
у близких видов. Что позволяет использовать одни и те же праймеры и сходные протоколы анализа.
В настоящее время микросателлитные ДНК-маркерные системы находят применение при решении различных фундаментальных и прикладных задач сельскохозяйственной биологии и биотехнологии таких как геномное картирование, характеристика генетической структуры популяции и степени инбредности, оценка генетических расстояний между линиями, породами и популяциями, филогенетические исследования, контроль происхождения (Эрнст Л. К., Зиновьева Н. А., 2008; Guichoux E., Lagache L., Wagner S., Chaumell P., Leger P., Lepais O., Lepoittevin C., Malausa T., Revardel E., Salin F., Petit R. J., 2011). Таким образом, технология ДНК-анализа микросателлитов становится незаменимым инструментом для решения задач селекции и целенаправленного повышения продуктивных качеств выращиваемых животных и является «золотым» стандартом современной-молекулярно-генетической идентификации животных, рекомендованным Международным обществом по изучению генетики животных (ISAG).
Наиболее распространенным методом анализа SSR является метод ПЦР,
с использованием праймеров, комплементарных уникальным последовательностям (доменам), которыми фланкирован каждый микросателлитный локус. Использование ПЦР для анализа микросателлитных локусов впервые было предложено в 1989 г. для анализа [CA]n[GT]n, которые являются одним из наиболее встречаемых мотивов повторов в геномной ДНК человека (Weber J. L., May P. E., 1989). Разделение продуктов амплификации проводили электрофорезом в полиакриламидном геле, что позволило резко повысить чувствительность и скорость анализа по сравнению с методами, основанными на гибридизации геномных блотов.
В настоящее время стандартной методикой анализа ДНК-микросателлитов с известными фланкирующими последовательностями является мультиплексный ПЦР с последующим разделением продуктов амплификации методом капиллярного электрофореза и их одновременной лазерной детекцией. Она отвечает современным требованиям молекулярно-генетического анализа: высокая информативность анализа, минимальные сроки исследования, простота проведения и низкая себестоимость исследования.
В практической части настоящей работы будут использоваться готовые отечественные современные решения для всех этапов генетического анализа. Компания «Гордиз» на сегодняшний день является единственным российским производителем продукции для молекулярно-генетической идентификации животных на основе микросателлитных маркеров ДНК (набор «COrDIS Cattle»).
ISAG в качестве стандартных маркеров рекомендовало использовать
11 локусов: ETH3, INRA023, TGLA227, TGLA126, TGLA122, SPS115, ETH225, TGLA53, BM2113, BM1824, ETH10. Кроме того, в набор фирмы Гордиз для определения молекулярно-генетической характеристики крупного рогатого скота с целью анализа родства и ДНК-индивидуализации животных на основе мультиплексного ПЦР-анализа локусов, содержащих STR, входят три дополнительных высокополиморфных микросателлитных локуса: CSSM66, ILSTS006
и CSRM60 (таблица 2).
Таблица 2 – Описание микросателлитных маркеров
| Название локуса |
Хромосомная локализация | Нуклеотидная последовательность единицы повтора |
| 1 | 2 | 3 |
| Основные | ||
| ETH3 | D19S2 | (GT)nAC(GT)6 |
| INRA023 | D3S10 | (AC)n |
| TGLA227 | D18S1 | (TG)n |
| TGLA126 | D20S1 | (TG)n |
| TGLA122 | D21S6 | (AC)n(AT)n |
| SPS115 | D15 | (CA)nTA(CA)6 |
| ETH225 | D9S2 | (TG)4CG(TG)(CA)n |
| TGLA53 | D16S3 | (TG)6CG(TG)4(TA)n |
| BM2113 | D2S26 | (CA)n |
| BM1824 | D1S34 | (GT)n |
| ETH10 | D5S3 | (AC)n |
| Дополнительные | ||
| CSSM66 | D14S31 | (AC)n |
| ILSTS006 | D7S8 | (GT)n |
| CSRM60 | D10S5 | (AC)n |
В стандартном наборе праймеры для ПЦР подбираются с учетом проведения амплификации всех 14-ти локусов в одной пробирке. Размер всех амплифицируемых ПЦР продуктов < 320 пар нуклеотидов (с учетом всех известных аллелей). Анализ результатов ПЦР обычно проводится методом капиллярного электрофореза с использованием автоматических генетических анализаторов
с лазериндуцированной флуоресцентной детекцией. В наборе COrDIS Cattle используется пять флуоресцентных красителей, характеризующихся разными длинами волн эмиссии для возможности одновременной детекции в разных каналах флуоресценции. Праймеры мечены четырьмя флуоресцентными красителями, детектируемыми в каналах Blue, Green, Yellow, Red. Стандарт длины S450 мечен пятым, флуоресцентным красителем и детектируется в отдельном канале Orange одновременно с продуктами ПЦР. С использованием этих реагентов для получения полного STR-профиля образца достаточно 0,2 нанограмм. Оптимальное количество – 0,5 нанограмм.
RAPD, AFLP. В отличие от микросателлитов или однолокусных минисателлитов, с помощью которых исследуются отдельные локусы генома, маркеры, обозначаемые как RAPD (Random Amplified Polymorphic DNA) и AFLP (Amplified Fragment Length Polymorphism), позволяют исследовать геном в целом, получая соответствующие фингерпринты.
Для анализа RAPD используют 10–20-членные нуклеотидные праймеры
с произвольно выбранными последовательностями. С их помощью амплифицируют анонимные участки ДНК, после чего амплифицированные фрагменты анализируют с помощью гель-электрофореза. Число и размер фрагментов зависят от длины и структуры произвольно выбранных праймеров. Комплементарные сайты связывания праймеров случайно распределены по геному, а полиморфизм в таких сайтах выражается в присутствии или отсутствии в геле соответствующих фрагментов (Алтухов Ю. П., Салменкова Е. А., 2002).
Метод AFLP основан на избирательной амплификации рестрикционных фрагментов геномной ДНК и их анализе. Фрагментацию ДНК обычно проводят двумя рестриктазами. Образующиеся «липкие» концы связывают с олигонуклеотидными адаптерами, которые и служат мишенью для отжига праймеров. Тем самым достигается избирательная амплификация только тех фрагментов, рестрикционные сайты которых фланкированы комплементарными нуклеотидами (Алтухов Ю. П., Салменкова Е. А., 2002; Grover A., Sharma P. C., 2016). Как и в случае метода RAPD полиморфизм фингерпринта проявляется в наличии или отсутствия конкретных полос в геле, но в отличие от RAPD обладает очень хорошей воспроизводимостью.
1.1.3 Полиморфизм единичного нуклеотидного сайта
SNP (Single Nucleotide Polymorphism) как новый тип маркеров стал использоваться в 1995 г. (цит. по A. J. Brookes, 1999). SNP обычно определяют как местоположение пары оснований ДНК, в которой частота появления наиболее распространенного основания пары ниже 99 %. В отличие от микросателлитов, которые имеют несколько аллелей, SNP, как правило, являются диаллельными, но гораздо более распространены во всем геноме, с оценочной частотой одного SNP на 300–500 пар оснований. В популяциях человека различия в нуклеотидной последовательности любых двух случайно выбранных особей встречаются с частотой примерно 1 на 1000 кб (Brookes A. J., 1999). Таким образом, SNP могут быть найдены в геномных областях, бедных микросателлитами. Вероятно, большинство полиморфизмов возникло в результате мутаций, закрепившихся в популяции. Наиболее часто встречающийся SNP – вариация оснований одного типа, т. е. пуринов или пиримидинов. SNP с аллелями пурин/пиримидин встречаются значительно реже. Частота встречаемости разных типов в геноме человека описывается следующим соотношением: A/G – 63 %, A/C – 17 %, G/C – 8 %, A/T – 4 %. На оставшиеся вставки и делеции приходится 8 % (Miller R. D., Taillon-Miller P., Kwok P.-Y., 2001).
Наибольший практический интерес представляют полиморфизмы, лежащие в пределах кодирующих областей генома. SNP, находящиеся в них, могут быть ассоциированы с определенными фенотипами, в частности, связанные
с индивидуальной предрасположенностью к заболеваниям, с устойчивостью
к воздействию окружающей среды и лекарственных препаратов или с биохимическим профилем. Особенности последовательности четырех нуклеотидов
в нетранскрибируемых регионах ДНК могут отражать различия между отдельными животными, популяциями и породами. SNP более стабильны, чем микросателлиты и имеют более низкую частоту мутации. В 2001 г. были впервые описаны условия генотипирования большого числа особей по любым SNP,
а также методы вычислений, которые в будущем позволили автоматизировать процесс генотипирования (Ranade K., Chang M. S., Ting G. T., Pei D., Hsiao C. F., Olivier M., Pesich R., Hebert J., Chen Y.-D., Dzau V. J., Curb D., Olshen R., Rish N., Cox D. R., Botstein D., 2001). Все вышесказанное объясняет появление огромного числа методов детекции SNP.
1.1.4 Методы детекции SNP
В самом простом случае для детекции SNP можно использовать две пары праймеров. В каждой паре один из праймеров общий для двух аллелей, другой – комплементарный только данному аллелю. Вариабельный нуклеотид располагается в 3′-концевой позиции аллель-специфичных праймеров. ПЦР для каждой пары проводят в отдельных пробирках (Кофиади И. А., Ребриков Д. В., 2006). Основной недостаток подхода в том, что поскольку анализ идет в различных пробирках, необходима четкая стандартизация исходного количества ДНК.
Проведение реакции в одной пробирке исключает ошибки смешивания реактивов и обеспечивает наличие конкуренции на стадии отжига, что в свою очередь ведет к лучшему разрешению аллелей.
Существует несколько подходов проведения аллель-специфичной ПЦР
в одной пробирке, схемы метода проведения которой изображены на рисунках 2–6 (Кофиади И. А., Ребриков Д. В., 2006).
Использование детектирующих амплификаторов для оценки количества продуктов накопления ПЦР имеет неоспоримые преимущества для разработки систем детекции SNP, позволяя упростить подбор условий реакции и, что самое главное, работать практически с любыми стартовыми количествами ДНК. Практически для любого из вышеупомянутых подходов требуется стандартное оборудование для ПЦР и возможность получения меченых нуклеотидов. Однако они являются относительно низкопропускными и позволяют силами одного сотрудника проводить анализ лишь нескольких сотен полиморфизмов в день, что не позволяет их использовать для широкомасштабного скрининга SNP на уровне целого генома (Кофиади И. А., Ребриков Д. В., 2006).
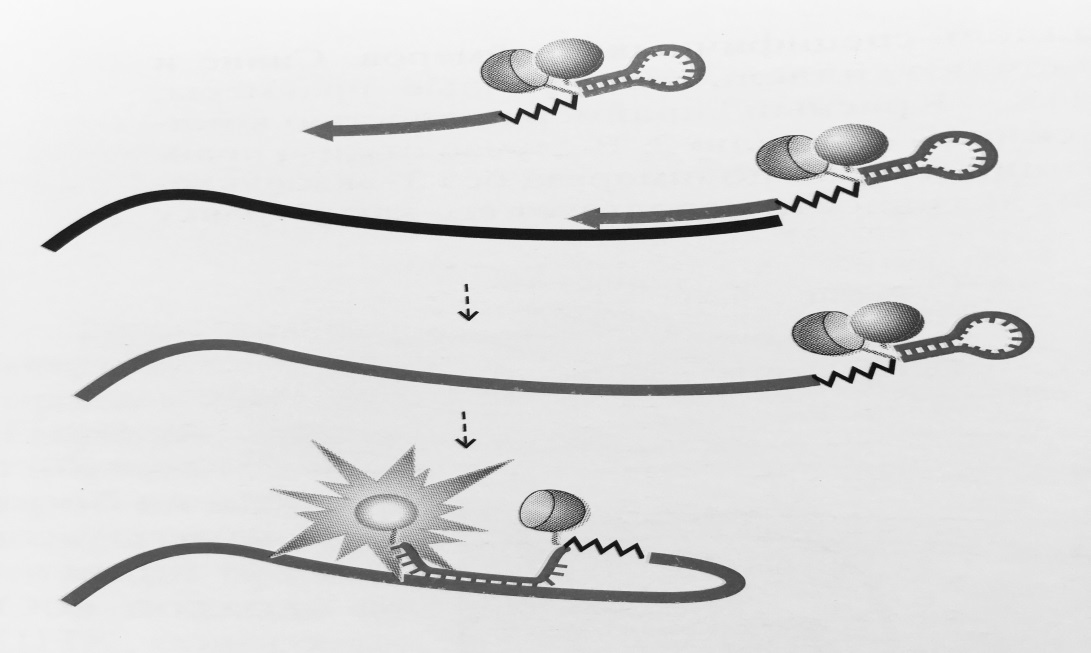
Рисунок 2 – Схема работы праймеров-«скорпионов»:
5′-концевая адаптерная часть праймера отделена от 3′-концевой аллель-специфичной части препятствующим синтезу второй цепи ДНК блокатором (обозначен ломаной линией).
При амплификации адаптер свободного праймера формирует шпилечную структуру со сближенным флуорофором и гасителем. Поскольку петлевая часть адаптера комплементарна внутренней части амплифицируемого фрагмента, после встраивания в ДНК адаптер
гибридизуется с фрагментом, приводя к разобщению флуорофора и гасителя и возрастанию флуоресценции
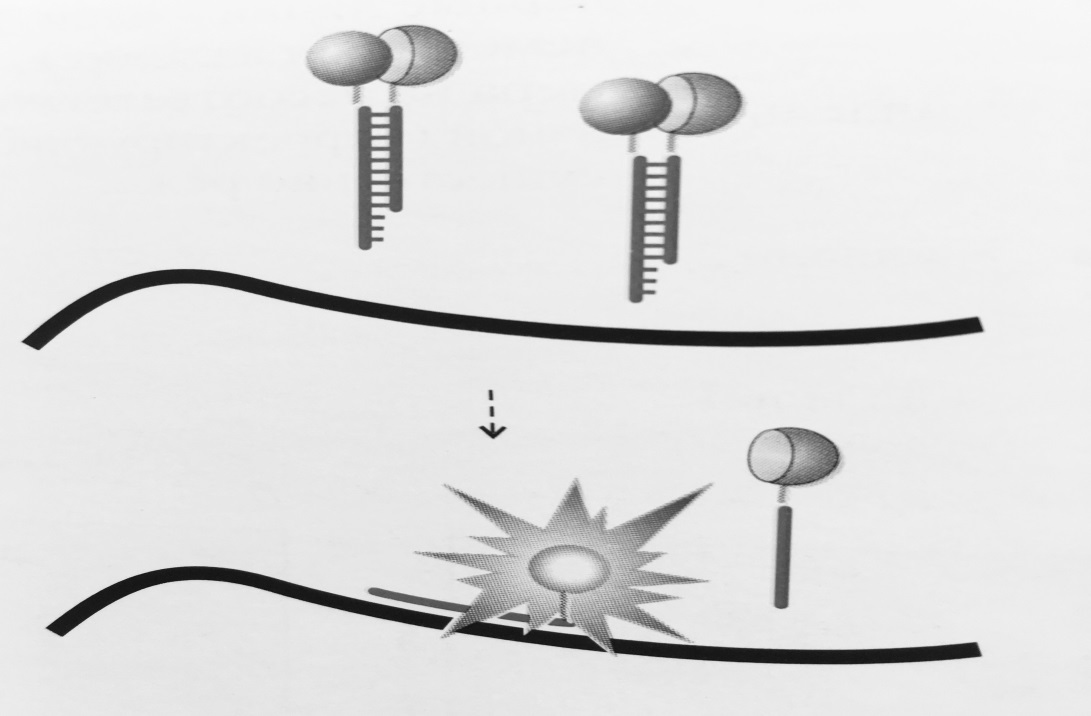
Рисунок 3 – Схема работы «вытесняющей пробы»:
Флуорофор и гаситель флуоресценции расположены на комплементарных нуклеотидах
так, что при образовании дуплекса они оказываются сближенными. В случае образования
специфичного продукта ПЦР часть проб гибридизуется с продуктом реакции,
приводя к увеличению уровня флуоресценции
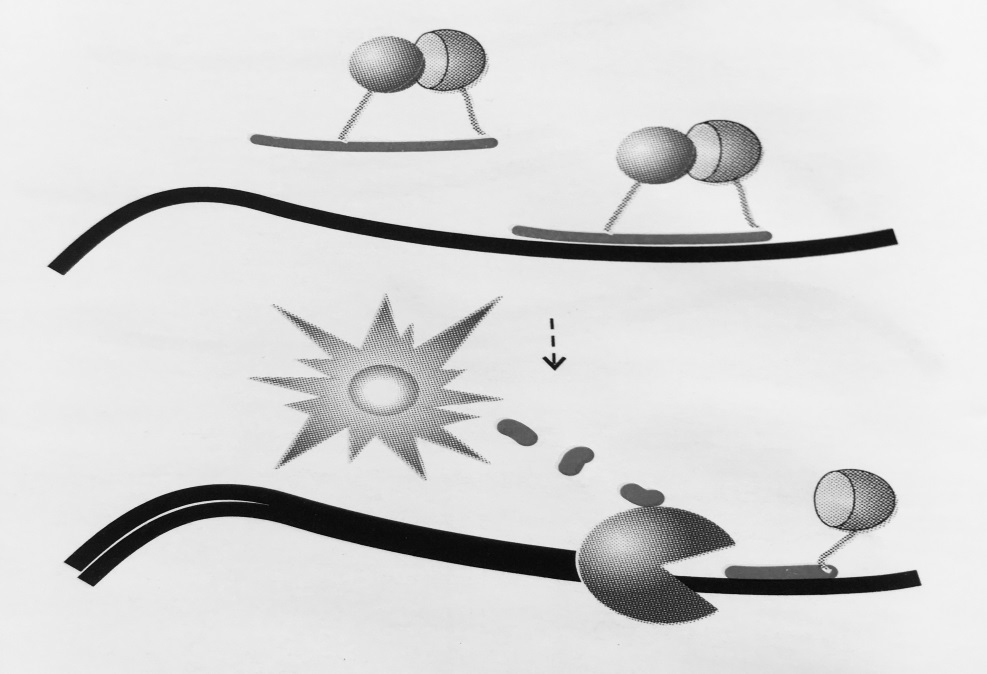
Рисунок 4 – Схема работы «разрушающей пробы»:
Пробы несут флуорофор и гаситель флуоресценции. Пока проба находится в растворе
за счет близкого расположения гаситель поглощает энергию флуорофора. В случае отжига
на специфический продукт реакции проба разрушается за счет 5′- экзонуклеазной активности полимеразы, что ведет к разобщению флуорофора и гасителя. Интенсивность сигнала
возрастает с каждым циклом ПЦР, пропорционально накоплению ампликонов
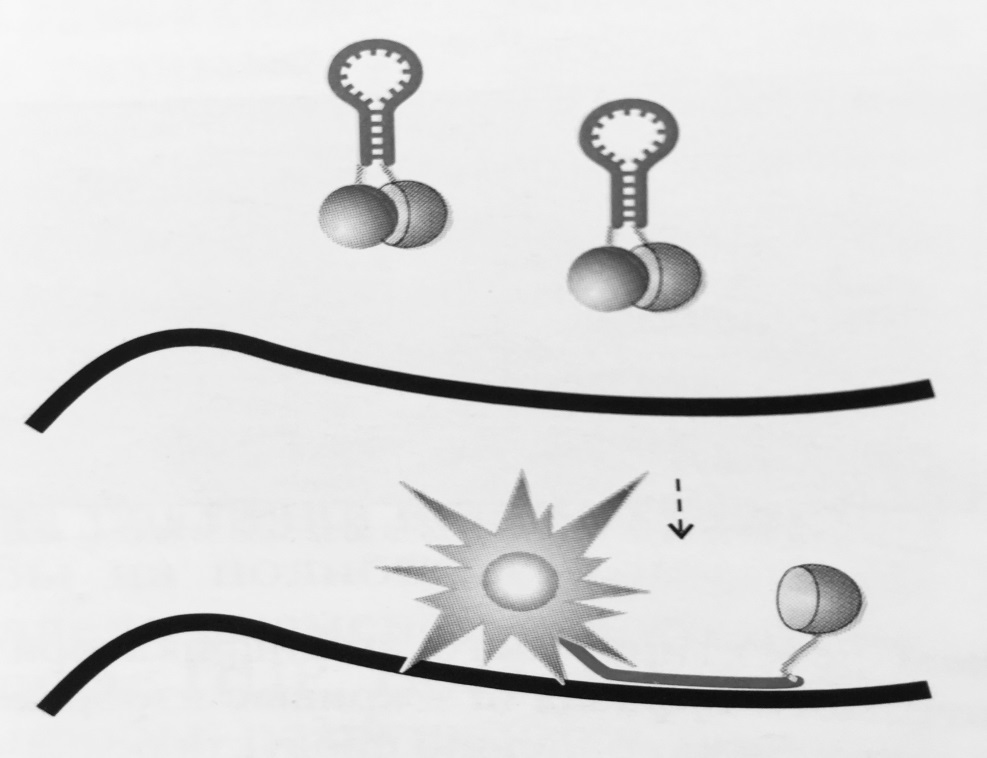
Рисунок 5 – Схема работы «молекулярных маячков»:
Пробы несут короткий инвертированный концевой повтор, по краям которого расположены флуорофор и гаситель флуоресценции. В растворе проба формирует шпилечную структуру,
в которой флуорофор и гаситель сближены. При отжиге на специфический продукт реакции проба «разворачивается», что ведет к разобщению флуорофора и гасителя и повышению уровня флуоресценции

Рисунок 6 – Схема работы «примыкающих проб»:
Система состоит их двух нуклеотидов, гибридизующихся на матрицу рядом. Один из
нуклеотидов несет флуорофор-донор, другой флуорофор-акцептор. Акцептор может
эффективно поглощать энергию, излучаемую донором. При образовании специфичного
продукта реакции олигонуклеотиды гибридизуются рядом, что ведет к эффективному
переносу энергии с донора на акцептор, а накопление продукта реакции амплификации
регистрируют по увеличению флуоресценции акцептора
Платформы для одновременного и высокопроизводительного генотипирования сотен и даже сотен тысяч SNP разработаны несколькими кампаниями.
К 2008 г. затраты на генотипирование были снижены до уровня ниже 0,01 долл. за генотип. Сейчас стоимость такого рода анализа составляет приблизительно 0, 0002 долл. за генотип (Веллер Дж. И., 2018). В настоящее время ведущей технологией высокопроизводительного SNP-генотипирования является «Infinium HD assay» (https://support.illumina.com/downloads/ infinium_hd
_ultra_assay_protocol_guide_11328087_b.html). По этой технологии были разработаны чипы средней плотности «BeadChips», включающие 50–60 тыс. маркеров для всех основных сельскохозяйственных животных. Был разработан чип «BovineHD BeadChip» (Illumina, Сан-Диего, Калифорния), который включает в себя 777 тыс. SNP, охватывающих весь геном КРС. Теперь доступен и чип для курицы, включающий более 580 тыс. маркеров (Веллер Дж. И., 2018). Для других основных сельскохозяйственных видов разрабатываются маркерные чипы высокой плотности, включающие более 500 тыс. маркеров.
1.2 Однонуклеотидные маркеры экономически важных признаков молочного скота
Гены-кандидаты маркеров экономически важных признаков, как правило, отбирают для исследования на основании их потенциальной способности влиять на интересующий фенотип. Критерии отбора включают влияние на физиологические процессы, место в метаболических путях и локализацию уже описанных QTLs. За последнее время было опубликовано много исследований по поиску QTLs с фенотипическими проявлениями: удой (М), выход молочного белка (Р), содержание белка в молоке (%Р), выход молочного жира (F), количество соматических клеток в молоке (SCS) и мастита (MST). Например, в работах (Смарагдов М. Г., 2006; Maiorano A., Lourenco D., Tsuruta S., Ospina A., Stafuzza N., Masuda Y., Filho A., Cyrillo J., Curi R., Silva J., 2018; Mokhber M., Moradi-Shahrbarak M., Sadeghi M., Moradi-Shahrbarak H., Stella A., Nicolzzi E., Rahmaninia J., Williams J. L., 2018) собраны все доступные сведения о локализации QTLs в аутосомах КРС. По данным 869 публикаций в настоящее время
в различных породах и популяциях КРС выявлено 116067 QTLs (https://www.animalgenom.org; обращение 13.11.2018).
Расчеты свидетельствуют, что каждый показатель молока КРС могут контролироваться вплоть до 100 QTLs (Hayes B., Goddard M. E., 2001). Причем
17 % этих локусов – мажорные, т. е. дающие существенный вклад (0,2–0,6)
в фенотипическую вариансу. Остальные являются умеренными и минорными. Большинство мажорных QTLs уже выявлено. 15 важнейших из них перечислены в таблице 3 (https://www.animalgenom.org).
Процедура анализа включает в себя взятие биологического материала (кровь, семя, волосы, выщипы), выделение ДНК, генотипирование на чипах, т. е. идентификация SNP, которые достоверно коррелируют с хозяйственно полезными признаками, и статистический анализ полученных данных (Лукъянов К. И., Солошенко В. А., Клименюк И. И., Юдин Н. С., 2015; Смарагдов М. Г., 2009).
Таблица 3 – Важнейшие экономически важные признаки КРС и количество определяющих их мажорных генетических локусов
| Признак | Число идентифицированных QTLs |
| Возраст полового созревания | 10545 |
| Окружность вымени | 10435 |
| Процентное содержание жира | 6652 |
| Выход жирного молока | 5489 |
| Процентное содержание каппа-казеина | 4784 |
| Процентное содержание нормальной спермы | 3596 |
| Легкость отела | 3540 |
| Процентное содержание молока | 3540 |
| Процентное содержание гликозилированной формы каппа-казеина | 2753 |
| Процентное содержание негликозилированной формы каппа-казеина | 2542 |
| Non return rate | 2419 |
| Продолжительность использования коров | 2233 |
| Выход молока | 2037 |
| Интервал между первой и последней лактацией | 1900 |
| Эффективность осеменения | 1705 |
Для оценки на уровень продуктивности того или иного признака необходимо разработать специальную математическую модель влияния каждого полиморфизма. Разрабатывают такую модель на стандартной или референсной популяции. Референсная популяция – это группа животных одной породы, оцененных по качеству потомства и по данным SNP-генотипирования. Эти животные должны иметь достоверные значения индексной оценки по селекционным признакам. Точность геномных оценок возрастает при увеличении объема референсной выборки (Лукъянов К. И., Солошенко В. А., Клименюк И. И., Юдин Н. С., 2015).
Для отбора QTL с использованием GS-моделей можно использовать несколько математических подходов. Очень подробно они изложены в работах (Веллер Дж. И., 2018; Смарагдов М. Г., 2009; Wiggans G. R., Cole J. B., Hubbard S. M., Sonstegard T. S., 2017). Если коротко, то:
1) каждый маркер – это QTL. Вычисляют одновременно влияние всех маркеров одновременно по их вкладу в генетическую вариансу. Этот подход предполагает, что каждый полиморфизм возникает один раз и существует в коммерческих популяциях животных в масштабе всей популяции. Варианса каждого маркера зависит от неравновесности по сцеплению «LD» (Linkage Disequilibrium), мерой которой служит r2 – квадрат коэффициента корреляции между локусами. LD измеряется между каждой парой локусов.
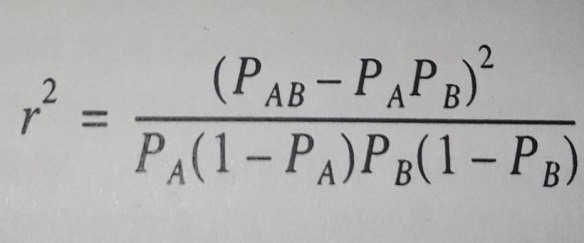
Рисунок 7 – Формула вычисления квадрата корреляции двух аллелей
для расчета LD, где РАВ – частота гаплотипа АВ, РА частота аллеля А,
РВ – аллеля В (Смарагдов М. Г., 2009)
Чем больше расстояние между маркером и QTL, тем меньше r2. При расстоянии между локусами 50 т.п.о. для голштинской породы КРС он составляет 0,35 (Смарагдов М. Г., 2009; McKay S. D., Schnabel R. D., Murdoch B. M., Matukumalli L., Aerts J., Coppieters W., Crews D., Neto E., Gill C., Gao C., Mannen H., Stothard P., Wang Z., Tassell C., Williams J., Taylor J., Moore S., 2007). Этот подход предполагает, что гаплотипы между парами локусов известны точно. Для особей, гетерозиготных для обоих локусов, гаплотипы можно определить однозначно, только если соответствующая родословная генотипирована для обоих локусов. Существует большое количество литературы для определения гаплотипов более сложных родословных, например (Веллер Дж. И., 2018; Weng Z., Zhang Zh., Zhang Q., He S., Ding X., 2013);
2) SNP конструируется из нескольких гаплотипов. В этом случае увеличивается LD между гаплотипом и QTL и, следовательно, величина вариансы QTL. При этом уменьшается точность определения каждого гаплотипа;
3) вместилищем всех QTL является гамета, эффекты которой в будущем определяют через маркеры. Анализ сцепления аллелей с QTLs прослеживают через родословные животных с помощью расчета вероятностей при условии, что любые два маркера идентичны по происхождению IBD (Identity By Descent) от общего предка в родословной.
Учитывая выгоды, получаемые от популяции внутри страны, программы разведения КРС на основе GS отличаются от большинства других экономических проектов тремя аспектами (Веллер Дж. И., 2018):
1. В связи с биологическими ограничениями, особенно относительно длительным интервалом между поколениями, программу разведения можно оценить только после длительного периода времени.
2. Генетические маркеры, как правило, одновременно определяют множество признаков.
3. В отличие от других агротехнических достижений, генетические тренды являются устойчивыми. Они «не изнашиваются», их техническое обслуживание не требует дополнительных инвестиций.
Основополагающие работы по результатам геномной селекции были осуществлены Мовиссеном с соавторами, в которых были проведены оценки распределения QTLs от эффектов маркеров и показана множественная их зависимость от используемых математических моделей (Meuwissen T. H., Hayes B. J., Goddart M. E., 2001; Meuwissen T. H., Karlsen A., Lien S., Olsaker I., Goddard M. E., 2002; Meuwissen T. H., Goddard M. E., 2004). Кроме того, коммерческие популяции КРС находятся между экстремальными состояниями, поскольку снижение генетического разнообразия всегда компенсируется увеличением количества мутаций, т. е. увеличением полиморфизма ДНК.
Все предыдущие разделы касались методов определения фактических полиморфизмов, ответственных за наблюдаемую генетическую вариантность
в количественных экономически важных признаках КРС. Они включены в программы отбора в качестве фиксированных эффектов. С появлением технологии ZFN, TALEN и CRISPR/Cas9 фактически стало возможным изменять тип эмбрионов. Эти технологии теперь позволяют модифицировать эпигеном целого локуса для изменения регуляторных путей, что, в конечном счете, приведет
к значительным изменениям фенотипа животного. Однако в настоящее время применение этих технологий для сельскохозяйственных животных вызывает большие этические возражения.
1.3 Выбор платформы для реализации исследования.Описание панели TruSeq Bovine Parentage Sequencing Panel (Illumina, США)
Идентификация животных играет ключевую роль в государственной сельскохозяйственной политике, позволяя управлять выплатами субсидий, перемещением скота, составлением графиков испытаний и борьбы с болезнями. Достижения в области геномики крупного рогатого скота позволили использовать врожденную генетическую изменчивость для уникальной идентификации отдельных животных с помощью профилирования ДНК, во многом благодаря тому, что было получено в области генотипирования человека за последние
20 лет. Тест на ДНК-профилирование, основанный на маркерах биаллельных однонуклеотидных полиморфизмах (SNP), обеспечивает значительные преимущества по сравнению с текущими отраслевыми тестами на основе стандартных коротких тандемных повторов (STR), его легче анализировать и интерпретировать.
Выявление и регистрация скота и контроль его перемещения является важной частью сельскохозяйственной политики национальных правительств. Такие схемы лежат в основе борьбы с болезнями, управления грантами и субсидиями, обеспечения гигиены пищевых продуктов и безопасности, все это облегчает отзыв продукции при необходимости.
В результате этих преимуществ, а также в ответ на основные проблемы со здоровьем животных, такие как BSE (губчатая энцефалопатия) и ящур, которые влияют на домашний скот, производителей и потребителей, во многих странах развитого мира приняли национальные базы данных, основанные на пронумерованных ушных бирках, для регистрации личности и перемещений крупного рогатого скота (Buick W., 2004; Houston R., 2001).
Глобализация торговли также усилила аргументы в пользу улучшения отслеживания животных. Современные потребители, сталкивающиеся с расширением выбора из нескольких источников, могут также столкнуться с повышенными рисками в результате повышенной вероятности химического (атогенного) загрязнения пищевых продуктов (Caporale V., Giovannini A., Di Francesco C., Calistri P., 2001; McKean J. D., 2001). Такие кризисы могут поставить под угрозу экономическое благополучие агропродовольственных отраслей, а также повлиять на здоровье и доверие потребителей. Кризис БФБ 1990-х гг. наглядно продемонстрировал это, и показал, что действия одного государства могут негативно повлиять на общественное здоровье других (McGrann J., Wiseman H., 2001).
В свете потенциальных опасностей, о которых говорилось выше, Европейский союз (ЕС) принял Директиву Совета 92/102/EEC, в которой говорится, что все коровы в государствах-членах должны быть идентифицированы с помощью ушной бирки с уникальным идентификационным кодом (Ammendrup S., Fussel A. E., 2001). Более расширенные правила Совета 1760/2000 (Regulation (EC) № 1760/2000, 2000) и ст. 18 правила 178/2002 (Council Regulation (EC) № 178/2002, 2002) устанавливают необходимость для государств-членов контролировать регистрацию и отслеживание крупного рогатого скота через компьютеризированную базу данных, основанную на системе с двумя ушными бирками. Эти расширенные правила отслеживаемости также распространяются на продукты, полученные от животного после убоя, гарантируя возможность отследить партию мяса вплоть до происхождения животного с помощью маркировки на тушах, отрубах и упаковке. Эти правила были разработаны для обеспечения того, чтобы полная прослеживаемость животных и мяса была «установлена на всех этапах производства, обработки и распределения».
Дальнейшая разработка схем меток может включать использование электронных идентификационных устройств, таких как метки радиочастотной идентификации (RFID), рубцовые болюсы и инъекционные транспондеры, которые автоматизируют считывание идентификационных данных животных, тем самым уменьшая ошибки перевода (Ribo O., Korn C., Meloni U., Cropper M., De Winne P., Cuypers M., 2001).
Недавний отчет ЕС об использовании электронной идентификации в агропродовольственной промышленности (проект IDEA – IDentification Electronique des Animaux; http://idea.jrc.it/), хотя и приветствовал способность таких систем улучшать управление сельским хозяйством, тем не менее подтвердил, что предпочтительный вариант – ушные бирки RFID – все еще страдает от той же случайной потери меток и проблем с мошеннической перестановкой, которыми страдают традиционные схемы (Report From The Commission To The Council And The European Parliament, 2005).
Неправильная идентификация животных в результате потери метки имеет серьезные эпидемиологические последствия и проблемы с трекингом, которые могут привести к дорогостоящим последствиям для производителей. Степень потерь метки и отрицательное влияние этого на способность восстановить правильный идентификатор были определены в недавнем исследовании на активно размножаемых буйволах, которое показало, что среднее время сохранения ушной метки составляет 272 дня (Fosgate G. T., Adesiyun A. A., Hird D. W., 2006). Аналогичным образом, недостатки в маркировке мяса на скотобойнях и в торговых точках приводят к потере коррекции между номерами партий и образцов. В недавнем исследовании с использованием типирования ДНК было показано, что 2 % случайно выбранных образцов из меченых тушек в скотобойне не соответствовали профилям животных, от которых они якобы исходили. Этот показатель увеличивался до 3 %, когда отбор проб проводился в торговой точке (Capoferri R., Bongioni G., Galli A., Aleandri R., 2006).
Корень этих проблем в обычных и электронных системах мечения/маркировки возникает из-за их зависимости от методов, с помощью которых отслеживают устройства, прикрепленные к животным и их продуктам, но не самих животных или продуктов. Тем не менее, профилирование ДНК, которое использует неизменные биологические свойства отдельных животных для получения уникального идентификатора, предлагает потенциальное решение для научной проверки идентичности животных (Pettitt R. G., 2001). В настоящее время не существует технологии, позволяющей считывать профили ДНК в режиме реального времени, в отличие от меток и ярлыков. Это ограничивает использование профилирования по ДНК в качестве основного идентификатора животных и производных пищевых продуктов. Тем не менее, несмотря на эти ограничения, профилирование по ДНК можно эффективно использовать в ретроспективных аудитах для проверки идентичности и качества меток для обеспечения существующих технологий отслеживания мяса (Cunningham E. P., Meghen C. M. , 2001; Pettitt R. G., 2001; Raspor P., 2004). Дополнительное преимущество проверки происхождения на основе ДНК делает ее мощным методом проверки информации из базы данных по национальным стадам.
1.3.1 ДНК-маркеры и ДНК-профилирование
Уровень полиморфной изменчивости, наблюдаемый для бычьих коротких тандемных повторов (STR), позволяет использовать относительно немного локусов для идентификации крупного рогатого скота и определения его происхождения (Glowatzki-Mullis M. L., Gaillard C., Wigger G., Fries R., 1995; Heyen D. W., Beever J. E., Da Y., Evert R. E., Green C., Bates S. R., Ziegle J. S., Lewin H. A., 1997).
Несколько хорошо определенных панелей STR для крупного рогатого скота имеются в продаже. Одна такая 11-маркерная панель была одобрена Международным обществом генетики животных (ISAG) для использования племенными сообществами для регистрации племенных быков, телок и их потомства.
Одиночные нуклеотидные полиморфизмы (SNP) представляют собой гораздо более простую форму вариации ДНК, чем микросателлиты, включая изменение одного нуклеотида в одной позиции генетического кода. Это отсутствие разнообразия делает маркеры SNP менее информативными, чем микросателлиты.
В результате, одиночные SNP нельзя использовать для профилирования индивидуумов. Тем не менее, генотипирование нескольких SNP может решить эту проблему (Werner F. A. O., Durstewitz G., Habermann F. A., Thaller G., Kramer W., Kollers S., Buitkamp J., Georges M., Brem G., Mosner J., Fries R., 2004).
Генотипирование с использованием SNP также имеет явные преимущества перед генотипированием с помощью STR. Из-за своего меньшего размера они менее подвержены мутациям в гаметах, и поэтому не имеют проблемы потери идентичности при смене поколений, которые могут влиять на анализ с использованием STR, поскольку они мутируют между поколениями (Werner F. A. O., Durstewitz G., Habermann F.A., Thaller G., Kramer W., Kollers S., Buitkamp J., Georges M., Brem G., Mosner J., Fries R., 2004). SNP также являются более надежными при их интерпретации/анализе по сравнению с STR (Krawczak M., 1999), и они могут быть генотипированы с помощью различных методов с высокой пропускной способностью на различных платформах, многие из которых могут быть автоматизированы с экономией при масштабировании (Kruglyak L., 1997). С появлением постоянно улучшающихся технологий генотипирования (Kim S., Misra A., 2007) в последнее время стало привлекательным создание системы профилирования ДНК на основе SNP для идентификации крупного рогатого скота. Для этой цели недавно были собраны две панели SNP (Heaton M. P., Harhay G. P., Bennett G. L., Stone R. T., Grosse W. M., Casas E., Keele J. W., Smith T. P. L., Chitkon-McKown C. G., Laegreid W. W., 2002; Werner F. A. O., Durstewitz G., Habermann F.A., Thaller G., Kramer W., Kollers S., Buitkamp J., Georges M., Brem G., Mosner J., Fries R., 2004).
TruSeq Bovine Parentage Sequencing Panel – это ценное решение для тестирования родства для нескольких пород мясного и молочного скота. В данном анализе предлагается больше информации в одном анализе, чем в одном только тесте родства, благодаря чему сервисные лаборатории могут использовать панель секвенирования TruSeq Bovine для получения более информированных рекомендаций по управлению стадом.
1.3.2 Преимущества
Расширенное содержание повышает ценность – панель включает в себя все рекомендуемые материалы от Международного общества генетики животных (ISAG), а также связанные с заболеваниями и экономически важные признаки.
Надежный анализ лимитирует затраты – интеллектуальный дизайн контента, высокая точность и надежность исключают необходимость в дорогостоящем многократном или повторном тестировании.
Интуитивно понятное программное обеспечение упрощает анализ интерпретации данных – программное обеспечение автоматически преобразует результаты секвенирования в генетический профиль без необходимости экспертизы в области биоинформатики.
Хотя микросателлиты или короткие тандемные повторы (STR) являлись предпочтительными генетическими маркерами для контроля происхождения
у крупного рогатого скота, мировая индустрия переходит к методам на основе однонуклеотидных полиморфизмов – SNP (ISAG cattle core and additional SNP panel 2013). SNP обладают рядом преимуществ (Allen A. R. [et al.], 2010; Fernández M. E. [et al.], 2013; Schütz E., Brenig B., 2015):
- низкая частота мутаций между поколениями;
- надежная интерпретация данных;
- совместимость с автоматизированным, высокопроизводительным
анализом; - подходит для стандартизированного представления результатов генотипирования.
В панели TruSeq Bovine Parentage используется проверенная технология секвенирования следующего поколения Illumina (NGS), обеспечивающая точные результаты генотипирования для определения происхождения на основе SNP. Оптимизированный рабочий процесс, включая подготовку библиотеки посредством анализа данных, повышает эффективность. Высокая точность
и надежность уменьшают необходимость повторного тестирования, помогая лимитировать расходы. В панели TruSeq Bovine объединены SNP для определения происхождения и добавлено генотипирование биологически значимых признаков в едином тесте.
Разработанная учеными Illumina в соответствии с рекомендациями ISAG, панель секвенирования коров TruSeq Bovine содержит 263 гена. Они охватывают весь рекомендуемый ISAG контент и дополнительные экономически ценные и связанные с заболеванием маркеры (таблица 4).
Таблица 4 – Содержимое панели секвенирования TruSeq Bovine
| Описание | Число SNP |
| SNP, используемые для оценки происхождения | |
| Основная панель SNP для оценки происхождения согласно ISAG (ISAG Core SNP Targets) |
100 |
| Дополнительная панель SNP для оценки происхождения согласно ISAG (ISAG Additional SNP Targets) | 100 |
| Продолжение таблицы 4 | |
| 1 | 2 |
| Вариантные мишени, ассоциированные с генетическими нарушениями/заболеваниями |
|
| Пренатальные/перинатальные смертельные заболевания (HH1, HH3, HH4, MH1 и т. д.) | 12 |
| Постнатальные смертельные заболевания (цитруллинемия, врожденная мышечная дистония, кардиомиопатия, миоклонус и т. д.) |
17 |
| Нелетальные патологии, ухудшающие состояние (синдром Марфана, сращение пальцев, протопорфирия, гемофилия A и т. д.) |
19 |
| Вариантные мишени, ассоциированные с экономически ценными признаками |
|
| Мужская фертильность | 1 |
| Высота в холке и внешний вид | 4 |
| Качество мяса | 4 |
| Качество молока | 6 |
| Общее количество SNP/вариантных мишеней | 263 |
1.3.3 Панель SNP, используемая для оценки происхождения
Международное общество генетики животных (ISAG) создало набор однонуклеотидных полиморфизмов (SNP) для тестирования происхождения (родства) крупного рогатого скота – Bos taurus, который следует использовать на международном уровне, чтобы сделать результаты сопоставимыми между лабораториями. Панель состоит из 100 основных SNP и дополнительного набора из 100 маркеров (CMMPT 2012). SNP в основной панели были отобраны преимущественно на основе данных по европейским тауринным породам и, таким образом, могут не быть идеальными для установления родства в родословно родственных породах (Lachance J., Tishkoff S. A. , 2013; Strucken E., Gudex B., Ferdosi M., Lee H., Song K., Gibson J, Kelly M., Piper E., Porto-Neto L., Lee S. H., Gondro C., 2014). Для решения этой проблемы
в ISAG была предложена дополнительная маркерная панель для увеличения мощности исключения в индиковых и синтетических породах, и эти породы были добавлены в сравнительные тесты общества (http://www.isag.us/
comptest.asp?autotry=true&ULnotkn=true).
1.3.4 SNP, ассоциированные с генетическими нарушениями/заболеваниями
Пренатальная/перинатальная смертность
Аборты, происходящие во время поздней беременности, приводят к значительным экономическим потерям. Оценка стоимости аборта для производителя варьировалась от 90 до 1900 долл. США (De Vries A., 2006; Hovingh E., 2009; Peter A. T., 2000) в зависимости от времени аборта, различий в прогнозируемой производительности коров, а также решениях о разведении и замене. Для голштинской породы известно несколько рецессивных патологических аномалий, связанных с гибелью и абортом плода, и отдельных локусов, которые снижают жизнеспособность эмбрионов или раннее развитие плода у крупного рогатого скота. Одной из распространенных генетических аномалий, вызывающих аборт у коров голштинской породы, является сложный порок позвоночника – CVM (Agerholm J. S., Bendixen C., Andersen O., Arnbjerg J., 2001). CVM вызывает пороки развития во время средней и поздней беременности, хотя некоторые беременности достигают срока и приводят к мертворождению.
Недавно генотипирование сотен тысяч животных в популярных молочных породах США с использованием панелей однонуклеотидного полиморфизма (SNP) по всему геному дало возможность найти области генома с несбалансированным H-W равновесием. Отсутствие ожидаемых гомозиготных гаплотипов в геномной области может быть связано с пагубными последствиями для развития плода и может вызвать аборт и мертворождение у голштинского крупного рогатого скота (Glusman G., Cox H. C., Roach J. C., 2014; VanRaden P. M., Olson K. M., Null D. J., Hutchison J. L., 2011)]. В 2011 г. три летальных гаплотипа, влияющих на развитие плода у крупного рогатого скота голштинской породы, были идентифицированы и локализованы на бычьих хромосомах 5 (HH1),
1 (HH2) и 8 (HH3) (VanRaden P. M., Olson K. M., Null D. J., Hutchison J. L., 2011). Два года спустя во всем мире было зарегистрировано 14 дополнительных летальных гаплотипов (HH4–HH17) у крупного рогатого скота голштинской породы, и тем временем некоторые из них были разрешены на молекулярном уровне, то есть HH1 (APAF1) и HH3 (SMC2), HH4 (GART) и HH5 (TFB1M).
Большинство рецессивных генов, связанных со снижением фертильности
и потерями беременности у крупного рогатого скота, весьма трудно обнаружить, и они передаются без обнаружения, поэтому они не включены в список факторов, ведущих к абортам. HH1 приводит к абортам в течение всего периода беременности, но HH3 приводит к потере эмбриона вскоре после зачатия (VanRaden P. M., O’Connell J. R., Wiggans G. R., Weigel K. A., 2011). В случае
с HH1, потеря фертильности на 34, 51 и 63 % связана с этим гаплотипом и происходит через 60, 100 и 140 дней после оплодотворения, соответственно. Для HH2 – 56 % связаны с этим гаплотипом и происходят через 60 дней после оплодотворения и 94 % – через 100 дней. Для HH3 потерей фертильности на
93 % связана с этим гаплотипом и происходят через 60 дней после оплодотворения (Norman H. D., Miller R. H., Wright J. R., Hutchison J. L., Olson K. M., 2012).
HH1 был картирован в бычьей хромосоме 5 (BTA5), на 58–66Mb (сборка генома UMD 3.0). Используя секвенирование всего генома быка Arlinda Chief из фермы Pawnee Farm (родился в 1962 г. и был одним из самых плодовитых быков в истории разведения голштинов) и трех его сыновей, Адамс с соавторами в 2016 г. идентифицировали причинную нонсенс-мутацию (p.Q579X) в факторе 1, активирующем апоптотическую протеазу (APAF1) в HH1 (Adams H. A., Sonstegard T. S., VanRaden P. M., Null D. J., Van Tassell C. P., Larkin D. M., Lewin H. A., 2016). Предполагается, что эта мутация отрезает 670 аминокислот (53,7 %) в белке APAF1, которые включают домен WD40 – критический для межбелковых взаимодействий. Белок, кодируемый APAF1, является центральным элементом апоптотического каскада, опосредованного цитохромом С (Apweiler R., Bairoch A., Wu C. H., Barker W. C., Boeckmann B., Ferro S., Gasteiger E., Huang H., Lopez R., Magrane M., Martin M. J., Natale D. A., O’Donovan C., Redaschi N., Yeh L. S., 2004), и напрямую связан с нарушениями развития и нейродегенеративными заболеваниями (Blake J. A., Bult C. J., Kadin J. A., Richardson J. E., Eppig J. T., 2011; Honarpour N., Gilbert S. L., Lahn B. T., Wang X., Herz J., 2001). Экспрессия APAF1 во время мышиного развития начинается на ранних стадиях в различных эмбриональных тканях и имеет важное значение для развития центральной нервной системы. Гомозиготный по нокаутному аллелю гена APAF1 у мышей приводит к эмбриональной летальности
к 16,5-му дню, а у APAF1-дефицитных мышей обнаруживается чрезмерный рост головного мозга и черепно-лицевые пороки развития (Honarpour N., Gilbert S. L., Lahn B. ., Wang X., Herz J., 2001; Müller M., Berger J., Gersdorff N., Cecconi F., Herken R., Quondamatteo F., 2005).
Гомозиготность по этому аллелю приводит к самопроизвольному аборту
и, следовательно, к снижению фертильности у быков-носителей, если они спариваются с коровами-носителями, потому что функциональный пептид APAF1 необходим для развития эмбрионов. По оценкам, спонтанные аборты, вызванные этой мутацией во всем мире, за последние 35 лет составили 525000, что составляет около 420 млн долл. убытков. После генотипирования 246773 голштинов было обнаружено, что 5299 коров являлись гетерозиготами по APAF1, и не было обнаружено гомозиготных коров по этой мутации, поэтому частота носителей мутации APAF1 составила 2,1 % (Adams H. A., Sonstegard T. S., VanRaden P. M., Null D. J., Van Tassell C. P., Larkin D. M., Lewin H. A., 2016).
HH2. В 2014 г. МакКлюр с соавторами уточнили картирование гаплотипа HH2 на BTA1 с 94860836 до 96553339 (UMD3.1) (McClure M. C., Bickhart D., Null D., Vanraden P., Xu L., Wiggans G., Liu G., Schroeder S., Glasscock J., Armstrong J., Cole J. B., Van Tassell C. P., Sonstegard T. S., 2014). В обширном исследовании, включающем захват экзома и секвенирование следующего поколения, МакКлюр с соавторами не смогли обнаружить какой-либо потенциально причинный вариант для гаплотипа HH2. Эффекты фертильности для HH2 были в значительной степени подтверждены путем сравнения нормальных показателей зачатия для голштинов (31 %) с частотой спаривания гетерозиготных быков с дочерями гетерозиготных быков (VanRaden P. M., Olson K. M., Null D. J., Hutchison J. L., 2011). Показатели зачатия носителей HH2 были ниже среднего. Большинство потерь эмбрионов для HH2 происходило до 100-го дня беременности (VanRaden P. M., Olson K. M., Null D. J., Hutchison J. L., 2011). Частота мертворождения для HH2 была невысока, но все-таки незначительно выше обычной. Бык-основатель гаплотипа HH2 был прослежен по происхождению до отца по имени Willowholme Mark Anthony (McClure M. C., Bickhart D., Null D., Vanraden P., Xu L., Wiggans G., Liu G., Schroeder S., Glasscock J., Armstrong J., Cole J. B., Van Tassell C. P., Sonstegard T. S., 2014).
HH3. В своей статье от 2014 г. МакКлюр с соавторами также уточнили картирование гаплотипа HH3 на BTA8 с 95,057,877 до 95,468,310 (UMD3.1) (McClure M. C., Bickhart D., Null D., Vanraden P., Xu L., Wiggans G., Liu G., Schroeder S., Glasscock J., Armstrong J., Cole J. B., Van Tassell C. P., Sonstegard T. S., 2014). Кроме того, в своей статье Детуайлер с совторами заявили, что в проекте, включающем 1000 бычьих геномов, в качестве носителя гаплотипа HH3 был идентифицирован один бык (Daetwyler H. D., Capitan A., Pausch H., Stothard P., van Binsbergen R., Brøndum R. F., Liao X., Djari A., Rodriguez S. C., Grohs C., Esquerré D., Bouchez O., Rossignol M. N., Klopp C., Rocha D., Fritz S., Eggen A., Bowman P. J., Coote D., Chamberlain A. J., Anderson C., Van Tassell C. P., Hulsegge I., Goddard M. E., Guldbrandtsen B., Lund M. S., Veerkamp R. F., Boichard D. A., Fries R., Hayes B. J., 2014). С использованием методик экзомного захвата и секвенирования следующего поколения, МакКлюр с соавторами установили, что данная мутация представляла SNP-замену (T/C) в 24 экзоне гена структурной сохранности хромосом 2 (SMC2). Для подтверждения этого, мутация была генотипирована путем секвенирования панели геномов 10 известных носителей HH3; все они были гетерозиготными по мутации, подтверждая связь между областью HH3 и аллелем g.95410507C. Из-за жизненно важной роли SMC2 в репарации ДНК и конденсации хромосом во время клеточного деления, эта мутация вызывает ненейтральную и непереносимую замену в НТФ-азном домене кодируемого белка из-за смены аминокислоты с фенилаланина на серин.
Кроме того, 5606 голштинских особей были генотипированы по мутации T>C с использованием стандартизированного метода Illumina BeadChip, и не было выявлено ни одного индивида с генотипом CC, что подтверждает гипотезу, что эта мутация вызывает эмбриональную летальность. Более того, частота зачатия была ниже, чем частота зачатия у здоровых голштинов (31 %), при спаривании гетерозиготных быков HH3 с дочерями гетерозиготных быков. Большинство потерь зачатия для HH3 происходило до 60 дней (VanRaden P. M., Olson K. M., Null D. J., Hutchison J. L., 2011). Бык-основатель для HH3 был прослежен до производителей по имени Glendell Arlinda Chief и Gray View Skyliner (McClure M. C., Bickhart D., Null D., Vanraden P., Xu L., Wiggans G., Liu G., Schroeder S., Glasscock J., Armstrong J., Cole J. B., Van Tassell C. P., Sonstegard T.S., 2014).
HH4. Фритц с соавторами в 2013 г. локализовали HH4 на BTA1 в области 1,9–3,3 Мб (сборка генома UMD 3.1) и предоставили значительные доказательства возможной причинной мутации в гене GART, который кодирует глицинамид рибонуклеотидтрансформазу, что привело к p.N290T (Fritz S., Capitan A., Djari A., Rodriguez S. C., Barbat A., Baur A., Grohs C., Weiss B., Boussaha M., Esquerré D., Klopp C., Rocha D., Boichard D., 2013). Было предсказано, что две мутации, связанные с HH4, приводят к патологии: трансверсия А-С в положении g.1277227A.C и переход G-A в положении g.2490314G.A на BTA1. Первая из них приводит к замене аспарагина в предполагаемом сайте связывания марганца треонином (p.N290T) в белке GART. Аспарагин-290 полностью консервативен среди эукариот, что доказывает ключевую роль этой аминокислоты
в функционировании GART. Кроме того, GART жизненно важен для биосинтеза пуринов de novo, которые являются важными элементами ДНК и РНК. Поэтому, ожидается, что потеря функции GART приведет к гибели эмбриона на ранних стадиях эмбриональной жизни. Эти данные убедительно подтверждают потенциальную роль мутации в GART (p.N290T) в снижении фертильности, наблюдаемой при спаривании между носителями HH4 и дочерями носителей HH4 (Fritz S., Capitan A., Djari A., Rodriguez S. C., Barbat A., Baur A., Grohs C., Weiss B., Boussaha M., Esquerré D., Klopp C., Rocha D., Boichard D., 2013). Вторая мутация – g.2490314G.A – приводит к замене цистеина на тирозин в остатке 13 гомолога A белка кинетохоры MIS18 (S. pombe) (MIS18A p.C13Y) (Fritz S., Capitan A., Djari A., Rodriguez S. C., Barbat A., Baur A., Grohs C., Weiss B., Boussaha M., Esquerré D., Klopp C., Rocha D., Boichard D., 2013). Хотя гомозиготный по нокаутному аллелю MIS18A является летальным на стадии эмбриона у мышей (Kim I. S., Lee M., Park K. C., Jeon Y., Park J. H., Hwang E. J., Jeon T. I., Ko S., Lee H., Baek S. H., Kim K. I., 2012), и хотя эта мутация, как было предсказано, является вредной в соответствии с SIFT и Polyphen для функции белка человека, менее вероятно, что эта мутация отвечает за данный фенотип, поскольку остаток 13 и соседняя аминокислотная последовательность имеют слабую консервативность у плацентарных млекопитающих (Fritz S., Capitan A., Djari A., Rodriguez S. C., Barbat A., Baur A., Grohs C., Weiss B., Boussaha M., Esquerré D., Klopp C., Rocha D., Boichard D., 2013).
Генотипирование 72 производителей-быков, представляющих все гаплотипы с частотой выше 1 %, выявило, что мутация g.1277227A.C была связана только с гаплотипом HH4, тогда как мутация g.2490314G.A могла быть связана с другими гаплотипами. Последующее генотипирование четырех животных, которые были составными гетерозиготами по гаплотипам, связанным
с g.2490314G.A, подтвердило их гомозиготность и определенно исключило эту кандидатурную мутацию.
HH5. С использованием стратегии, аналогичной стратегии ван Радена
и его соавторов (VanRaden P. M., O’Connell J. R., Wiggans G. R., Weigel K. A., 2011), Купером с соавторами был выделен гаплотип HH5, связанный со сниженной рождаемостью, который находится на 92350052–93910957 п.н. на BTA9 (Cole J. B., VanRaden P. M., Null D. J., Hutchison J. L., Cooper T. A., Hubbard S. M., 2015; Cooper T. A., Wiggans G. R., Null D. J., Hutchison J. L., 2013). Шульц с соавторами определили вероятную причину этого гаплотипа – мутацию в виде делеции 138kb, охватывающей от 93233 до 93371 kb на BTA9, содержащую только диметиладенозинтрансферазу 1 (TFB1M) (Schütz E., Wehrhahn C., Wanjek M., Bortfeld R., Wemheuer W. E., Beck J., Brenig B., 2016). Края делеции фланкированы бычьими длинными вкрапленными ядерными элементами Bov-B (выше делеции) и L1ME3 (ниже делеции), что позволяет предположить, что гомологичная рекомбинация ответственна за это событие. TFB1M необходима для синтеза и функционирования малой рибосомальной субъединицы митохондрий. Связь этой делеции с данным гаплотипом подтверждается тем фактом, что гомозиготные мыши, нокаутированные по TFB1M, нежизнеспособны
и гибнут на стадии эмбриона. Летальность мышей HH5 – TFB1M-/- демонстрирует эмбриональную гибель на 8,5 день (Metodiev M. D., Lesko N., Park C. B., Camara Y., Shi Y., Wibom R., Hultenby K., Gustafsson C. M., Larsson N. G., 2009).
Кроме того, клеточный нокаут подтвердил серьезное летальное воздействие на клетки млекопитающих вследствие полной потери трансляции белка
в митохондриях (Metodiev M. D., Lesko N., Park C. B., Camara Y., Shi Y., Wibom R., Hultenby K., Gustafsson C. M., Larsson N. G., 2009). Шульц с соавторами проверили имеет ли делеция TFB1M ко-доминантный эффект у крупного рогатого скота-носителя in vivo, для этого соотношение митохондриальной ДНК
к ядерной ДНК было определено с помощью ddPCR в ДНК, выделенной из лейкоцитов у случайным образом выбранных представителей крупного рогатого скота. По данному параметру не наблюдалось существенной разницы между носителями и диким типом, что предполагает рецессивный эффект, который согласуется с фенотипом животных-носителей, у которых не наблюдается каких-либо заметных отклонений. Учитывая тот факт, что никакой другой ген не находится в делетированном участке, вместе с вышеупомянутыми данными
и высокой сегрегацией по данным HH5, эти результаты могут служить доказательством того, что эта делеция является причиной HH5 (Schütz E., Wehrhahn C., Wanjek M., Bortfeld R., Wemheuer W. E., Beck J., Brenig B., 2016). Бык-основатель HH5 восходит к Thornlea Texal Supreme, родившемуся в 1957 г. Частота гетерозиготных носителей составляет примерно 4–5 % в европейской
и североамериканской фризской популяции голштинов, и приводит к преждевременному прерыванию беременности до 60-го дня беременности (Schütz E., Wehrhahn C., Wanjek M., Bortfeld R., Wemheuer W. E., Beck J., Brenig B., 2016).
Частое использование определенных производителей и их потомков в современном скотоводстве несет риск появления генетических нарушений в популяции в течение короткого периода времени. Было показано, что после выявления причинной мутации генотипирование очень эффективно устраняет патологических носителей в популяции. За последнее время с использованием большого массива данных по SNP у коров голштинской породы были обнаружены несколько гаплотипов, связанных с летальными фенотипами, и для некоторых из них были определены гены, вызывающие данные гаплотипы. Одной из эффективных стратегий в случае определенных рецессивных мутаций является обнаружение носителей с целью их исключения из размножающейся популяции или применении различных стратегий спаривания для контроля распространения нежелательных аллелей внутри популяции или сохранения их частот на низких уровнях.
2 ОПИСАТЕЛЬНАЯ ЧАСТЬ
2.1 Описание экспериментальной площадки – учхоза «Краснодарское» Кубанского ГАУ
Государственный племенной завод учебно-опытное хозяйство «Крас-нодарское» Кубанского ордена Трудового Красного Знамени аграрного университета организован в апреле 1958 г. на базе племенного свиноводческого совхоза «Краснодарский», созданного еще в 1939 г. Как племенной завод хозяйство было утверждено приказом Министерства сельского хозяйства СССР в августе 1979 г.
Учхоз «Краснодарское» расположен в пригороде г. Краснодара на Прикубанской равнине, рельеф которой имеет слабый уклон с востока на запад. Земельный массив учхоза вытянут с востока на запад на 10 км, с севера на юг – на 3,5 км. Центральная усадьба учхоза находится в 20 км от железнодорожной станции « Краснодар-2» и в 6 км от автомагистрали Краснодар–Ростов. Жилые поселки, фермы и другие производственные участки племзавода связаны между собой и базами снабжения и сбыта дорогами с асфальтным покрытием.
На январь 2018 г. земельная площадь учхоза составляет 4080 га, в том числе сельскохозяйственные угодья – 3755 га, из них пашни – 3146 га.
По агрономическим свойствам почвы преимущественно западно-прикавказские тяжелосуглинистые черноземы разной степени выщелоченности, которые характеризуются высоким плодородием и вполне пригодны для выращивания основных сельскохозяйственных культур. По пониженным элементам рельефа размещаются деградированные глинистые черноземы и черноземно-луговые тяжело-суглинистые почвы. Среднегодовое количество осадков составляет около 600 мм. В течение года осадки распределяются неравномерно, при этом большая их часть выпадает осенью и зимой. Средняя продолжительность безморозного периода 180–200 дней. Зима сравнительно теплая. Вегетационный период продолжается 230–250 дней. Отрицательными моментами климата являются восточные суховеи, град, в отдельные годы засуха. Потребности хозяйства в воде удовлетворяются за счет скважин по водопроводу.
В учхозе выращивают озимые зерновые культуры, кукурузу на зерно и силос, сою, подсолнечник и многолетние травы. Однако основная задача отрасли растениеводства – обеспечить животноводство полноценными кормами.
До 2009 г. животноводство было представлено двумя отраслями: свиноводство – это разведение племенных свиней крупно-белой породы и скотоводство – выращивание племенного молодняка черно-пестрой породы и производство товарной продукции молока и мяса. В 2009 г. произошло закрытие фермы по выращиваю свиней в связи с отсутствием эффективности их производства.
В настоящее время животноводство представлено одной отраслью: производство товарной продукции молока и мяса КРС. За весь изучаемый период большую долю в структуре товарной продукции хозяйства занимает продукция животноводства, в основном это молоко (в 2017 г. доля составляла 57 %). Следовательно, основное направление производственной деятельности учхоза – это молочное скотоводство.
Доля растениеводческой продукции в общей структуре товарной продукции за три года колеблется с 25 до 32 %. С 2014 г. хозяйство стало выращивать сою, а с 2015 г. реализовывает ее, удельный вес которой в структуре товарной продукции составил 5 %.
За период с 2013 г. по 2015 г. площадь земель сократилась на 231 га. Это связано с расширением границ города.
Незначительно выросла численность работников по сравнению с 2013 г. на 16 человек, а занятых в сельском хозяйстве – на 21 человека.
Увеличилась стоимость основных производственных фондов на 54 %. Причинами такой динамики является то, что Племзавод УОХ «Краснодарское» ведет постоянную инвестиционную деятельность, связанную с расширением производства и модернизацией имеющихся зданий и сооружений, обновлением техники.
Поголовье крупного рогатого скота остается неизменным, а численность дойного стада за три года увеличилась на 13 %.
Производство молока в 2017 г. составило 8686 т, что на 1859 т больше, чем в 2013 г., или на 27 %. Увеличилось и производство мяса КРС на 30 т, или на 7 %. Увеличение производства продукции животноводства произошло за счет повышения продуктивности животных. Такие показатели были достигнуты благодаря современной системе управления стадом, позволяющая отслеживать производительность работы фермы как ежедневно, так и за длительный период, и новым кормораздатчикам с модулем Wi-Fi в комплексе с современным программным обеспечением, позволяющим оптимизировать систему кормления.
Хозяйство достигло высоких показателей продуктивности коров. Удой на одну корову в 2014 г. составил 9443 кг, а в 2015 г. – 9905 кг. Это позволило занять учхозу в 2013 г. 3-е место, а в 2014, 2015 гг. – 4-е место среди животноводческих хозяйств края.
Увеличился среднесуточный прирост молодняка КРС на 19 % и составил
в 2017 г. 895 г. Незначительно вырос показатель, характеризующий получение приплода телят, – на 13 % по сравнению с 2013 г. Однако показатель по воспроизводству стада остается довольно низким – это 93 теленка на 100 коров, нетелей и телок старше 2-х лет и 68 телят на 100 коров.
В целом, финансовое состояние Племзавода УОХ «Краснодарское» Кубанского ГАУ можно определить как хорошее. Такое состояние позволяет учхозу вкладывать средства в долгосрочные инвестиции.
В 2013 г. было выполнено капитальных вложений на сумму 54399 тыс. руб., а в 2015 – в 1,4 раза больше – на сумму 77320 тыс. руб. Из этих средств
на реконструкцию и строительство в 2013 г. было потрачено 2,5 млн руб.,
а в 2015 г. – 3,1 млн руб.
За 2013–2015 гг. проведены реконструкция корпусов дойного стада и техническое перевооружение доильного зала. В 2013 г. установлена современная система управления стадом, позволяющая отслеживать производительность работы фермы как ежедневно, так и за длительный период. Тогда же были приобретены для растениеводства два трактора Challenger. В 2014 г. приобретены современные кормораздатчики на ферму КРС, трактор «Axion» в подразделение «Растениеводство». В 2015 г. установлен навигатор стада – пока единственный в стране. Также в 2015 г. приобретены сеялка Optima за 7,5 млн руб., автосамосвал КАМАЗ, дисковые бороны Challenger, был полностью реконструирован заправочный комплекс.
Источниками финансирования всех капитальных вложений являются только собственные средства хозяйства.
2.2 Применяемые для получения первичных данных подходы, материалы и методы
Для наиболее полого и качественного выполнения поставленных в проекте задач, нами был разработан ряд методических указаний и карт выполнения процедур. В процессе работы это значительно сократило вероятность ошибок
и применяется нами не только в экспериментальных научно-практических, но
и в педагогических целях при обучении персонала, аспирантов и студентов новым методам и подходам.
2.2.1 Подготовка прибора MiSeq к запуску процесса секвенирования
Перед началом работы:
- В ночь перед секвенированием поместить в холодильник:
- картридж и реагенты (MiSeq Reagent Kit v3 – две коробки);
- пробирку с PAL-библиотекой.
- Убедиться, что есть свежий (1–2 мес) 10%-й Tween 20. Если его нет, то добавить 5 ml 100%-го Tween 20 to 45 ml laboratory-grade water. This results in 10 % Tween 20.
ИНФО:
- Ни в коем случае не менять региональные настройки (в том числе не менять язык на русский)!!!
- Перед и после запуска прибора выполняем «Post-Run Wash» (промывка водой).
- Раз в две недели выполняем «Maintenance Wash» (три промывки – «Tween – Вода – Вода»).
- После «Maintenance Wash» ставим либо секвенирование, либо «Post-Run Wash» и выключаем. В растворе Tween не оставляем!!!
Приготовление свежего раствора Tween:
Всегда используйте свежий моющий раствор для каждого этапа мойки инструмента. Повторное использование моющего раствора может возвращать отходы в линии подачи жидкости.
Для приготовления моющего раствора:
- добавить к 5 мл 100%-го Tween от 20 до 45 мл лабораторной воды. Это создаст 10%-й Tween 20;
- добавьте 25 мл 10%-го Tween от 20 до 475 мл лабораторной воды;
- перевернуть пробирку несколько раз, чтобы смешать содержимое;
- заполните каждый из моющих компонентов свежим моющим раствором. Добавьте 6 мл моющего раствора в каждый резервуар моющего лотка;
- добавьте 350 мл моющего раствора в бутылку на 500 мл.
Подготовительный этап:
Включить прибор MiSeq (за два часа до предполагаемого запуска, чтобы успел охладиться холодильник).
Термостат для денатурации – 98 °C для пробирки 1,5 мл.
Лёд – для денатурации библиотеки.
12 мл 70–80%-го EtOH – смешать 10 мл спирта и 2 мл воды.
Носики: на 10 или 20 мкл – 1 шт.; на 1000 мкл – 1 уп.; на 5 мл.
Самплер: на 10–20 мкл; на 1000 мкл; на 5 мл.
Если не было возможности разморозить картридж с реагентами с вечера, то надо набрать в емкость воду комнатной температуры до ватерлинии на картридже и оставить его на час в ней.
Подготовка прибора к запуску:
Включить прибор (загрузка занимает 5–10 мин).
Если после прошлого запуска прибора прошло менее 7 дней, то выполняем «Post-Run Wash» (промывка водой):
- наполнить все лунки промывочного картриджа (с меткой «H2O») деионизованной водой (по 6 мл);
- налить в PR2-бутыль порядка 200 мл деионизованной воды (треть
бутылки); - запустить промывку.
Если прибор стоял более 7 дней, он потребует сделать «Standby-Wash». При этом, если прошло всего около двух недель – игнорируем это предупреждение, и запускаем «Maintenance Wash» (один час, три шага):
- нажать «Perform wash»;
- выбираем «Maintenance Wash»;
- необходимо убедиться, что использованная ячейка (flow cell) находится в приборе;
- приготовьте свежий моющий раствор: добавьте 25 мл 10%-го Tween 20
к 475 мл лабораторной чистой воды (деионизованной воды); - перевернуть пробирку несколько раз, чтобы смешать содержимое;
- выбрать команду «Next»;
- заполните каждый резервуар промывочного лотка 6 мл промывочного раствора;
- заполните моющую бутылку объемом 500 мл на 350 мл моющим
раствором; - установите лоток «wash tray» и бутылку на прибор;
- выбрать команду «Next»;
- повторить цикл еще два раза с ВОДОЙ вместо раствора Tween!!!
- остатки раствора Tween вылить из использованного картриджа, сполоснуть его водой и поставить сушиться вверх дном;
- создать файл запуска при помощи «Illumina Experiment Manager».
2.2.2 Секвенирование
Взять чистую LoBind пробирку на 1,5 мл, подписать ее как «DAL» (Diluted Amplicon Library) и поставить текущую дату.
Внести в пробирку 600 мкл гибридизационного буфера (HT1 из коробки
с реагентами), 8 мкл Pooled DNA (из PAL-пробирки). Перемешать пипетированием (носиком на 1 мл). Медленно!
98 °C 2 мин → в лёд.
Подготовить файл со списком образцов:
Открыть «Illumina Experiment Manager» – «create sample plate», а потом «create sample sheet». Чтобы найти «manifest file» на рабочем столе компьютера выбрать ярлык «sample sheets» и там найти папку «manifests».
2.2.3 Выключение прибора
Запустить «Post-Run Wash» (промывка водой).
Наполнить все лунки промывочного картриджа (с меткой «H2O») деионизованной водой (по 6 мл).
Налить в PR2-бутыль порядка 200 мл деионизованной воды (треть бутылки).
Запустить промывку.
После окончания промывки ВСЁ (и ячейка, и картридж, и реагенты) остается в приборе!
Запустить Internet Explorer. Откроется страница MiSeq Reporter (http://localhost:8042/default.htm). Убедиться, что в левом окошке в списке анализов появился наш анализ и рядом с ним стоит зеленая галочка. Это значит, что прибор закончил расчеты и его можно выключать. Если галочки нет,
то ждем пока появится.
Выключить прибор.
2.2.4 Промывка прибора
Включить прибор.
Дождаться, когда на экране появится окно «Welcome to Illumina MySeq».
В это время сделать в журнале запись о запуске прибора (указать дату).
«Maintenance Wash» используется, если прибор стоял без дела/был выключен/бездействовал две недели.
- Проверить наличие 100% Tween 20, воды mq, большой пипетки (на
10 мл) и наконечников к ней. - Одеть перчатки. Важно! 10%-й Tween 20 всегда готовится новый, вне зависимости от свежести.
- Вставить в подставку пробирку на 50 мл и подписать «10% Tween 20».
- В нее налить 45 мл воды и в воду добавить 5 мл 100%-го Tween 20
и хорошо пропипетировать. - Из прозрачной бутылки с надписью Tween (стоит справа от секвенатора) вылить все содержимое вне зависимости от свежести, так как Tween хорошо загрязняется цветением. Промыть эту бутылку водой (воды брать немного).
- В промытую бутылку налить 475 мл воды и 25 мл 10%-го Tween 20 (можно тем же наконечником, которым готовили 10%-й Tween 20).
- Перевернуть аккуратно несколько раз бутылку, чтобы хорошо перемешать. Если появились пузырьки (а они появятся), то подождать пока перестанут идти.
- Справа от секвенатора взять плату с надписью «Wash Tray», которая подписана «Tween».
- В каждую из ячеек (круглые дырки) этой платы налить 6 мл Tween 20 из большой бутылки, которую только что приготовили (можно использовать тот же наконечник).
- На экране прибора нажимаем «perform wash» → «perform maintenance wash».
- Проверяем на месте ли ячейка (на экране изображается как и где проверять: слева поднять черную крышка и, придерживая держатель ячейки, нажимаем белую кнопку. Если ячейка на месте, аккуратно закрываем до шелчка держатель, и опускаем на место черную крышку). При этом важно, что ячейка должна ровно стоять на месте для нее предназначенном, при этом может чуть-чуть люфтить, что является нормальным.
- Нажимаем на экране прибора кнопку «Next».
- Открываем черную дверцу, поднимаем серую ручку справа до щелчка вверх (эта ручка располагается между бутылками).
- Выливаем в раковину все из «waste bottle» (бутылка справа) и вставляем ее обратно на место.
- Вытаскиваем бутылку с водой и закрываем крышкой с надписью H2O.
- На место этой бутылки вставляем бутылку с Tween 20.
- Опускаем серую ручку до упора вниз.
- Опускаем ручку (белая ручка справа от бутылки с Tween 20/водой) холодильника и вытаскиваем «wash tray» с водой (воду не выливаем). «Wash tray» с водой ставим внутрь пластикового пакета, чтобы ничего не попало внутрь бутылки.
- На место «wash tray» с водой ставим «wash tray» с Tween 20.
- Нажимаем на экране прибора кнопку «Next».
- После завершения промывания раствором Tween 20 программа на экране покажет «поменяйте wash tray».
- Сменить наконечник у большой пипетки на новый (чистый).
- Достать из полиэтиленового пакета «wash tray» с водой и в каждую дырку долить воды (из бутылки с водой) до уровня отмеченного.
- Открываем черную дверцу. Поднимаем серую ручку, меняем (ВНИМАНИЕ!) бутылку с Tween 20 на бутылку с водой. Опускаем серую ручку до упора.
- Открываем дверцу холодильника и меняем «wash tray» с Tween 20 на «wash tray» с водой.
- Нажимаем «Next».
- Из «wash tray» с Tween 20 выливаем все в раковину. Промываем «wash tray», где был раствор Tween 20, водой. Воду выливаем.
- После окончания работы прибор предложит поменять картриджи
и нажать «Next». Менять ничего не нужно. Тем не менее, мы вытаскиваем бутылку (предварительно подняв серую ручку), потом вытаскиваем из холодильника «wash tray» с водой и доливаем необходимое количество воды в лунки (холодильник держим закрытым). Вставляем обратно бутылку, а также «wash tray» с долитой до визуально адекватного уровня водой. - Нажимаем «Next».
- Внимательно следим за прибором.
- Нажимаем «Done».
- Выключаем компьютер.
2.3 Работа с панелью TruSeq Bovine Parentage Sequencing Panel Illumina
Структура карты исполнения:
1. Гибридизация олигов.
2. Удаление несвязавшихся олигонуклеотидов.
3. Удлинение и лигирование связавшихся олигов.
4. Амплификация библиотеки.
5. Очистка библиотек.
6. Нормализация библиотек.
7. Пулирование библиотек.
8. Приложение.
Перед началом работы:
Достать из первой коробки размораживаться буфер OHS2 (он размораживается очень долго).
Приготовить свежий 10 мл 50 mM NaOH (возраст менее одной недели; приготовленный из 10N NaOH) – 9950 мл Н2О + 50 мкл 10N NaOH.
Убедиться, что в амплификатор внесена программа отжига TruSeq Bovine Hyb и программа амплификации библиотеки Lib_amp.
Подготовить:
- любой заклеивающийся 96-луночный планшет для разведения ДНК;
- носики: на 20 мкл – 3 уп., на 100 мкл – 5 уп., на 1000 мкл – 1 уп.;
- пленку для заклеивания планшета – 1 шт.;
- самплер: на 20 мкл, на 100–200 мкл, на 1000 мкл.
| Box 1 (минус 20 °C) |  |
| Box 4 (минус 20 °C) |  |
| TruSeq Custom Amplicon Index Kit (384 samples) |
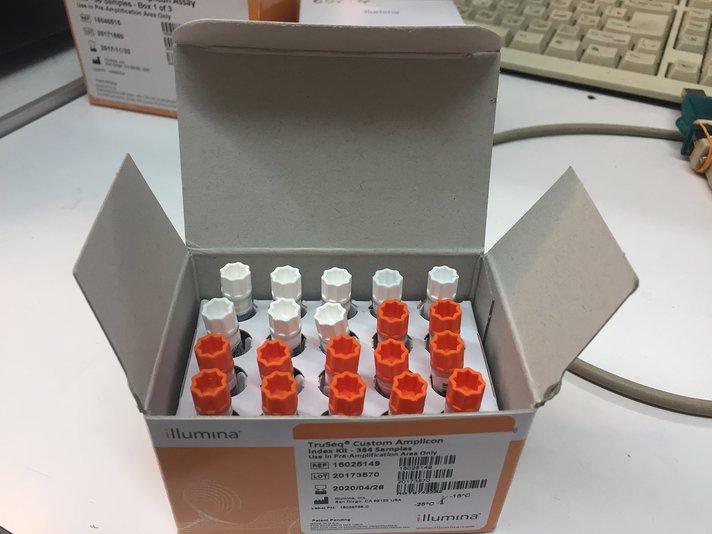 |
| Index Adapter Replacement Caps |
Исходная ДНК:
High-quality genomic DNA 50 ng (10–25 ng/?l). При необходимости разведите ДНК до данных концентраций (10–25 ng/?l).
Разводим до 75 нг в 15 мкл в отдельной плашке. Из нее потом будем многоканальной пипеткой отбирать по 10 мкл (50 нг).
ACD1 и ACP1
Включайте ACD1 и ACP1 в каждый синтез библиотеки. Использование этих контролей поможет специалистам из техподдержки Illumina оказать нам помощь в случае необходимости. При отсутствии данного контроля помощь не предоставляется!
Внимание! Запишите информацию об образцах ПЕРЕД началом приготовления библиотеки.
С этого момента до «Безопасной Точки Остановки» все выполняется без перерыва и остановок!
Используются ТОЛЬКО наконечники с фильтром.
2.3.1 Гибридизация пула олигонуклеотидов
Расходники на минус 20 °C
Box 1:
- ACD1 – Amplicon Control DNA;
- ACP1 – Control Oligo Pool;
- OHS2 – Oligo Hybridization for Sequencing 2.
Эта коробка также содержит метки штрих-кодов HYP, FPU и IAP.
Box 4:
- BVP1 – Bovine Parentage Oligo Tube.
| Геномная ДНК коров | Достать из заморозки за 30 мин до начала исследования для достижения образца комнатной температуры.
Не вортексировать! Перемешать обстукиванием, сбросить кратким центрифугированием |
| – ACD1
– ACP1 – BVP1 |
1. Разморозить при комнатной температуре.
2. Вортексировать. 3. Сбросить центрифугированием. |
| – OHS2 | 1. Разморозить при комнатной температуре.
2. Энергично вортексировать перед каждым использованием. 3. Ввиду вязкости реагента отбирать и перемешивать медленно! 4. Посмотреть на просвет, убедиться, что весь осадок (мелкие прозрачные кристаллы) растворился. Важно его долго и тщательно вортексировать!!! До полного растворения! |
Внимание! Не смешивайте BVP1 и OHS2 для хранения. В смешанном виде BVP1 становится нестабильным, даже если хранится в замороженном виде.
При сбрасывании планшетов в противовесный планшет внести по 1,5 мл воды!
- Установить программу для «TruSeq Bovine Hyb» на амплификаторе «Applied Biosystems» (либо прогреть воздушный инкубатор до 37 °C).
- Наклеить «HYP barcode label» на новую 96-луночную ПЦР-плашку.
- Смешать 3,5 мл (3 × 1 мл + 500 мкл) OHS2 и 500 мкл BVP1. Тщательно перемешать пипетированием.
- Внести в 95 лунок (оставить одну для контроля) по 40 мкл полученной смеси (описано в п. 3).
- В последнюю лунку:
- внести 5 μl TE или воды и 5 μl ACD1;
- добавить 5 μl ACP1 в контрольную лунку, содержащую ACD1 (96-я);
- добавить 35 μl OHS2 в контрольную лунку (96-я).
- Добавить 50 ng (по 10 мкл) gDNA в первые 95 лунок.
- Заклеить планшет термопленкой.
- Центрифугировать при 1000×g в течение минуты.
- Установить плашку в термостат (Veriti) и запустить программу TruSeq Bovine Hyb.
- Пока инкубируется (1 ч 15 мин) – остатки разведенной ДНК упаковать в самоклеющуюся пленку и выбросить в биоотходы.
- Переходим к следующему этапу – достаем реактивы из морозильной камеры и холодильника.
- Через 30 мин от начала инкубации собираем FPU (Filter Plate Unit)
и промываем ее.
2.3.2 Удаление несвязавшихся олигонуклеотидов
Этот этап удаляет несвязавшиеся олигонуклеотиды из геномной ДНК, используя размерный фильтр. Две промывки с использованием SW1 гарантируют удаление несвязавшихся олигов. Третья промывка использующая UB1 удаляет остатки буфера SW1 и подготавливает пробы к следующему шагу.
Реагенты на минус 20 °С
Box 1:
- ELM4 – Extension-ligation Mix 4 – достать из морозилки и оставить нагреваться до комнатной температуры.
Реагенты на 2–8 °C
Box 1:
- SW1 – Stringent Wash 1;
- UB1 – Universal Buffer 1.
Достать реагенты из холодильника и оставить нагреваться до комнатной температуры.
Дополнительные приспособления
| Filter plate with lid |  |
| Adapter collar | |
| Midi plate |  |
Носики на 100 мкл (для многоканальной пипетки) – 2 уп.
Собрать FPU – Filter Plate Unit – от верха к низу:
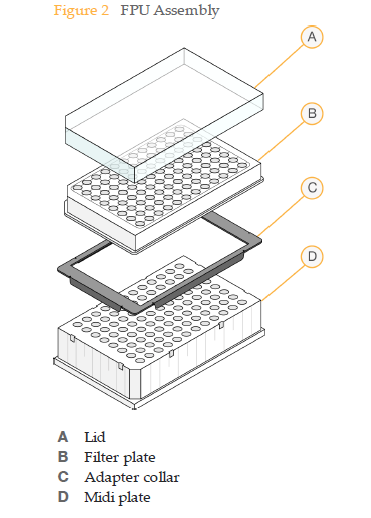
1. Маркируйте законченную сборку FPU.
2. Промыть лунки для анализа следующим образом. Используйте только новые чистые лунки:
- добавить 45 мкл SW1 в каждую лунку (одним наконечником);
- накройте пластину FPU;
- центрифугировать при 2400×g в течение 10 мин.
Внимание! Если значительное количество (> 15 мкл / лунка) остаточного буфера остается в нескольких лунках (≥ 10 лунок / пластина), переключитесь на новую фильтровальную пластину.
3. Убедитесь, что нагревательный блок остыл до 40 °C.
4. Снять блок с нагрева.
5. Центрифугировать при 1000 × g в течение минуты.
6. Перенести каждый образец на пластину FPU (каждой лунке свой наконечник).
7. Накрыть и центрифугировать при 2400×g в течение 2 мин.
8. Промыть два раза следующим образом:
- добавить 45 мкл SW1 в каждую лунку для образца;
- накрыть и центрифугировать при 2400×g в течение 2 мин;
- если SW1 не полностью удален, центрифугируйте в течение 10 мин.
9. Откажитесь от потока.
10. Соберите пластину FPU для дальнейшего использования.
11. Добавить 45 мкл UB1 в каждую лунку для образца.
12. Накройте и центрифугируйте при 2400×g в течение 2 мин.
13. Если UB1 не полностью удален, отцентрифугируйте в течение 10 мин.
2.3.3 Достройка и лигирование связавшихся олигонуклеотидов
Этот шаг включает в себя гибридизацию прямого и обратного олигонуклеотидов, построение начиная с прямого праймера ДНК-полимеразой комплиментарной копии целевого участка, с последующим лигированием с 5′-концом обратного праймера с использованием ДНК-лигазы. Результатом является образование продуктов, содержащих целевые области ДНК, фланкированные последовательностями, необходимыми для амплификации.
Реагенты – ELM4 (уже разморожен на предыдущем шаге).
Процесс:
1. Добавьте 45 мкл ELM4 в каждую лунку для образцов в планшете FPU.
2. Запечатайте и инкубируйте при 37 °С в течение 45 мин.
3. Во время инкубации перейдите к следующему шагу.
2.3.4 Амплификация библиотеки
Этот шаг усиливает продукты расширения-лигирования и добавляет адаптеры индекса 1 (i7), адаптеры индекса 2 (i5) и последовательности, необходимые для формирования кластера.
Реагенты на минус 20 °С
Box 1:
- PMM2– PCR Master Mix 2;
- TDP1 – TruSeq DNA Polymerase 1.
| PMM2 | 1. Разморозить при комнатной температуре (20 мин).2. Перемешать вортексированием.3. Сбросить центрифугированием. |
| Index i5 adapters (A5XX)
Index i7 adapters (A7XX) |
1. Разморозить при комнатной температуре (20 мин).2. Перемешать вортексированием.3. Сбросить центрифугированием (используя пробирки объемом 1,5 мл в качестве адаптеров). |
| TDP1 |
TruSeq Custom Amplicon Index Kit
Box 1 (минус 20 °C) – i5 и i7 Index Primers.
Box 2 (Room Temp) – белые и оранжевые одноразовые крышечки для праймеров.
About Reagents
- PMM2/TDP1:
- комбинируйте PMM2 и TDP1 непосредственно перед использованием. Не смешивайте и не храните комбинированную смесь PMM2 / TDP1;
- при смешивании перемешивайте тщательно.
Расходники
| 96-well skirted PCR plate |  |
| IAP (Indexed Amplification Plate) barcode label |
- Расположите адаптеры Index 1 (i7) в столбцах 1–12.
- Расположите адаптеры Index 2 (i5) в строках A – H.
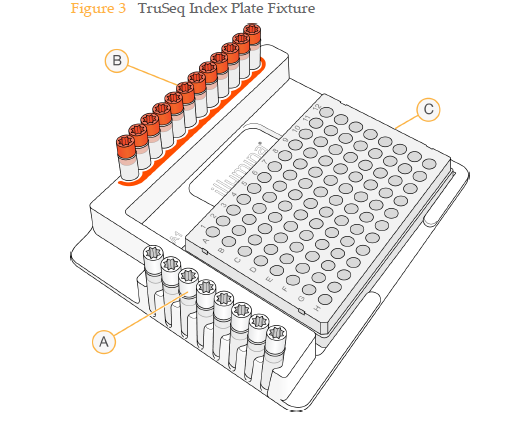

1. Нанесите метку IAP на новый 96-луночный планшет для ПЦР.
2. Разместите планшет на TruSeq Index Plate Fixture.
3. Используя многоканальную пипетку, добавьте 4 мкл каждого адаптера Index 1 (i7) в каждую колонку. Замените крышку на каждой переходной трубке i7 новой оранжевой крышкой.
4. Используя многоканальную пипетку, добавьте 4 мкл каждого адаптера Index 2 (i5) в каждый ряд. Замените крышку на каждой переходной трубке i5 новой белой крышкой.
5. Добавьте 56 мкл TDP1 в полную пробирку (2,8 мл) к PMM2. переверните, чтобы смешать.
6. Перенести 22 мкл смеси PMM2 / TDP1 на планшет IAP.
7. Извлеките пластину FPU из инкубатора и сделайте следующее:
-
- заменить алюминиевую фольгу на крышку фильтра;
- центрифугировать при 2400×g в течение 2 мин (40 мин ×
2000 об./мин); - добавить 25 мкл 50 mm NaOH в каждую лунку. Перемешать пипетированием;
- инкубировать при комнатной температуре в течение 5 мин.
8. Перенесите элюированные образцы с планшета FPU на планшет IAP следующим образом:
-
- используя тонкие наконечники, пипеткой перемешать в первой колонке планшета FPU;
- перенести 20 мкл NaOH в соответствующий столбик планшета IAP. Перемешать пипетированием;
- перенести оставшиеся колонки с планшета FPU на IAP;
- выбросить планшет midi для сбора отходов.
9. Закрыть плашку силиконовым матом. Центрифугировать при 1000×g в течение минуты.
10. Перенесите планшет IAP в зону пост-амплификации.
11. Поместите на предварительно запрограммированный термоциклер
и запустите программу ПЦР (Lib_amp).
Безопасная Точка Остановки!
Если вы останавливаетесь, закройте пластину и храните при температуре от 2 до 8 °C до 2 дней. Кроме того, оставьте ее на термоциклере на ночь.
2.3.5 Очистка синтезированной библиотеки
Подготовить:
- достать AMPure XP beads (хранится в холодильнике ПЦР-зоны) нагреваться до комнатной температуры (~ 30 мин).
- EBT – Elution Buffer with Tris (хранится в форезной комнате при комнатной температуре).
- 96-well midi plates (2 шт.).
- Баркодированные метки (в Box 3 в ПЦР-зоне):
- CLP – Cleanup Plate – нанести на «midi plate»;
- LNP – Library Normalization Plate – нанести на другую «midi plate».
- Самоклеющаяся пленка для планшета.
- Приготовить 40 мл свежего 80%-го ethanol (EtOH).
- 96-луночный магнитный штатив.
Процесс:
1. Центрифугировать планшет IAP при 1000×g в течение минуты.
2. Опционально: отобрать по 5 мкл и прогнать их в 4%-м агарозном геле. Должны быть ПЦР-продукты в районе 200–300 п.о.
3. Добавьте 60 мкл гранул AMPure XP в каждую лунку планшета CLP.
4. Перенесите весь супернатант из каждой лунки планшета IAP в соответствующую лунку планшета CLP.
5. Закупорить и встряхнуть при 1800 rpm в течение 2 мин.
6. Инкубировать при комнатной температуре в течение 10 мин.
7. Сбросить капли кратковременным центрифугированием.
8. Поместите на магнитную подставку и подождите, пока жидкость не станет прозрачной (~ 2 мин).
9. Удалите весь супернатант из каждой лунки.
10. Промыть два раза следующим образом:
- добавьте 200 мкл 80%-го EtOH в каждую лунку для образца;
- инкубировать на магнитной подставке в течение 30 с;
- удалить весь супернатант из каждой лунки.
11. Используя 20 мкл пипетки, удалите остаточный EtOH из каждой лунки.
12. Снимите с магнитной подставки и высушите на воздухе в течение 10 мин.
13. Добавьте 30 мкл EBT в каждую лунку.
14. Запечатать и встряхнуть при 1800 rpm в течение 2 мин.
15. Убедитесь, что все шарики ресуспендированы. При необходимости, пипеткой перемешать и повторить шаг встряхивания.
16. Инкубировать при комнатной температуре в течение 2 мин.
17. Сбросить капли кратковременным центрифугированием.
18. Поместить на магнитную подставку и подождать, пока жидкость не станет прозрачной (~ 2 мин).
19. Перенести 20 мкл супернатанта из каждой лунки планшета CLP
в соответствующую лунку планшета LNP.
20. Запечатать и центрифугировать при 1000×g в течение минуты.
2.3.6 Нормализация (выравнивание) библиотеки
Этот шаг нормализует количество каждой библиотеки для сбалансированного представления в объединенных библиотеках. Только образцы, содержащие ДНК, требуют обработки на последующих этапах.
Подготовить
Box 2:
- LNB1 – Library Normalization Beads 1.
Box 3:
- LNA1 – Library Normalization Additives 1;
- LNW1 – Library Normalization Wash 1;
- LNS2 – Library Normalization Storage buffer 2;
- SGP – Storage Plate – barcode label.
| LNB1 | 8 °C | Оставить на 30 мин на столе для доведения до RT.
Вортексировать как минимум минуту. Периодически переворачивать для перемешивания. Убедиться, что дно пробирки свободно от осадка. |
| LNA1 | Минус 20 °C | Разморозить при комнатной температуре.
Оставить на 30 мин на столе для доведения до RT. Перемешать вортексированием. Убедиться, что весь осадок растворился. |
| LNW1 | 8 °C | Оставить на 30 мин на столе для доведения до RT. Убедиться, что весь осадок растворился. |
| LNS2 | RT | Если заморожено, разморозить при комнатной температуре 20 мин. Перемешать вортексированием. |
- 0,1N NaOH (свежеприготовленный).
- 96-well PCR plate, skirted.
- Коническая пробирка объемом 15 мл.
Процесс:
1. Для 96 образцов добавьте 4,4 мл LNA1 в новую коническую пробирку на 15 мл.
2. Используйте пипетку на 1000 мкл для того, чтобы ресуспендировать LNB1.
3. Перенести 800 мкл LNB1 в пробирку объемом 15 мл с LNA1. Перемешать переворачиванием.
4. Добавьте 45 мкл LNA1 / LNB1 в каждую лунку планшета LNP.
5. Заклеить планшет пленкой и перемешать при 1800 rpm в течение 30 мин. Длительность, не превышающая 30 мин, может повлиять на представление библиотеки и плотность кластера.
6. Поместить на магнитную площадку и подождать, пока жидкость не стнает прозрачной (~ 2 мин).
7. Удалить весь супернатант.
8. Убрать с магнитной платформы.
9. Промыть два раза следующим образом:
- добавить 45 мкл LNW1 в каждую лунку библиотеки;
- закрыть и встряхнуть при 1800 rpm в течение 5 мин;
- поместить на магнитную платформу и подождать пока жидкость не станет прозрачной (~ 2 мин);
- удалить весь супернатант.
10. Использовать 20 мкл пипетки для удаления остатков LNW1 из каждой лунки.
11. Снять плашку с магнитного штатива.
12. Добавить 30 мкл свежего 0,1N NaOH в каждую лунку.
13. Закрыть и встряхнуть при 1800 rpm в течение 5 мин.
14. Если библиотеки не ресуспендируют, пипеткой перемешать, а затем встряхнуть при 1800 rpm в течение 5 мин.
15. Поместить планшет LNP на магнитную платформу и подождать пока жидкость не станет прозрачной (~ 2 мин).
16. Пометить новую плашку меткой SGP.
17. Добавить 30 мкл LNS2 в каждую лунку планшета SGP.
18. Перенести 30 мкл супернатанта из каждой лунки планшета LNP в соответствующую лунку планшета SGP. Перемешать пипетированием.
19. Закрыть и центрифугировать при 1000×g в течение минуты.
Безопасная Точка Остановки!
Если вы останавливаетесь, закройте пластину и храните при температуре от минус 25 до минус 15 °С до 30 дней.
2.3.7 «Пулирование» (объединение) библиотек
Объединение библиотек соединяет одинаковые объемы нормализованных библиотек в одной пробирке. После объединения разбавьте и денатурируйте пул библиотек перед загрузкой библиотек для запуска секвенирования.
Подготовить:
- PAL – Pooled Amplicon Library – этикетка со штрих-кодом.
- Микроцинтрефужные пробирки LoBind (объемом 1,5 мл).
- RNase/DNase-free 8-tube strips and caps.
Процесс:
1. Если SGP плашка хранилась замороженной, то:
- разморозить ее при комнатной температуре;
- центрифугироват при 1000×g в течение минуты;
- перемешать пипетированием.
2. Поставить размораживаться реагенты для секвенатора (см. инструкцию «Запуск секвенирования»).
3. Наклеить на новую пробирку объемом 1,5 мл метку PAL.
4. Центрифугировать при 1000×g в течение минуты.
5. Разместить 5 мкл каждой библиотеки на 8 колонок, колонку за колонкой. Закрыть планшет и хранить при минус 15–25 °С.
6. Поместить содержимое 8-ми колонок на планшет PAL. Перемешать пипетированием.
7. Денатурируйте и разбавляйте объединенные библиотеки до концентрации нагрузки для используемого вами инструмента. Смотрите руководство по денатурированию и разбавлению библиотек для вашего инструмента (600 мкл гибридизационного буфера (HT1 из коробки с картриджем) + 8 мкл Pooled DNA. 98 °C 2 мин → в лёд).
8. Хранить планшет PAL при минус 15–25 °C для дальнейшего использования.
2.3.8 Приложение
Программа для отжига праймеров (TruSeq Bovine Hyb):
Программа для амплификации библиотеки (Lib_amp):
- 95 °C for 3 minutes;
- 25 cycles of:
- 95 °C for 30 seconds
- 66 °C for 30 seconds
- 72 °C for 60 seconds
- 72 °C for 5 minutes
Хранить при 10 °C.
2.4 Измерение концентрации ДНК
Для измерения концентрации использовать краситель PicoGreen либо Qubit dsDNA Assay Kit. (УФ-методы детекции в нашем случае гораздо менее точные).
Внимание! Не использовать для раствора стеклянную посуду!
Содержимое набора Invitrogen «Quant-iTTM PicoGreenTM dsDNA Assay Kit»:
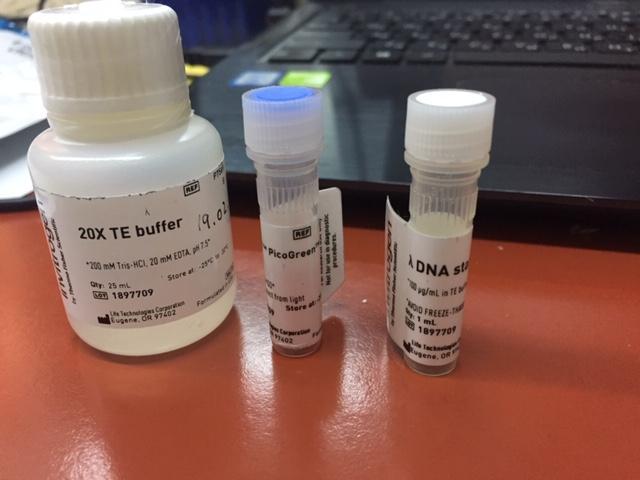
Подготовка красителя:
Внимание! Дайте реагенту Quant-iT™ PicoGreen® нагреться до комнатной температуры, прежде чем открывать пробирку с ним.
Для наилучшего результата раствор необходимо использовать в течение нескольких часов после приготовления.
Защищайте приготовленный раствор с красителем от света (помещая его
в темное место или накрывая фольгой).
Проверить корректность установки даты и времени в приборе (Settings → «Instrument settings» → «Date/Time»). При необходимости установить верные значения.
1. В чистую, объемом 15 мл пробирку внести 1005 мкл 20х TE буфера.
2. Добавить в пробирку 19095 мкл Н2О.
3. Добавить к ним 101 мкл красителя.
4. Тщательно перемешать.
Приготовить контрольную ДНК для построения кривой (0 и 100 нг в 5 мкл):
1. Внести в пробирку объемом 0,2мл 8 мкл 1хТЕ буфера.
2. Добавить 2 мкл контрольной ДНК из набора (100нг/мкл). Хорошо перемешать пипетированием.
3. Подготовить две оптически прозрачные пробирки объемом 0,5 мл.
4. Одну подписать как К1, другую – К2:
- в К1 внести 200 мкл раствора для измерения концентрации;
- в К2 внести 195 мкл раствора для измерения и 5 мкл разведенной контрольной ДНК (с шага b).
5. В оптически прозрачные пробирки объемом 0,5 мл внести по 195 мкл.
6. Внести в каждую пробирку по 5 мкл соответствующей коровьей ДНК.
7. Внести в пробирку К1 5 мкл раствора ДНК второго разведения
(38 + 2 мкл).
8. Внести в пробирку К2 5 мкл раствора ДНК первого разведения
(8 + 2 мкл).
9. Включить прибор, подключить его USB-шнуром к компьютеру.
10. Выбрать на первом экране программу PicoGreen.
Опционально. Если было проведено новое разведение красителя, то нажимаем строку «Read standarts». Если краситель из того же разведения, то сразу нажимаем «Run samples».
Разведение до нужной концентрации
High-quality genomic DNA 50 ng (10–25 ng/?l). При необходимости разведите ДНК до данных концентраций (10–25 ng/?l).
Разводим до 75 нг в 15 мкл в отдельной плашке. Из нее потом будем многоканальной пипеткой отбирать по 10 мкл (50 нг).
3 ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
3.1 Выделение геномной ДНК и создание библиотек для секвенирования с использованием панели TruSeq Bovine Parentage Sequencing Panel Illumina
Выделение тотальной геномной ДНК производилось из образцов цельной крови коров по ранее освоенным методикам. В частности использовались наборы ДНК-ЭКСТРАН-1 («Синтол», Россия) и К-сорб («Синтол», Россия).
3.1.1 Выделение ДНК набором ДНК-ЭКСТРАН-1 («Синтол», Россия)
Примечание:
– для выделения следует использовать цельную кровь с антикоагулянтами;
– если в лизирующем растворе № 2 есть осадок, раствор следует прогреть при 60 °С в течение 15–30 мин до растворения осадка;
– если после лизиса с лизирующим буфером № 2 не происходит выпадение белого осадка и активного помутнения раствора, следует охладить раствор во льду 1–5 мин.
Протокол:
1. В чистые, объемом 1,5 мл пробирки внести по 300 мкл цельной крови.
2. В пробирки с образцами добавить по 900 мкл лизирующего буфера № 1.
3. Содержимое пробирок тщательно перемешать переворачиванием, избегая образования пены.
4. Инкубировать образцы при комнатной температуре в течение 10 мин, периодически перемешивая их переворачиванием.
5. Центрифугировать пробирки 2 мин при 1300 об./мин.
6. Убрать супернатант, оставив в пробирке ~ 20 мкл надосадочной
жидкости.
7. Ресуспендировать осадок на вортексе.
8. Добавить в пробирки по 300 мкл лизирующего буфера № 2 и 10 мкл протеиназы К.
9. Перемешать содержимое пробирок на вортексе.
10. Инкубировать образцы при температуре 65 °С до полного растворения осадка.
11. Охладить пробирки во льду 3–5 мин.
12. Сбросить пробирки на центрифуге.
13. К лизату добавить 100 мкл осаждающего раствора № 1. Убедиться в образовании белого осадка, если осадок не образуется – дополнительно охладить образец во льду в течение минуты.
14. Перемешать содержимое пробирок на вортексе.
15. Центрифугировать пробирки 2 мин при 13000 об./мин.
16. Супернатант перенести в чистые, объемом 1,5 мл пробирки и подписать.
17. Добавить к супернатанту осаждающий раствор № 2 и перемешать переворачиванием до конденсации видимого осадка ДНК (12–15 раз).
18. Центрифугировать смесь № 2 мин при 13000 об./мин.
19. Очень осторожно удалить супернатант, отслеживая положение осадка.
20. К осадку добавить 400 мкл промывающего раствора.
21. Тщательно перемешать содержимое пробирки, переворачивая ее
(10–15 раз).
22. Центрифугировать содержимое пробирки 2 мин при 13000 об./мин.
23. Тщательно, с максимальной осторожностью удалить супернатант, отслеживая положение осадка.
24. Оставить пробирки открытыми (в штативе) в чистом, работающем ПЦР-боксе для просушки при комнатной температуре до полного испарения жидкости.
25. Внести в пробирки по 50 мкл элюирующего раствора.
26. Тщательно перемешать содержимое пробирок на вортексе.
27. Инкубировать пробирки в термостате 5 мин при температуре 60 °С, после чего остудить их при комнатной температуре и снова перемешать на вортексе.
28. Кратковременно центрифугировать пробирки для удаления раствора ДНК со стенок и крышки пробирки.
29. Полученный раствор ДНК можно использовать немедленно или оставить для кратковременного (до двух недель) хранения при плюс 4–8 °С либо долговременного хранения при минус 20 °С.
3.1.2 Выделение ДНК набором К-сорб («Синтол», Россия)
1. Перед началом работы сделать аликвоту элюирующего буфера требуемого объема (от 50 до 120 мкл на образец).
2. Внести во все пробирки для исследуемых образцов и тщательно перемешать:
– 400–500 мкл физиологического раствора;
– 200 мкл лизирующего буфера (раствор № 2);
– 10 мкл протеиназы К (хранение при минус 20 °С);
– 10 мкл 1M раствора DTT.
3. Приготовить и внести в пробирки образцы, которые готовятся путем иссечения фрагмента марли с предварительно нанесенной на нее и высушенной кровью, площадь фрагмента – от 0,3 до 1 см2.
4. Тщательно перемешать содержимое пробирок.
5. Термостатировать пробирки при 65 °С в течение 1–2 ч.
6. По окончании термостатирования извлечь пробирки с образцами из термостата, перевести его на 70 °С, поставить в него пробирки с аликвотами элюирующего буфера (до окончания выделения).
7. Выполнить краткосрочное центрифугирование пробирки с образцами для удаления испарений и капель со дна крышки и стенок, минимальная длительность центрифугирования при максимальном числе оборотов в минуту.
8. В каждую пробирку внести по 200 мкл осаждающего раствора. Тщательно перемешать. Выполнить краткосрочное центрифугирование.
9. Жидкость из пробирок перенести в колонки К-сорб. Центрифугировать колонки минуту при 8.000 rpm. По окончанию центрифугирования проконтролировать полное прохождение элюента через колонку. В случае неполного прохождения увеличить время центрифугирования и/или число оборотов.
10. Элюат удалить из пробирок-коллекторов в контейнер для жидких медицинских отходов. Колонки вернуть в пробирки-коллекторы.
11. Внести в колонки по 400 мкл промывочного раствора № 1. Центрифугировать минуту при 8.000 rpm, удалить элюат, колонки вернуть в пробирки-коллекторы.
12. Внести в колонки по 400 мкл промывочного раствора № 2. Центрифугировать минуту при 8.000 rpm, удалить элюат, колонки вернуть в пробирки-коллекторы.
13. Внести в колонки по 200 мкл промывочного раствора № 2. Центрифугировать 2 мин при 13.000 rpm (или при максимальном числе оборотов центрифуги).
14. Перенести колонки в чистые пробирки. Центрифугировать 3 мин при 13.000 rpm (или при максимальном числе оборотов центрифуги).
15. Перенести колонки в чистые пробирки.
16. Внести в каждую колонку требуемую аликвоту элюирующего буфера, прогретого до 70 ºС. Пробирки брать из термостата по мере расходования предыдущих.
17. Инкубировать колонки под элюирующим буфером 3–5 мин при комнатной температуре в горизонтальном положении.
18. Центрифугировать минуту при 8.000 rpm.
19. По окончании центрифугирования проконтролировать полноту прохождения элюирующего буфера во всех пробирках. Колонки поместить в пакет для твердых медицинских отходов. Пробирки маркировать с указанием данных образца и даты выделения.
20. Полученные образцы ДНК хранить при 4–8 °С (до одной недели). При более длительном сроке хранения температура должна быть минус 20 °С.
3.1.3 Протокол для фрагментации дцДНК и приготовления NSG-библиотек из 5–500 нг очищенной дцДНК
Исходный материал: 5–500 нг очищенной дцДНК (например, бактериальной геномной ДНК или геномной ДНК человека).
1. Фрагментация дцДНК.
В ПЦР-пробирку, установленную на лед, внести следующие компоненты:
| Компонент | Объем, мкл |
| 10x SD-буфер | 2,5 |
| MgCl2 (50 мМ) | 1,75 |
| дцДНК (50–500 нг) | 1–5 |
| Смесь ферментов для фрагментации (SDpol + ДНКаза I) | 1 |
| ddH2O | До 25 |
Инкубировать в ПЦР-амплификаторе в следующем режиме:
| Температура, оС | Время |
| 30 | 10–60* мин |
| 70 | 20 мин |
2. Лигирование индексированных вилочковых адаптеров с фрагментированной ДНК.
В пробирку с фрагментированной ДНК добавляют следующие компоненты, и перемешивают их встряхиванием:
| Компонент | Объем, мкл |
| Фрагментированная дцДНК | 25 |
| 10x T4 лигазный буфер | 5 |
| Индексированные вилочковые адаптеры* (12,5 мкM) | 2 |
| ddH2O | 16 |
| T4 ДНК-лигаза (5 Ед/мкл) | 2 |
Инкубировать лигазную смесь (50 мкл) в течение 20 мин при комнатной температуре. После инкубации развести лигазную смесь 0,1х ТЕ-буфером до конечного объема 100 мкл. Образцы на этом этапе можно заморозить для хранения при минус 20 °C.
*В наборе используются индексированные адаптеры Illumina. Набор содержит следующие адаптеры:
| № | Индекс Illumina |
Структура |
| 1 | Universal | 5’AATGATACGGCGACCACCGAGATCTACACTCTTTCCCTACACGACGCTCTTCCGATC*T |
| 2 | Index 2 | 5’PGATCGGAAGAGCACACGTCTGAACTCCAGTCACCGATGTATCTCGTATGCCGTCTTCTGCTTG |
| 3 | Index 3 | 5’PGATCGGAAGAGCACACGTCTGAACTCCAGTCACTTAGGCATCTCGTATGCCGTCTTCTGCTTG |
| 4 | Index 4 | 5’PGATCGGAAGAGCACACGTCTGAACTCCAGTCACTGACCAATCTCGTATGCCGTCTTCTGCTTG |
| 5 | Index 5 | 5’PGATCGGAAGAGCACACGTCTGAACTCCAGTCACACAGTGATCTCGTATGCCGTCTTCTGCTTG |
| 6 | Index 6 | 5’PGATCGGAAGAGCACACGTCTGAACTCCAGTCACGCCAATATCTCGTATGCCGTCTTCTGCTTG |
| 7 | Index 9 | 5’PGATCGGAAGAGCACACGTCTGAACTCCAGTCACGATCAGATCTCGTATGCCGTCTTCTGCTTG |
| 8 | Index 10 | 5’PGATCGGAAGAGCACACGTCTGAACTCCAGTCACTAGCTTATCTCGTATGCCGTCTTCTGCTTG |
| 9 | Index 11 | 5’PGATCGGAAGAGCACACGTCTGAACTCCAGTCACGGCTACATCTCGTATGCCGTCTTCTGCTTG |
| 10 | Index 12 | 5’PGATCGGAAGAGCACACGTCTGAACTCCAGTCACCTTGTAATCTCGTATGCCGTCTTCTGCTTG |
| 11 | Index 14 | 5’PGATCGGAAGAGCACACGTCTGAACTCCAGTCACAGTTCCGTATCTCGTATGCCGTCTTCTGCTTG |
| 12 | Index 15 | 5’PGATCGGAAGAGCACACGTCTGAACTCCAGTCACATGTCAGAATCTCGTATGCCGTCTTCTGCTTG |
| 13 | Index 16 | 5’PGATCGGAAGAGCACACGTCTGAACTCCAGTCACCCGTCCCGATCTCGTATGCCGTCTTCTGCTTG |
3. Очистка ДНК, лигированной с адаптерами.
Необходимо дать шарикам AMPure XP прогреться до комнатной температуры не менее 30 мин перед использованием. Для ресуспендирования встряхнуть шарики AMPure XP на вортексе.
Добавить 80 мкл (0,8X) ресуспендированных шариков к реакции лигирования адаптеров. Хорошо перемешать пипетированием по меньшей мере 10 раз. Манипуляции следует проводить осторожно, чтобы исключить всю жидкость из наконечника во время последнего смешивания. Также можно воспользоваться смешивание на вортексе в течение 3–5 с. Если после центрифугирования
образцы центрифугируются, необходимо остановить центрифугирование до того, как шарики начнут оседать.
Инкубировать образцы на столе не менее 5 мин при комнатной температуре.
Чтобы отделить шарики от супернатанта, планшет/пробирки помещают на соответствующую магнитную подставку. При необходимости образец центрифугируют, чтобы собрать жидкость со стенок лунки пробирки или планшета перед постановкой на магнитную подставку. Через 5 мин (или когда раствор станет прозрачным) удаляют супернатант. Это действие надо проводить осторожно, чтобы не потревожить шарики, содержащие целевую ДНК.
Добавить 200 мкл 80%-го свежеприготовленного этанола в пробирку/планшет, находящиеся в магнитной подставке. Инкубировать при комнатной температуре в течение 30 с, а затем осторожно удалить и отбросить супернатант.
Повторяют эту стадию промывки один раз, чтобы получить в итоге две стадии промывки. Обязательно удаляют всю видимую жидкость после второй промывки. При необходимости, кратковременно центрифугируйте пробирку/планшет, поместите обратно на магнит и удалите следы этанола с помощью автоматического дозатора на 20 мкл. Это действие надо проводить осторожно, чтобы не потревожить шарики, содержащие целевую ДНК.
Высушить шарики на воздухе до 5 мин, пока пробирка/планшет находятся на магнитной подставке с открытой крышкой.
Внимание: важно не пересушивать шарики! Это может привести к снижению выделения ДНК-мишени. Образцы следует элюировать, когда шарики остаются темно-коричневого цвета и выглядят блестящими, но при этом вся видимая жидкость испарилась. Когда шарики становятся светло-коричневыми,
и их поверхность начинает покрываться трещинами, они являются уже пересушенными.
Снимите пробирку/планшет с магнитной подставки. Элюируйте ДНК-мишень с шариков, добавляя 22–23 мкл 10 мМ Трис-HCl или 0,1X TE. Хорошо перемешайте суспензию пипетированием 10 раз или встряхиванием на вортексе. Инкубируйте суспензию не менее 2 мин при комнатной температуре. При необходимости шарики можно центрифугировать в течение короткого времени, чтобы собрать жидкость со стенок пробирки или лунок планшета, прежде чем поставить их обратно на магнитную подставку.
Поместите пробирку/планшет на магнитную подставку. Через 5 мин (или когда раствор станет прозрачным) перенести 20 мкл образца в новую ПЦР-пробирку для амплификации.
Примечание: образцы можно хранить при минус 20 °C.
4. ПЦР-амплификация лигированной ДНК адаптера.
В новую ПЦР-пробирку обавить следующие компоненты:
| Компонент | Объем, мкл |
| ДНК, лигированная с адаптерами | 20 |
| Смесь праймеров для ПЦР* (20 мкM – по 10 мкМ каждого праймера) |
4 |
| 10x SD-буфер | 5 |
| MgCl2 (50 мM) | 3,5 |
| Смесь ферментов для амплификации (20 Ед/мкл) | 1 |
| ddH2O | 16,5 |
| Примечание: * – ПЦР-праймеры: P5.2_PCR 5’ AATGATACGGCGACCACCGA 3’; P7.1_PCR 5’ CAAGCAGAAGACGGCATACGAGAT 3’ | |
Смешать компоненты быстрым встряхиванием на вортексе и сбросить капли центрифугированием. Поместить пробирку в термоциклер и провести ПЦР-амплификации с использованием следующих условий циклов:
| Температура, оС | Время, с | Циклы |
| 94 | 120 | 1 |
| 95 | 20 | 5–12** |
| 62 | 20 | |
| 68 | 45 | |
| 68 | 45 | 1 |
| 4 | ∞ | |
| Примечание: ** – количество циклов ПЦР следует выбирать, исходя из количества введенной ДНК. Число циклов должно быть достаточно большим, чтобы обеспечить достаточное количество фрагментов библиотеки для успешного секвенирования, но также должно быть достаточно низким, чтобы избежать появления артефактов вследствие ПЦР и избыточной амплификации (появления высокомолекулярных фрагментов на Биоаналайзере). |
||
Рекомендованное количество циклов ПЦР приведено ниже. Его можно использовать в качестве отправной точки для определения числа циклов ПЦР, наилучшего для приготовления образцов библиотек ДНК.
| Количество исходной ДНК, нг | Число циклов ПЦР, требуемое для выхода библиотеки примерно 250–500 нг |
| ≥ 250 | 5 |
| 200 | 6 |
| 100 | 7 |
| 40–50 | 8–9 |
| 20 | 10 |
| 10 | 11 |
| 5 | 12 |
5. Очистка ПЦР-реакции.
Необходимо дать шарикам AMPure XP прогреться до комнатной температуры не менее 30 мин перед использованием. Для ресуспендирования встряхнуть шарики AMPure XP на вортексе.
Добавить 45 мкл (0,9X) ресуспендированных шариков к ПЦР-реакции. Хорошо перемешать с пипетированием по меньшей мере 10 раз. Манипуляции следует проводить осторожно, чтобы исключить всю жидкость из наконечника во время последнего смешивания. Также можно использоваться смешивание на вортексе в течение 3–5 с. Если после центрифугирования образцы центрифугируются, необходимо остановить центрифугирование до того, как шарики начнут оседать.
Инкубировать образцы на столе не менее 5 мин при комнатной температуре.
Чтобы отделить шарики от супернатанта, планшет/пробирки помещают на соответствующую магнитную подставку. При необходимости образец центрифугируют, чтобы собрать жидкость со стенок лунки пробирки или планшета перед постановкой на магнитную подставку. Через 5 мин (или когда раствор станет прозрачным) удаляют супернатант. Это действие надо проводить осторожно, чтобы не потревожить шарики, содержащие целевую ДНК.
Добавить 200 мкл 80%-го свежеприготовленного этанола в пробирку/планшет, находящиеся в магнитной подставке. Инкубируют при комнатной температуре в течение 30 с, а затем осторожно удаляют и отбрасывают супернатант.
Повторяют эту стадию промывки один раз, чтобы получить в итоге две стадии промывки. Обязательно удаляют всю видимую жидкость после второй промывки. При необходимости, кратковременно центрифугируют пробирку/планшет, помещая обратно на магнит и удаляя следы этанола с помощью автоматического дозатора на 20 мкл. Это действие надо проводить осторожно, чтобы не потревожить шарики, содержащие целевую ДНК.
Высушить шарики на воздухе до 5 мин, пока пробирка/планшет находятся на магнитной подставке с открытой крышкой.
Внимание: важно не пересушивать шарики! Это может привести к снижению выделения ДНК-мишени. Образцы следует элюировать, когда шарики остаются темно-коричневого цвета и выглядят блестящими, но при этом видно, что вся видимая жидкость испарилась. Когда шарики становятся светло-коричневыми, и их поверхность начинает покрываться трещинами, они являются уже пересушенными.
Снимите пробирку/планшет с магнитной подставки. Элюируйте ДНК-мишень с шариков, добавляя 32–33 мкл 0,1X TE. Хорошо перемешайте суспензию пипетированием 10 раз или встряхиванием на вортексе. Инкубируйте суспензию не менее 2 мин при комнатной температуре. При необходимости шарики можно центрифугировать в течение короткого времени, чтобы собрать жидкость со стенок пробирки или лунок планшета, прежде чем поставить их обратно на магнитную подставку.
Поместите пробирку/планшет на магнитную подставку. Через 5 мин (или когда раствор станет прозрачным) перенести 30 мкл в новую ПЦР-пробирку для хранения при минус 20 °C.
Далее необходимо проверить распределение фрагментов библиотеки (например, с использованием Agilent Bioanalyzer High Sensitivity DNA chip).
3.1.4 Измерение концентрации ДНК с помощью PicoGreen dsDNA Assay Kit
В ходе работы не следует использовать для раствора стеклянную посуду.
Подготовка красителя:
Перед началом работы следует дать реагенту Quant-iT™ PicoGreen® нагреться до комнатной температуры, прежде чем открывать пробирку с ним. Для наилучшего результата раствор необходимо использовать в течение нескольких часов после приготовления. Приготовленный раствор с красителем следует защищать от света (поместив его в темное место или накрыв фольгой).
- Проверить корректность установки даты и времени в приборе («Settings» → «Instrument settings» → «Date/Time»). При необходимости установить верные значения.
- В чистую, объемом 15 мл пробирку внести 1005 мкл 20х TE буфера.
- Добавить в пробирку 19095 мкл Н2О, а также 101 мкл красителя.
- Тщательно перемешать.
- Приготовить контрольную ДНК для построения кривой (0 и 100 нг в 5 мкл):
- внести в пробирку на 0,2 мл 8 мкл 1хТЕ буфера;
- добавить 2 мкл контрольной ДНК из набора (100 нг/мкл). Хорошо перемешать пипетированием;
- подготовить две оптически прозрачные пробирки объемом 0,5 мл;
- одну подписать как К1, другую – К2;
- в К1 внести 200 мкл раствора для измерения концентрации;
- в К2 внести 195 мкл раствора для измерения и 5 мкл разведенной контрольной ДНК (из шага b).
- Внести по 195 мкл в оптически прозрачные пробирки объемом 0,5 мл.
- Добавить в каждую пробирку по 5 мкл соответствующей коровьей ДНК.
- В пробирку К1 внести 5 мкл раствора ДНК второго разведения
(38 + 2 мкл). - В пробирку К2 внести 5 мкл раствора ДНК первого разведения (8 + 2 мкл).
- Включить прибор, подключить его к компьютеру.
- Выбрать на первом экране программу PicoGreen.
- Произвести измерения.
3.1.5 Подготовка библиотеки к нанесению в картридже
После объединения образцов измерить концентрацию ДНК.
Произвести пересчет массовой концентрации в молярность: http://molbiol.ru/scripts/01_07.html (программа выдает данные в пикомолях, необходимо пересчитать в наномоли).
Далее необходимо следовать инструкциям Protocol A из «MiSeq System Denature and Dilute Libraries Guide».
3.1.6 Разведение щелочи (NaOH)
К 100 мкл стокового раствора (10М) добавить 900 мкл Н2О.
К отобранным 200 мкл полученного раствора (1N NaOH) добавить
800 мкл Н2О.
В результате получится 1 мл 0,2N NaOH.
3.1.7 Денатурация библиотеки
1. В микроцентрифужной пробирке смешать следующие объемы.
- 4 нM библиотека (5 мкл);
- 0,2N NaOH (5 мкл).
2. Перемешать кратким встряхиванием на вортексе и центрифугировать при 280×g в течение минуты.
3. Инкубировать при комнатной температуре в течение 5 мин.
4. В пробирку, содержащую денатурированную библиотеку, добавить
990 мкл холодного НТ1-буфера.
5. В результате получается 1 мл денатурированной библиотеки с концентрацией 20 пМ.
6. Нанести в картридж 600 мкл.
3.2 Перечень однонуклеотидных полиморфизмов (SNP), входящих в использованную панель TruSeq Bovine Parentage Sequencing Panel
Все исследуемые SNP можно разделить на Блок 1 и Блок 2.
Блок 1. Родство и достоверность происхождения (200 SNP)
Исходя из рекомендаций Международного общества по изучению генетики животных (ISAG), по каждому животному определяются важные индивидуальные мутации. Достоверность происхождения контролируется по 200 SNP.
Блок 2. Генетически наследуемые заболевания (53 SNP)
В таблице 5 приведен подробный перечень SNP, которые были использованы нами для характеристики исследуемых экспериментальных стад.
Таблица 5 – Исследованные мутации
| № п/п | Идентификатор SNP | Значение замены | Сочетание аллелей | ||
| позитивно | нейтрально (носительство) |
негативно | |||
| 1 | 2 | 3 | 4 | 5 | 6 |
| 201 | _SNPchr19_27956790 | Мутация в белке, продукте гена SHBG, связывающем и транспортирующем стероидные половые гормоны. Ассоциирована с потерей плодовитости, связанным с гаплотипом MH1 и эмбриональной смертностью | C/C | C/T | T/T |
| 202 | _SNPchr26_36951321 | Мышечная аномалия переднего пояса у КРС – аутосомно-рецессивное расстройство | C/C | C/T | T/T |
| 203 | _SNPchr6_89169686 | Синдром легочной гипоплазии и анасарка (РНА) – редкая врожденная аномалия у крупного рогатого скота, которая характеризуется водянкой плода, включая сильный подкожный отек (anasarca) и неразвитые или слабо сформированные легкие (легочная гипоплазия) | A/A | A/G | G/G |
| Продолжение таблицы 5 | |||||
| 1 | 2 | 3 | 4 | 5 | 6 |
| 204 | FBN1_SNPchr10_62054844 | Синдром Марфана (MFS) – наследственное аутосомно-доминантное заболевание крупного рогатого скота, связанное с мутацией в гене FBN1 10 хромосомы Вos taurus | G/G | A/G
A/A |
|
| 205 | FBN1_SNPchr10_62141461 | Синдром Марфана (MFS) – наследственное аутосомно-доминантное заболевание. Патологические процессы развиваются в сердечно-сосудистой и костно-мышечной системах, глазах, коже и кожных покровах | G/G | A/G
A/A |
|
| 206 | _SNPchr7_17645428 | Мутация, вызывающая нейропатическую гидроцефалию (NH). Большинство случаев NH приводят к ранним абортам. NH телят, которые рождаются намного меньшим весом для регистрационного возраста | A/A | A/G | G/G |
| 207 | _SNPchr8_108833985 | Кератоконъюктивит – быстро распространяющаяся, доброкачественно протекающая болезнь животных различных видов, вызываемая риккетсиоподобными микроорганизмами. С каждым аллелем ‘G’ животное уменьшает свой риск заражения на 8–13 %. Таким образом, животное, которое является гомозиготным G/G аллелей, имеет риск инфицирования меньше на 16– 26 % по сравнению с гомозиготой A/A |
G/G | A/G | A/A |
| 209 | ASS1_SNPchr11_100802781 | Цитруллинемия – это аутосомно-рецессивное расстройство, которое для животных является летальным в раннем постнатальном периоде | C/C | C/T | T/T |
| 210 | CWC15_SNPchr15_15707169 | Гаплотип джерсейского скота (JH1). Аутосомно-рецессивная смертельная мутация гомолога CWC15 (гомолог сплайсосома-ассоциированного белка CWC15) на хромосоме 15 | C/C | C/T | T/T |
| 211 | LRP4_SNPchr15_77667135 | Синдактилия (мулья нога). Во многих странах у многих пород крупного рогатого скота сообщалось о синдактилии. Однако большая часть документации связана с ее появлением у голштинской породы, разводимой в Америке | C/C | C/T | T/T |
| Продолжение таблицы 5 | |||||
| 1 | 2 | 3 | 4 | 5 | 6 |
| 212 | LRP4_SNPchr15_77675440+LRP4_SNPchr15_77675516+LRP4_SNPchr15_77675517 | Множественные мутации в гене белка 4, связанным с рецептором LDL. Синдактилия (мулья нога). Пораженный крупный рогатый скот может иметь 1–4 объединенных копыта, проявляет разную степень хромоты, имеет высокую ступенчатую походку и медленно ходит | G/G | G/A | A/A |
| 213 | LRP4_SNPchr15_77682052 | Мутация обнаружена в гене белка 4, связанным с рецептором LDL. Сопровождается синдактилией (нога мула) | G/G | G/A | A/A |
| 214 | LRP4_SNPchr15_77686731 | Мутация обнаружена в гене белка 4, связанным с рецептором LDL. Сопровождается синдактилией (нога мула) | G/G | G/A | A/A |
| 217 | GART_SNPchr1_1277227 | Гаплотип голштинского скота (HH4). Повышенная вероятность эмбриональной и постэмбриональной смертности | A/A | A/C | C/C |
| 218 | ITGB2_SNPchr1_145114963 | Синдром врожденного иммунодефицита (Bovine Leukocyte Adgesion Defiency, BLAD) | T/T | T/C | C/C |
| 219 | CLCN7_SNPchr25_1137017+CLCN7_SNPchr25_1137019 | Остеопетроз (OS) – заболевание костей, приводящее к тяжелым нарушениям в организме животного. Дефект активности остеокластов приводит к образованию чрезмерно хрупких костей. Больные телята рождаются, как правило, раньше срока мертвыми. Заболевание имеет аутосомно-рецессивный тип наследования | G/G | G/T | T/T |
| 220 | COL7A1_SNPchr22_51873390 | Буллезный эпидермолиз (БЭ) – наследственное буллезное заболевание животных, характеризующееся крайней хрупкостью кожи и слизистых оболочек при механическом воздействии | C/C | C/T | T/T |
| 221 | FECH_SNPchr24_57298883 | Врожденная эритропоэтическая протопорфирия – генетическое заболевание, приводящее к тяжелой форме фотосенсибилизации (повышенной чувствительности организма, чаще кожи к свету). Кроме боязни света у животных наблюдается повреждение кожи и слизистых оболочек, иногда случаются припадки и судороги |
G/G | G/T | T/T |
| Продолжение таблицы 5 | |||||
| 1 | 2 | 3 | 4 | 5 | 6 |
| 223 | FANCI_SNPchr21_21184873 | Синдром брахиспина (BS) – редкий рецессивный генетический дефект молочного голштинского крупного рогатого скота. Риск эмбриональной смертности | T/T | T/C | C/C |
| 225 | ATP2A1_SNPchr25_26197204 | Врожденная мышечная дистония 1-го и 2-го типов – наследственное заболевание с аутосомно-рецессивным типом наследования, широко распространенное в бельгийской голубой породе. У всех телят с CMD наблюдаются генерализованные мышечные контрактуры (непроизвольное обширное напряжение мышц), нарушение глотания, высокая степень утомляемости, длительные мышечные спазмы | C/C | C/A | A/A |
| 226 | ATP2A1_SNPchr25_26197429 | Врожденная мышечная дистония 1-го и 2-го типов | C/C | C/A | A/A |
| 227 | ATP2A1_SNPchr25_26198573 | Врожденная мышечная дистония 1-го и 2-го типов | C/C | C/T | T/T |
| 229 | SLC6A5_SNPchr29_24610561 | Врожденная мышечная дистония 1-го и 2-го типов | A/A | A/G | G/G |
| 230 | SLC37A2_SNPchr29_28879810 | Аборт из-за гаплотипа MH2 у Bos taurus |
T/T | T/C | C/C |
| 231 | PYGM_SNPchr29_43611783 | Гликогенное заболевание (V). Наследственное заболевание с аутосомно-рецессивным типом наследования | G/G | G/A | A/A |
| 235 | KRT5_SNPchr5_27545478 | Врожденный буллезный эпидермолиз – наследственное заболевание, которое характеризуется преимущественно хрупкостью и другими дефектами кожи и слизистых оболочек. Может наблюдаться деформация и отслоение копыт. Пораженные телята чаще всего погибают в возрасте 1–4 нед. Буллезный эпидермолиз распространен в голштинской породе КРС |
G/G | G/A | A/A |
| 236 | APAF1_SNPchr5_63150400 | Гаплотип (HH1), связанный с эмбриональной смертностью. Гаплотип HH1, связанный с эмбриональной смертностью на разных сроках стельности, картирован на 5-й хромосоме | C/C | C/T | T/T |
| Продолжение таблицы 5 | |||||
| 1 | 2 | 3 | 4 | 5 | 6 |
| 237 | HEPHL1_SNPchr29_695072 | Гипотрихоз. У крупного рогатого скота с мутацией в гене HEPHL1 отсутствуют волосяной покров при рождении частично или по всему телу: часто грудь, шея и ноги. Пострадавшие животные более чувствительны к окружающей среде, кожным инфекциям, вредителям, солнечным ожогам, холодовому стрессу | T/T | T/A | A/A |
| 238 | IFNGR2_SNPchr1_1390292 | Мутация в гене IFNGR2 ассоциирована с туберкулезом. Дефекты в IFNGR2 являются причиной менделевской восприимчивости к микобактериальному заболеванию (MSMD), также известному как семейная диссеминированная атипичная микобактериальная инфекция. MSMD представляет собой генетически гетерогенное заболевание с аутосомно-рецессивным, аутосомно-доминантным или X-связанным наследованием | G/G | G/A | A/A |
| 239 | KDM2B_SNPchr17_56010031 | ДисплазияPaunch Calf Syndrome. Телята обычно мертворожденны, имеют аномальное развитие нескольких органов, лицевые деформации и расширенный желудок, наполненный жидкости. У некоторых пораженных телят также есть выступающий язык и расщепленное нёбо | G/G | G/A | A/A |
| 240 | KDSR_SNPchr24_62138835 | Спинальная мышечная атрофия (SMA). Относится к нейродегенеративным заболеваниям. Причиной болезни является мутация в гене KDSR, кодирующем 3-кетодигидросфингозинредуктазу, которая катализирует важнейшие процессы в метаболизме гликосфинголипидов | C/C | C/T | T/T |
| 241 | KRT74_SNPchr5_27490704 | Мутация в гене кератин 74. Аутосомно-доминантная патология волосяного покрова животного (ADWH), также связана с мутацией гена KRT74 – это редкое заболевание, характеризующееся плотно скрученными волосами | C/C | C/T | T/T |
| Продолжение таблицы 5 | |||||
| 1 | 2 | 3 | 4 | 5 | 6 |
| 245 | LYST_SNPchr28_8508619 | Синдром Чедиак-Хигаси (CHS) представляет собой аутосомно-рецессивное расстройство, которое поражает несколько видов, включая мышей, людей и крупный рогатый скот. Вызывает нарушение работы белка регулятора транспорта лизосом, что приводит к изменениям в размере, структуре и функции лизосом | T/T | T/C | C/C |
| 246 | MFN2_SNPchr16_42562057 | Aксонопатия. Телята часто имеют широкую постановку ног и начинают терять контроль над задними лапами в возрасте 1 мес, в дальнейшем они не могут стоять. Хотя болезнь не смертельна, пораженные телята, как правило, подвергаются эвтаназии в возрасте 10 мес |
C/C | C/T | T/T |
| 247 | MITF_SNPchr22_31746502 | Синдром белого симментала (GWFS) – аутосомально-доминантное генетическое заболевание, которое характеризуется гипопигментацией, гетерохромией радужки, дефектами оболочки глаз и двусторонней потерей слуха | C/C | C/A
A/A |
|
| 248 | MAN2B1_SNPchr7_13956640 | Маннозидоз – это рецессивное генетическое заболевание, связанное с нарушением работы лизосомальной альфа-маннозидазы – низкоспецифичной экзогликозидазы, основная функция которой – деградация гликопротеинов | G/G | G/A | A/A |
| 249 | MAN2B1_SNPchr7_13957949 | Маннозидоз. AM_961 (обнаружена в линиях Angus и Murray Grey): |
T/T | T/C | C/C |
| 250 | MITF_SNPchr22_31769189 | Синдром белого симментала (GWFS) – аутосомально-доминантное генетическое заболевание, которое характеризуется гипопигментацией, гетерохромией радужки, дефектами оболочки глаз и двусторонней потерей слуха | T/T | T/A
A/A |
|
| 251 | OPA3_SNPchr18_53546443 | Кардиомиопатии – тяжелые дегенеративные заболевания миокарда, которые приводят к сердечной недостаточности. В течение последних трех десятилетий дилатационная кардиомиопатия быков (BDCMP) получила широкое распространение в голштинской и симментальской породах крупного рогатого скота | G/G | G/A | A/A |
| Продолжение таблицы 5 | |||||
| 1 | 2 | 3 | 4 | 5 | 6 |
| 252 | GLRA1_SNPchr7_65080197 | Врожденный миоклонус – это наследственное аутосомно-рецессивное заболевание, которое ранее было известно, как нейроаксиальный отек. У животных с ICM наблюдается аномальная реакция на различные тактильные, зрительные и слуховые раздражители в виде гиперестезии и миоклонических судорог скелетных мышц | G/G | G/T | T/T |
| 253 | PLD4_SNPchr21_71001232 | Врожденный дефицит цинка (ZDL).Это заболевание характеризуется нарушением функции иммунной системы, замедлением роста и кожными изменениями, вследствие нарушения абсорбции цинка в кишечнике | G/G | G/A | A/A |
| 254 | RASGRP2_SNPchr29_43599204 | Тромбопатия – генетическое заболевание, связанное с нарушением свертываемости крови, происходящим в результате блокировки высвобождения АДФ из тромбоцитов. У больных животных количество тромбоцитов находится в норме, но их функция нарушена, что приводит к нарушению свертываемости крoви | A/A | A/G | G/G |
| 255 | RNF11_SNPchr3_95601697 | Карликовость. Мутация в гене RNF11 (ассоциирована с нарушением регуляции белкового комплекса А20, что приводит к различным патологиям роста и развития) | A/A | A/G | G/G |
| 256 | SLC35A3_SNPchr3_43412427 | Комплексный порок позвоночника (CVM) – сложный тератологический синдром телят голштинской породы, заключающийся, прежде всего, в выкидышах, преждевременных родах, мертворождении или смерти телят в первые дни жизни, а значит увеличивающий смертность молодняка и воспроизводительные способности скота. Самым ярким проявлением этой аномалии, о чем говорит уже само ее название, являются многочисленные уродства скелета. Cмертность на стадии эмбриона | G/G | G/T | T/T |
| Продолжение таблицы 5 | |||||
| 1 | 2 | 3 | 4 | 5 | 6 |
| 257 | SLC39A4_SNPchr14_1723331 | Акродерматит энтеропатический. Заболевание, обусловленное нарушением всасываемости в кишечнике за счет дефицита цинка и отсутствия фермента олигопептидазы. Мутации в гене SLC39A4 приводят к дефициту протеина, ответственного за всасывание цинка, что ведет к грубому изменению метаболизма цинка | G/G | G/A | A/A |
| 258 | IARS_SNPchr8_85341291 | Перинатальный синдром слабого теленка. Соответствует понятию «физиологическая незрелость» новорожденных (иногда называют «синдром слабых телят»). У таких животных снижен мышечный тонус, тормозится реализация позы стояния (они не могут подняться на ноги в течение первых двух часов после рождения), сосательный рефлекс появляется с запозданием и слабо выражен, отмечают безучастное отношение к окружающему, сгорбленную позу | C/C | C/G | G/G |
| 259 | SMC2_SNPchr8_95410507 | Голштинский гаплотип НН3. Оказывает влияние на процент успешных осеменений (с наступлением стельности) и ассоциирован с эмбриональной и ранней постэмбриональной смертностью | T/T | T/C | C/C |
| 260 | SPAST_SNPchr11_14760164 | Спинальная демиелинизация (SDM). Нейродегенаритивное заболевание характеризуется патологическими изменениями миелиновой оболочки спинного мозга. Болезнь связана с нарушением работы белка спастина, которое приводит к потере АТФазной активности этого белка | G/G | G/A | A/A |
| 261 | TG_SNPchr14_9487659 | Врожденный зоб щитовидной железы (CG) – это аутосомно-рецессивное наследственное заболевание, вызванное мутацией в гене TG, который кодирует белок тиреоглобулин. CG является первым генетическим заболеванием среди всех доместицированных видов животных, для которых была описана молекулярно-генетическая природа | G/G | G/A | A/A |
| Продолжение таблицы 5 | |||||
| 1 | 2 | 3 | 4 | 5 | 6 |
| 262 | TYRP1_SNPchr8_31711945 | Глазокожный альбинизм | G/G | G/A | A/A |
| 263 | UBE3B_SNPchr17_65921497 | Мутация в гене UBE3B – убиквитин-белковой лигазы E3B | C/C | C/T | T/T |
| 264 | UMPS_SNPchr1_69756880 | Дефицит уридинмонофосфатсинтетазы (Deficiency of Uridine-5-Monophosphate Synthase, DUMPS).
DUMPS – это наследственное летальное аутосомно-рецессивное заболевание в голштинской популяции, приводящее к ранней эмбриональной смертности на стадии имплантации эмбриона в матку. Это связано с нарушением синтеза уридин-монофосфата в организме, который является одним из ключевых элементов при синтезе de novo пиримидиновых нуклеотидных оснований. Смертность эмбриона наступает примерно на 40 день после оплодотворения |
C/C | C/T | T/T |
| 265 | F8_SNPchrX_38971744 | Гемофилия А (FVIIID; OMIA 000437-9913) представляет собой нарушение свертываемости крови, описанное как у человека, так и у многих видов домашних животных (лошадей, овец), в том числе у крупного рогатого скота. Заболевание связано с дефицитом фактора VIII свертывания крови – плазменного гликопротеина, ген которого расположен на Х хромосоме |
A/A | A/T | T/T |
В первом столбце указан порядковый номер SNP на панели. Во втором столбце приведено наименование однонуклеотидного полиморфизма (SNP) (например, для SNP с порядковым номером 2 – это «ARS-BFGL-BAC-19454» (один из 100 дополнительно введенных в целях более точного определения родства полиморфизм), а для SNP с порядковым номером 265 – это «F8_SNPchrX_38971744» (мутация, приводящая к развитию гемофилии А, дефицит фактора VIII свертываемости крови). В столбце 3 описано влияние замены на физиологическое состояние индивида, т. е. дана информация о генетически наследуемом признаке или заболевании, с которым ассоциирован данный однонуклеотидный полиморфизм (так для SNP с порядковым номером 265 – «F8_SNPchrX_38971744», в таблице это гемофилия А).
В четвертом столбце указан конкретный нуклеотид аллеля, представленный в качестве референсного (наиболее представленного) для соответствующего SNP в базах данных (так, для SNP № 2 «ARS-BFGL-BAC-19454» – это цитозин – С), а в пятом столбце указаны альтернативные аллели в соответствующей популяции (или возможные, помимо референсного), в случае «ARS-BFGL-BAC-19454» – это тимин.
4 РАЗРАБОТКА ПРОГРАММНОГО ПРОДУКТА
4.1 Основные цели и задачи, поставленные при разработке созданного программного продукта
Прибор для высокопроизводительного секвенирования MiSeq (Illumina, США) за один цикл работы (запуск) с использованием панели TruSeq Bovine Parentage Sequencing Panel Illumina способен обработать до 96 образцов ДНК-материала. В итоге, на выходе прибор генерирует CSV-файл объемом примерно 1,03 Мб, который включает себя 25440 строк данных, в случае обработки
96 образцов генетического материала – библиотеки полногеномной ДНК КРС. Соответственно, время обработки такого объема информации вручную будет нерационально велико, особенно, если необходимо получить на выходе сразу несколько документов, к примеру, и индивидуальные паспорта, и 2D-диаграмму (ковер) для всего стада.
Данная работа занимает слишком много человеко-часов и является экономически невыгодной, так как для того, чтобы обрабатывать предложенный объем данных в сутки (время берется с учетом максимальной суточной производительности работы прибора в размере 96 образцов), потребуется около 30 специально обученных работников.
Таким образом, в целях рационализации экономических затрат и человеческих ресурсов, возникает необходимость в разработке комплекса программного обеспечения, которое сможет выполнять необходимые аналитические задачи
и автоматизировать документооборот компании.
Что касается основных целей, которые предъявляются будущему программному продукту, то их можно выделить в следующий список:
- мультисистемность;
- высокая скорость работы;
- гибкость настройки;
- удобство для пользователя.
Требование мультисистемности выражается в том, что программный комплекс должен запускаться на электронно-вычислительных машинах с любой современной операционной системой и при условии того, что ЭВМ может не обладать большой вычислительной мощностью. Для этого на перспективу поставлены задачи по переносу пакета ПО в web-интерфейс с целью обеспечить вышеперечисленные требования. Выбор такого подхода обусловлен тем, что для работы с wed-интерфейсом нет необходимости установки на вычислительную машину каких-либо дополнительных программ, пакетов или сред визуализации, так как браузер присутствует в списке базового оборудования в любой современной операционной системе, будь то системы семейства Windows или же ОС, в основе которых лежит Linux-подобная система, к примеру, MacOS.
Высокая скорость работы необходима для того, чтобы не возникало очереди при обработке данных, которая может повлечь простои в работе всей фирмы, и соответственно, негативно отразиться на ее экономической картине.
Гибкость настройки необходима для того, чтобы пользователям не было необходимости изучать структуру программы и саму среду программирования. То есть пользователи, у которых есть доступ к файлам, могут изменять, к примеру, структуру паспорта или ковра, просто меняя исходный Excel файл, при учете согласования этих изменений с программистом, таким образом, им нет необходимости разбираться в структуре программы и каким-либо образом изменять исходный код, что также поможет избежать ненужных задержек при ежедневной эксплуатации программного пакета.
Ну, и наконец, продукт должен быть удобен для рядового пользователя,
с таким условием, чтобы человеку не пришлось специально переобучаться для работы с ним. Это достигается, опять же, за счет введения в будущем web-интерфейса с выверенным UI дизайном, который позволит выполнять необходимые цели уже после получаса инструктажа.
Теперь остановимся на задачах с точки зрения программиста.
Для начала, при разработке программы необходимо учитывать то, что ей необходимо взаимодействовать с пакетами MS Office, а именно с Microsoft Excel, так как шаблоны как для паспорта, так и для ковра создавались именно
в MS Excel и имеют соответствующий формат «.xlsx».
Также, одним из требований было то, что результирующие файлы, получаемые в итоге, также должны быть «.xlsx» и «.pdf», что также означает, что программный код должен быть написан с учетом поддержки MS Excel.
Еще одним требованием является то, что программа не должна слишком перегружать ресурсы вычислительной машины промежуточными данными, так как предполагается ее дальнейшее использование в Web-интерфейсе, где перегрузка хостинга данными будет вести к неминуемому снижению скорости работы программы или даже непредвиденным ошибкам переполнения выделенной памяти. Для этого было принято решение сохранять промежуточные данные также в формате «.xlsx» c последующим удалением их после целевой отработки.
В качестве следующего требования выделялось то, что программному обеспечению предстоит работать с «.csv» форматом, для которого необходимы специфические библиотеки, таким образом, был выбран язык программирования Python 3.6 с библиотекой Pandas, так как данная библиотека оптимально подходит как для работы с «.csv» форматом, так и для обработки данных, получаемых из таких файлов посредством встроенной в нее структуры DataFrame. Данная структура представляет собой особый вид массивов, отличительной чертой которого является его автоматическая нумерация по строкам и озаглавливание по столбцам.
Данные особенности также крайне необходимы, когда дело доходит до занесения результирующих DataFrame-массивов на лист рабочей книги Excel.
Для обеспечения гибкости настройки файлы-шаблоны всегда доступны
к редактированию пользователям, у которых есть на это доступ, так как программа при каждом своем запуске заново инициализирует шаблон, что позволяет изменять результирующие файлы согласно требованиям практически мгновенно при необходимости и без прямого участия программиста.
Для обеспечения удобства использования уже на данном этапе применен вывод некоторой информации о ходе работы программы в консоль, что позволяет пользователю отслеживать прогресс обработки данных, а также пользователь в консоли сам может вводить определенные данные, такие как порода коровы, ее возраст и т. д., что позволяет ему лишний раз не редактировать результирующий файл, и соответственно, экономить время.
4.2 Обоснование выбора средств и достижения целей
Нами был проведен анализ рынка программного обеспечения, распространяемого открыто по свободной лицензии, ограниченной лицензии и иным формам приобретения. Производился поиск программы или программного пакета, которые удовлетворяли бы цели и задачи, выдвигаемые к ним, такие как мультисистемность, высокая скорость работы, гибкость настройки. Также немаловажным являлось то, что софт предполагалось использовать в узкоспециализированной предметной области и, поэтому он должен быть разработан с учетом требований данной отрасли.
В результате анализа рынка было принято решение создать оригинальный (самостоятельно разработанный) комплекс программ, так как адекватные альтернативы попросту отсутствуют в свободном доступе или же не подходят под требования, предъявляемые программам.
Также немаловажным фактором является то, что в команде работают вместе специалисты в области генной инженерии и программирования, что позволяет обеспечить диалог, в результате которого программный продукт пишется
в соответствии с учетом всех требований генной инженерии. Также тесный диалог между программистом и специалистами в области генотипирования обеспечивает более глубокое понимание целей и задач, выдвигаемых программному продукту. Так как программист находится непосредственно внутри группы разработчиков, он может изменять и добавлять функциональность программ в соответствии с требованиями, избегая взаимодействия между предприятием-заказчиком и предприятием-исполнителем, тем самым сокращая время исполнения поставленных задач.
На данный момент в лаборатории имеются две программы.
4.2.1 Разработка программ Just Append Cows и Covers
Первая программа имеет рабочее название «Just Append Cows» и находится в полностью рабочем состоянии, ее актуальная версия 2.0. Данный программный продукт отвечает за автоматизированное создание паспортов на каждое конкретное животное в стаде.
Вторая программа имеет рабочее название «Covers» и также находится
в полностью рабочем состоянии, ее актуальная версия 1.1. Данный программный продукт отвечает за анализ генетических показателей каждой отдельной особи в стаде и сведения полученной информации в общую наглядную таблицу, цветную 2D-диаграмму, так называемый «ковер».
Для разработки комплекта программного обеспечения была выбрана среда Python актуальной версии 3.6.
Python – современный язык программирования, имеющий возможность работать на всех актуальных операционных системах для персональных компьютеров. Язык программирования Python находится в стадии разработки чуть более 20 лет. На сегодняшний день параллельно активно используется две версии языка – более старая версия 2 и современная версия (третья). Версия 2 более не развивается, но до сих пор еще используется, поскольку очень много программного обеспечения и библиотек разработано именно для этой версии.
Между версиями есть существенная несовместимость, в том числе в синтаксисе команд ввода-вывода (программа на языке Python 2-й версии может не работать в 3-й версии и наоборот), но в целом они очень похожи. В случае
с разработкой описанных выше продуктов было принято решение использовать именно третью версию, она является актуальной, и соответственно, будет обновляться и поддерживаться разработчиком, а также только с ней работают актуальные версии необходимых в разработке библиотек.
Python – современный универсальный интерпретируемый язык программирования. Особенностями его являются кроссплатформенность, современность, наличие обширной встроенной библиотеки и множества сторонних библиотек.
Одной из ключевых особенностей выбора данного языка являлась его гибкость. С одной стороны, Python является объектно-ориентированным языком. Вся мощь объектно-ориентированного подхода доступна при разработке на Python. С другой стороны, язык Python не вынуждает программиста всегда использовать ООП.
Ключевой особенностью, из-за которой был выбран именно этот язык, является то, что код выполняется (интерпретируется) из исходного текста, без предварительного перевода в машинный код. Код, написанный на компилируемых языках, типа C++, сначала переводятся в машинный код.
Принято считать, что интерпретируемые языки программирования работают медленнее, чем компилируемые – из-за того, что трансляция осуществляется не сразу. Но отладка и написание кода происходит быстрее, потому что не нужно ожидать, пока компилятор закончит работать.
Python работает как интерпретируемый, его код перед выполнением компилируется. Многие его компоненты работают на полной скорости компьютера, поскольку написаны они на языке С.
Данная особенность крайне важна, так как планируется дальнейший перевод программного комплекса на web-платформу, где интерпретируемость является необходимым условием.
В качестве дистрибутива было принято решение об использовании пакета Anaconda.
Anaconda – дистрибутив языка программирования Python, в который входит набор популярных свободных библиотек, позволяющих, в том числе, производит работу с данными. Основная особенность Anaconda – поставка единым согласованным комплектом наиболее необходимых библиотек и модулей (таких как NumPy, SciPy, Pandas и др.) с разрешением возникающих зависимостей и конфликтов, которые могут возникать при одиночной установке. По состоянию на 2019 г. Anaconda содержит более 1,5 тыс. модулей.
Основной особенностью дистрибутива является оригинальный менеджер разрешения зависимостей conda с графическим интерфейсом Anaconda Navigator, которые позволяет отказаться от стандартных менеджеров пакетов для Python, таких, как pip, к примеру. Дистрибутив скачивается единожды,
и вся последующая конфигурация, в том числе установка дополнительных модулей, возможна без подключения к сети Интернет. Кроме того, обеспечивается возможность ведения нескольких изолированных сред с раздельным разрешением версионных зависимостей в каждой.
В качестве среды разработки была выбрана IDE Spyder, так как она имеет ряд преимуществ и удобна для проектирования и написания программного кода.
Spyder (ранее Pydee) – свободная среда разработки для Python. Она входит в стандартный пакет Anaconda, является кроссплатформенной и доступна для Windows, Linux и MacOS. Название Spyder расшифровывается как Scientific PYthon Development EnviRonment, что в переводе означает «научная среда разработки для Python». Данная среда разработки создавалась для научных расчетов, и в этой сфере она действительно удобна.
Spyder является высокопроизводительной и дружелюбной по отношению
к разработчику интерактивной средой разработки для языка программирования Python с продвинутыми возможностями редактирования, интерактивного тестирования, отладки и интроспекции.
Также Spyder представляет из себя часть модуля spyderlib для Python, являющегося совокупностью мощных виджетов на PyQt4, таких как редактор кода, консоль Python (при необходимости доступная для встраивания в приложения), редактор списков/кортежей и массивов Pandas.
При разработке пакета программного обеспечения, используются следующие библиотеки: Pandas, Openpyxl, os, Datetame.
Выбор данного перечня библиотек обусловлен задачами, которые предъявляются к программам. Перечисленные библиотеки, с точки зрения разработчика, наиболее оптимально подходят для их выполнения. Ниже приведена краткая характеристика каждой из них и указаны особенности, которые послужили причиной их выбора.
Библиотека os включает в себя множество функций для работы с операционной системой, причем их поведение, как правило, не зависит от ОС, что позволяет сделать программу универсальной и переносимой. В данном случае библиотека os отвечает за работу с директориями, а конкретно за удаление при необходимости временных файлов, в которых хранятся неупорядоченные данные по каждому конкретному образцу.
Библиотека DateTime представляет собой классы для обработки времени
и даты различными способами. Эта библиотека выполняет задачу получения актуальной даты, которая в дальнейшем интегрируется в документ с целью уменьшить ручную работу, выполняемую пользователем.
Pandas является программной библиотекой на языке Python для обработки и анализа данных. Работа Pandas с данными строится поверх библиотеки NumPy, являющейся инструментом более низкого уровня. Предоставляет специальные структуры данных, такие как DataFrame, и операции для манипулирования ими. Корректное использование библиотеки Pandas в купе с другими использованными библиотеками обеспечивается пакетом Anaconda.
Основной областью применения данной библиотеки является обеспечение работы в рамках языка программирования Python в целях сбора (очистки), анализа и моделирования данных, без необходимости перехода на более ориентированные для данных целей языки, такие как R и Octave.
В данной библиотеке одной из ключевых особенностей является использование объектов DataFrame, необходимых для манипулирования индексированными массивами двумерных данных. В концепции рассматриваемой программы объекты DataFrame играют ведущую роль, в связи с этим использование библиотеки Pandas является необходимым.
Также данная библиотека позволяет реализовать удобную и мало затратную с точки зрения ресурсов обработку и редактирование DataFrame-объектов, что позволяет быстро и качественно интегрировать данные объекты на Excel-листы.
Не стоит забывать, что данная библиотека также помогает преобразовывать CSV-данные в DataFrame массивы, и обеспечивает дальнейшую корректную работу с ними.
Библиотека Openpyxl предназначена для того, чтобы обеспечивать возможность чтения и редактирования файлов Excel-форматов, таких как «.xlsx», «.xlsm», «.xltx» и др.
Целью обеих программ является создание результирующих документов
в формате «.xlsx» по заранее созданному шаблону, таким образом, для выполнения этих целей была выбрана именно библиотека Openpyxl, так как она наиболее точно подходила для реализации поставленной задачи.
Данная библиотека отвечает за загрузку в память программы файла-шаблона и последующее заполнение шаблона необходимыми данными, чтобы на выходе получать необходимые результирующие документы, будь то генетический паспорт или ковер (2D-диаграмма) для всего стада.
4.2.2 Организация работы программных продуктов. Блок-схемы программ Just Append Cows и Covers
4.2.2.1 Блок-схемы программы Just Append Cows
На рисунках 8–10 представлена полная блок-схема программы, которая реализует обработку данных для создания паспортов для каждого определенного животного. Данная блок схема включает в себя описание вспомогательных
и главной функций.
Рассмотрим первую вспомогательную функцию report. Данная функция необходима для корректного заполнения результирующего документа для каждой конкретной особи. На вход функции поступают три DataFrame (на схеме df1, df2, df3), которые являются массивами данных блоков SNP для каждой конкретной особи. Так, df1 отвечает за блок 200 SNP «Родство и достоверность происхождения», df2 – за блок 53 SNP «Генетически наследуемые заболевания», df3 – за блок 12 SNP «Экономически значимые признаки». Данные DataFrame уже являются упорядоченными и отсортированными согласно спецификации, так как формирование паспорта происходит на финальной стадии работы программы. Также на вход в функцию поступает переменная idat, которая является идентификатором очередного образца, для которого формируется документ.
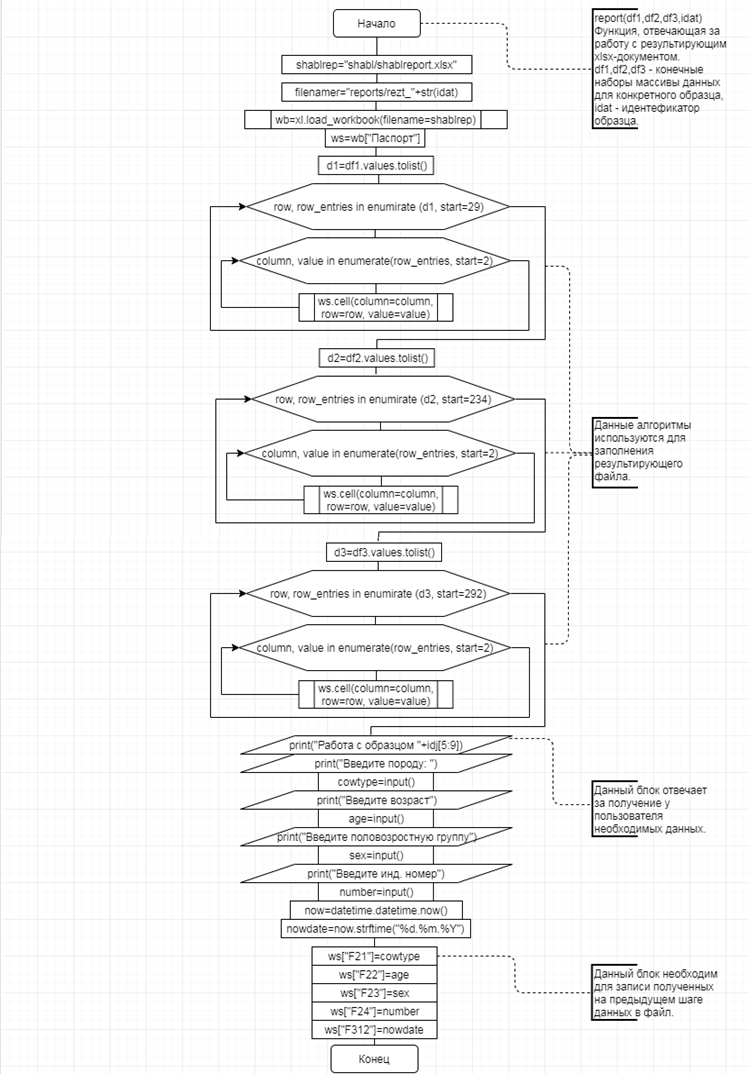
Рисунок 8 – Блок-схема реализации первой вспомогательной функции report
При вызове функции report происходит инициализация функции, далее
в переменную shablrep заносится путь, по которому программа определяет директорию, в которой хранится шаблон паспорта, после этого в переменной filenamer описывается соответствующая папка, в которой хранятся все паспорта для стада и формируется имя файла для каждой конкретной особи. После этого происходит инициализация переменной wb с помощью библиотеки openpyxl. Данная переменная является интерпретированной для работы в Python Excel-книгой, а в переменной ws указывается на каком именно листе будет производится работа с этой самой книгой. Далее идут три цикла занесения данных на лист. Они подобны друг другу, опишем первый, в остальных меняются лишь некоторые переменные и их значения.
Для начала переменной d1 присваиваются значения df1-массива с указанием того, что эти значения будут занесены на лист. Далее следует цикл, суть которого заключается в том, что мы берем все строки со стартовой (в данном случае это двадцать девятая строка, согласно шаблона), далее мы заносим в эту строку в каждый столбец со стартового (в данном случае это второй столбец, согласно шаблона) и заносим поочередно в каждую ячейку строки значения, где номер прохода соответствует номеру строки в переменной d1. Так, к примеру, в двадцать девятую строку заносится первая строка массива, в тридцатую вторая и так до окончания массива.
Как уже говорилось выше, два следующих цикла идентичны, меняются только значения (d2 присваиваются значения df2, стартовая строка у второго блока 234-ая, d3 присваиваются значения df3, стартовая строка у третьего блока 292-ая).
Далее следует блок, который отвечает за запрашивание у пользователя через консоль необходимых для окончательного формирования паспорта данных, таких как порода, возраст, половозрастная группа, идентификационный номер и занесения их в паспорт. Для начала в консоли пишется номер очередного образца, для которого происходит создание паспорта, а потом последовательно запрашиваются при помощи консоли необходимые данные. Также в данном блоке определяется дата формирования паспорта, она автоматически получается из системного времени с помощью библиотеки datetime. Полученные данные заносятся в соответствующие переменные: cowtype, sex, age, number, nowdate.
После этого следует блок записи этих данных на Excel-лист в тех местах, где это необходимо согласно требованиям.
На этом функция report завершает свою работу.
Рассмотрим работу вспомогательной функции sort, которая отвечает за сортировку данных каждого конкретного образца по блокам, при этом в каждом блоке происходит распределение по порядку, который необходим для занесения этих данных в результирующий документ.
Функция получает на вход идентификатор очередного образца (inpdata). Далее происходит инициализация переменных shabl1, shabl2, shabl3, в которые заносятся пути расположения шаблонов, согласно которым происходит сортировка данных. Далее происходит чтение временного файла, в котором находятся данные для очередного образца, представляющие собой массив из 265 строк в произвольном порядке, так как прибор для секвенирования заранее не упорядочивает их. Данные из временного файла заносятся в переменную dfdata. После этого происходит чтение файлов шаблонов – dfshabl1, dfshabl2, dfshabl3 для первого, второго и третьего блоков соответственно.
После получения всех необходимых данных, в переменные a, b, d заносятся размеры блоков (200, 53 и 12 строк соответственно), а в переменную b заносится размер всего массива данных (265 строк). После этого можно приступать к циклам сортировки.
Все три алгоритма сортировки работают аналогичным способом, поэтому будет описан принцип работы алгоритма для сортировки и заполнения массива первого шаблона, в остальных циклах меняются лишь некоторые переменные.
Внешний цикл алгоритма отвечает за последовательный построчный перебор DataFrame-массива первого шаблона. Перебирается i от 0 до 264, при этом на каждом проходе переменной ind присваивается значение, соответствующее i-ому наименованию маркера первого шаблона. Внутри этого цикла происходит построчный перебор исходного массива данных, притом при каждом проходе переменной prov присваивается значение, соответствующее j-ому наименованию маркера исходного набора данных. Далее проверяется, равна ли переменная ind переменной prov, и если да, то в массив шаблона с очередным наименованием маркера вносятся значения соответствующего маркера из исходного массива данных. Цикл устроен таким образом, что на каждом очередном проходе для строки шаблона подыскивается соответствующая строка в исходном массиве, таким образом, на выходе получается DF-массив dfshabl1, который уже упорядочен, так как за основу его взят массив из шаблона, и заполнен соответствующими данными из исходного массива.
Для остальных двух циклов меняются лишь величина внешних проходов согласно размерности шаблонных массивов (вместо а подставляется c и d)
и сами шаблонные массивы (вместо dfshabl1 подставляется dfshabl2 и dfshabl3).
Далее происходит вызов функции report, суть и цели которой описаны выше, в которую отправляются полученные заполненные массивы dfshabl1, dfshabl2, dfshabl3 и идентификатор очередного образца inpdata.
Далее рассмотрим главную функцию программы. Блок-схема этой функции представлена на рисунках 9а и 9б. При запуске программы происходит инициализация данной функции, другими словами, данная функция срабатывает при запуске программы, и в ней уже происходит вызов вспомогательных функций, рассмотренных выше. В программе, отвечающей за генерацию паспортов, используются следующие библиотеки: Pandas, Openpyxl, os, Datetame.
Pandas – программная библиотека на языке Python для обработки и анализа данных. Работа Pandas с данными строится поверх библиотеки NumPy, являющейся инструментом более низкого уровня. Предоставляет специальные структуры данных, такие как DataFrame, и операции для манипулирования ими.
Основная область применения – обеспечение работы в рамках языка программирования Python в целях сбора (очистки), анализа и моделирования данных, без необходимости перехода на более ориентированные для данных целей языки, такие, как R и Octave.
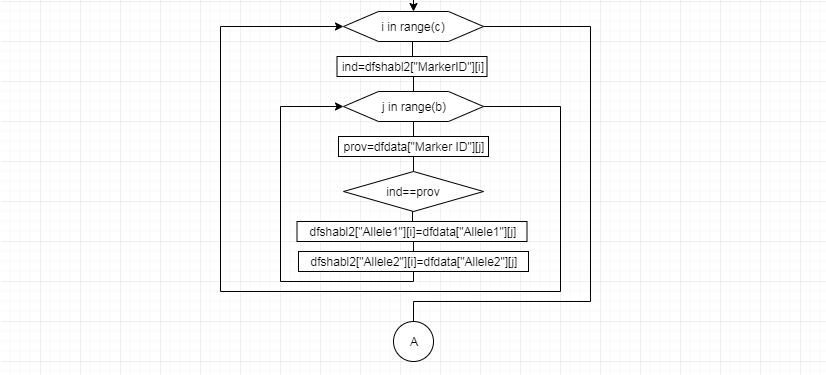
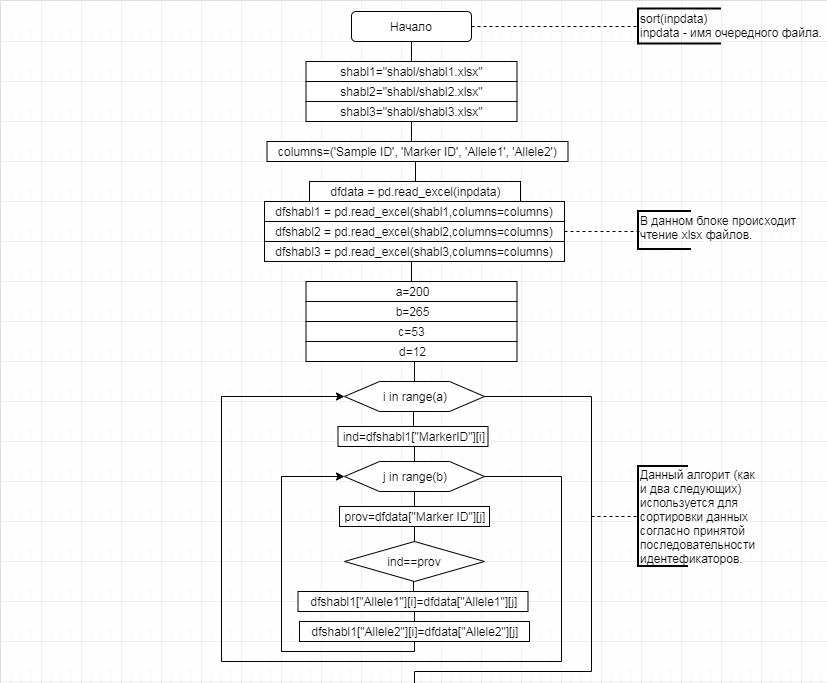
Рисунок 9а – Блок-схема главной функции программы Just Append Cows
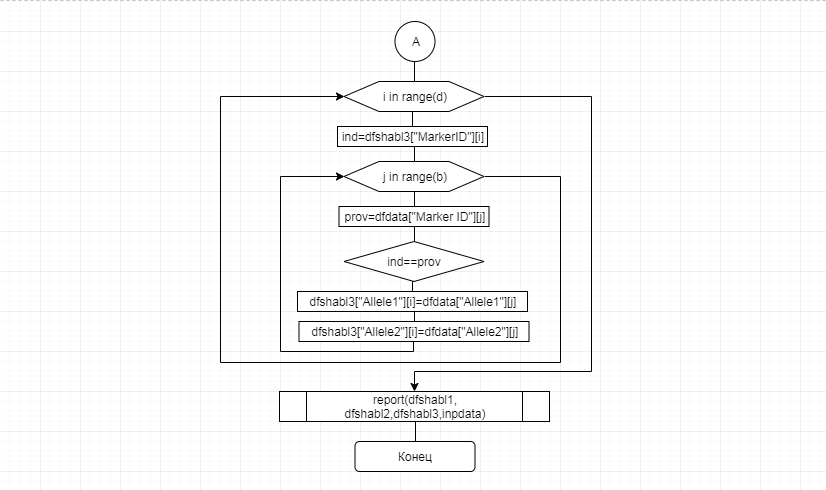
Рисунок 9б (продолжение 9а) – Блок-схема главной функции программы
Just Append Cows
В данной библиотеке одной из ключевых особенностей является использование объектов DataFrame, необходимых для манипулирования индексированными массивами двумерных данных. В концепции рассматриваемой программы объекты DataFrame играют ведущую роль, соответственно, по этой причине использование библиотеки Pandas является необходимым.
Также данная библиотека позволяет реализовать удобную и мало затратную с точки зрения ресурсов работу с DataFrame объектами, что позволяет быстро и качественно интегрировать данные объекты на Excel-листы.
Не стоит забывать, что данная библиотека также помогает преобразовывать CSV-данные в DataFrame массивы и обеспечивает дальнейшую корректную работу с ними.
Библиотека Openpyxl необходима для того, чтобы читать и редактировать файлы Excel-форматов, таких как «.xlsx», «.xlsm», «.xltx» и др.
Целью программы является создание результирующих документов в формате «.xlsx», причем по определенному шаблону, таким образом, для выполнения этих целей была выбрана именно библиотека Openpyxl, так как она наиболее точно подходила под выдвигаемые требования.
Данная библиотека отвечает за загрузку в память программы файла-шаблона и последующее его заполнение необходимыми данными, чтобы на выходе получить заполненный паспорт для каждого образца в соответствии с требованиями.
Библиотека os включает в себя множество функций для работы с операционной системой, причем их поведение, как правило, не зависит от ОС, что позволяет сделать программу универсальной и переносимой. В данной программе библиотека os отвечает за работу с директориями, а конкретно за удаление при необходимости временных файлов, в которых хранятся неупорядоченные данные по каждому конкретному образцу.
Библиотека DateTime представляет собой классы для обработки времени
и даты различными способами. Нас в данной библиотеке интересует именно дата, а точнее получение актуальной даты для занесения ее в определенную ячейку результирующего документа.
После инициализации главной функции и подключения необходимых библиотек, пользователю предлагается в консоли ввести имя «.csv» файла, который он получил в результате работы прибора для секвенирования. Важно заметить, что для обеспечения корректности работы программы имя файла вводится без расширения и сам файл должен находится в корневой папке с исполняемым «.py» файлом.
Далее в переменной on происходит преобразование введенного пользователем имени файла в необходимы для дальнейшей работы формат, а после этого происходит открытие файла и запись данных из него в переменную df. После этого мы имеем DataFrame массив, хранимый в переменной df, с которым уже
и будет производится дальнейшая обработка.
В переменной filename инициализируется имя первого временного файла,
в который будут занесены данные о первом образце. Далее инициализируется список filenames, который включает в себя информацию обо всех временных файлах, сгенерированных программой. В данный список заносится имя первого файла, хранимое в переменной filename.
В переменной last хранится индекс последней строки, или, другими словами, строковая размерность массива df. Далее инициализируется переменная listzn, в которую заносится пустой объект DataFrame. Данный объект будет использоваться для накопления данных о каждом очередном образце, с целью последующего занесения этих данных в соответствующий временный файл. Вначале в listzn заносится значение первой строки из переменной df. Этот шаг необходим для корректной работы цикла в дальнейшем. Далее создается переменная t, которая также необходима для корректной работы цикла.
После этого в цикле, который поочередно отрабатывает все строки массива df, происходит сортировка данных по образцам и сохранение их во временных файлах. Алгоритм сортировки в цикле построен по принципу сравнения с предыдущим (именно поэтому были манипуляции с listzn и создание переменной t). Если очередная строка массива df равна предыдущей, то эта строка заносится в массив listzn в строку с номером t, где t является счетчиком. Если же очередная строка не совпадает с предыдущей, это означает, что получена вся информация по данному образцу. При этом массив listzn сохраняется во временный файл с необходимым именем, после чего массив обнуляется и на место его первой строки заносится не совпавшая строка, таким образом, происходит формирование массива, содержащего информацию о следующем образце. Также происходит присвоение нового значения переменной filename, так как это уже будет новый временный файл с данными о следующем образце. Новое значение filename добавляется в список filenames. После завершения работы цикла записывается последний временный файл с соответствующим именем.
Далее происходит преобразование каждого временного файла с помощью функции sort, в которую поочередно передаются все файлы, которые сгенерировала программа. Имена данных файлов располагаются в списке filenames.
В конце пользователю предлагается выбрать, необходимо ли удалять временные файлы. В случае, если нет необходимости, программа завершает работу, а ином случае, программа удаляет все временные файлы из списка filenames с помощью функции remove из библиотеки os (рисунок 10).
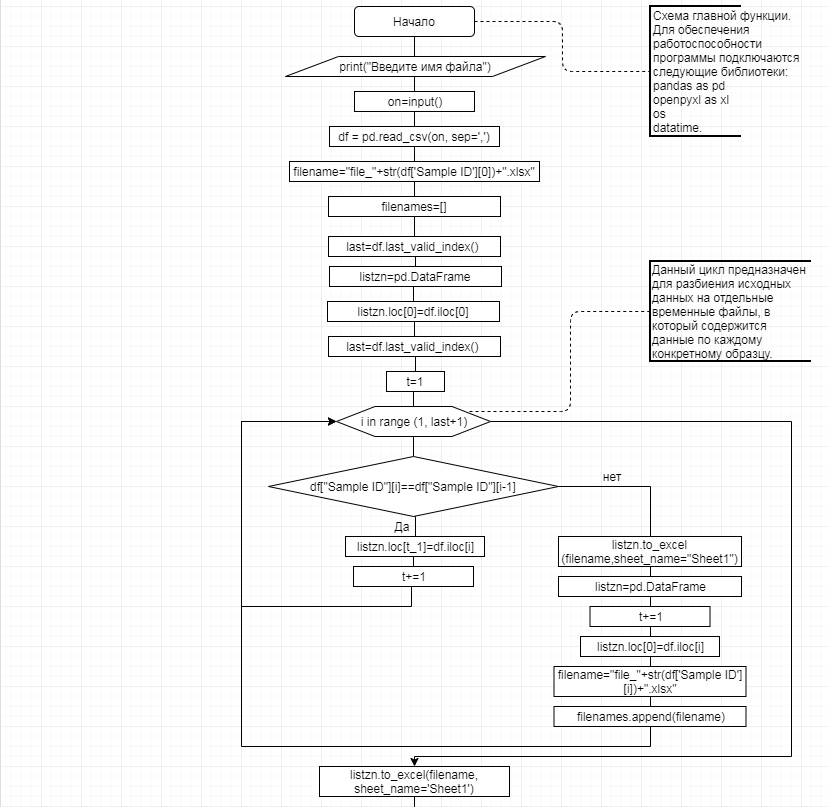
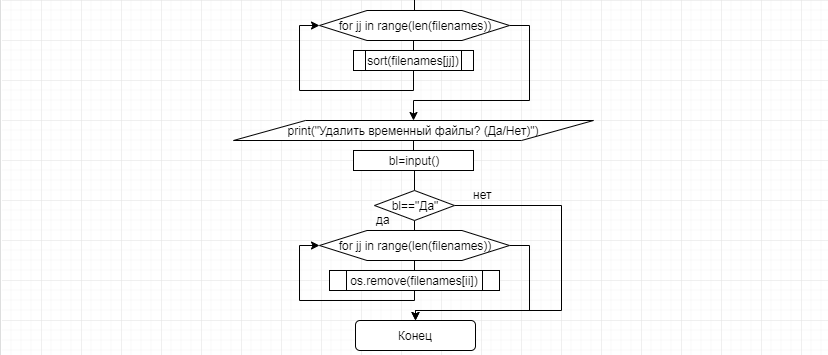
Рисунок 10 – Блок-схема функций завершающих генерацию генетического
паспорта программой Just Append Cows
4.2.2.2. Блок-схемы программы Covers для генерации цветной 2D-диаграммы («ковра»)
На рисунках 11–13 представлена полная блок-схема программы, которая реализует анализ данных для генерации 2D-диаграммы («ковра») для экспериментальных групп животных. Данная блок схема включает в себя описание вспомогательных и главной функций.
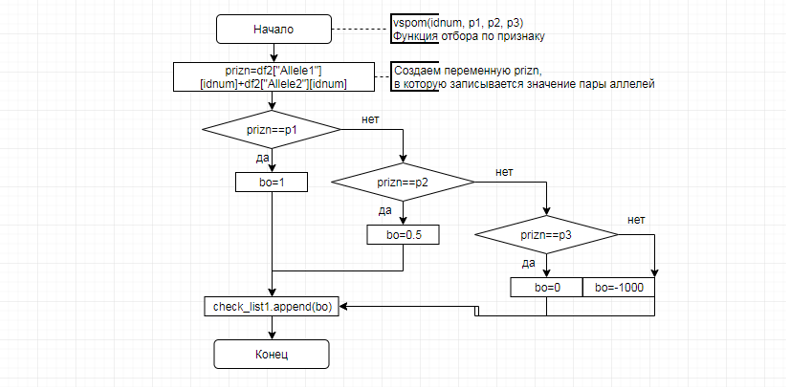
Рисунок 11 – Блок-схема реализации вспомогательной функции vspom1
Рассмотрим вспомогательную функцию vspom. Данная функция предназначения для анализа пары аллелей определенного признака, присвоения им условного коэффициента (веса) и сохранения информации о результате в список. При вызове функции в нее передается информация о номере строки, в которой располагается необходимый для анализа признак. Номер строки обозначен в функции vspom в качестве переменной idnum. Также в функцию передаются сочетания аллелей для анализа, где p1 – это оптимальное сочетание, p2 – серединное сочетание, p3 – негативное сочетание. Стоит отметить, что данная функция предназначена только для работы со вторым блоком «Генетически наследуемые заболевания», данные о котором после сортировки попадают
в DataFrame массив df2.
После инициализации функции и передачи в нее необходимых атрибутов, начинается ее работа. В начале в переменную prizn заносится значение сочетания аллелей Allele1 и Allele2 для рассматриваемого признака. Далее прописывается несколько условий. Если сочетание аллелей равно переменной p1, т. е. совпадает с оптимальным сочетанием, то переменной bo присваивается условный коэффициент оптимальности, который в данном случае равен единице.
Если сочетание аллелей не равно значению переменной p1, то проверяется, равен ли оно значению p2, т. е. совпадает ли с серединным сочетанием. Если обнаружено равенство, то переменной bo присваивается условный коэффициент оптимальности, который в данном случае равен 0,5.
Если сочетание аллелей не равно значению переменной p1 и переменной p2, то проверяется, равен ли оно значению p3, т. е. совпадает ли с негативным сочетанием. Если обнаружено равенство, то переменной bo присваивается условный коэффициент оптимальности, который в данном случае равен нулю.
В противном случае, если сочетание аллелей не равно ни одному из сочетаний, переменной bo присваивается значение «-1000», что означает, что для данного образца (или для всего стада) отсутствует условие для поиска конкретной болезни по данному признаку.
После анализа сочетания аллелей переменная bo заносится в список check_list1, в котором сохраняются все значения переменных bo для очередного образца. Список check_list1 используется для хранения аналитических коэффициентов для второго блока «Генетически наследуемые заболевания».
Рассмотрим вспомогательную функцию vspom2 (рисунок 12). Данная функция предназначена для анализа пары аллелей определенного признака, присвоения им условного коэффициента (веса) и сохранения информации о результате в список. При вызове функции в нее передается информация о номере строки, в которой располагается необходимый для анализа признак. Номер строки обозначен в функции vspom2 в качестве переменной idnum. Также
в функцию передаются сочетания аллелей для анализа, где p1 – это оптимальное сочетание, p2 – серединное сочетание, p3 – негативное сочетание. Стоит отметить, что данная функция предназначена только для работы с третьим блоком «Экономически значимые признаки», данные о котором после сортировки попадают в DataFrame массив df3.
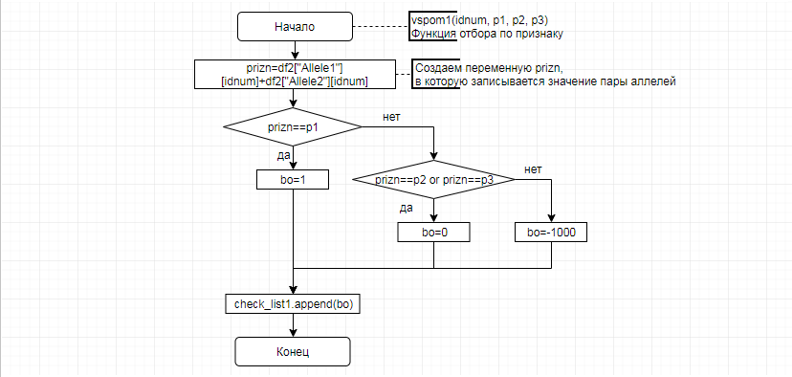
Рисунок 12 – Блок-схема реализации вспомогательной функции vspom2
После инициализации функции и передачи в нее необходимых атрибутов начинается ее работа. В начале в переменную prizn заносится значение сочетание аллелей Allele1 и Allele2 для рассматриваемого признака. Далее прописывается несколько условий. Если сочетание аллелей равно переменной p1, т. е. совпадает с оптимальным сочетанием, то переменной bo присваивается условный коэффициент оптимальности, который в данном случае равен единице.
Если сочетание аллелей не равно значению переменной p1, то проверяется, равен ли оно значению p2, т. е. совпадает ли с серединным сочетанием. Если обнаружено равенство, то переменной bo присваивается условный коэффициент оптимальности, который в данном случае равен 0,5.
Если сочетание аллелей не равно значению переменной p1 и переменной p2, то проверяется, равен ли оно значению p3, т. е. совпадает ли с негативным сочетанием. Если обнаружено равенство, то переменной bo присваивается условный коэффициент оптимальности, который в данном случае равен нулю.
В противном случае, если сочетание аллелей не равно ни одному из сочетаний, переменной bo присваивается значение «-1000», что означает, что для данного образца (или для всего стада) отсутствует условие для поиска конкретной болезни по данному признаку.
После анализа сочетания аллелей переменная bo заносится в список check_list2, в котором сохраняются все значения переменных bo для очередного образца. Список check_list2 используется для хранения аналитических коэффициентов для второго блока «Экономически значимые признаки».
Рассмотрим вспомогательную функцию vspom3 (рисунок 13). Данная функция предназначения для анализа пары аллелей определенного признака, присвоения им условного коэффициента (веса) и сохранения информации о результате в список. При вызове функции, в нее передается информация о номере строки, в которой располагается необходимый для анализа признак. Номер строки обозначен в функции vspom3 в качестве переменной idnum. Также
в функцию передаются сочетания аллелей для анализа, где p1 – это оптимальное сочетание, p2 и p3 – серединное сочетание. Стоит отметить, что данная функция предназначена только для работы с третьим блоком «Экономически значимые признаки», данные о котором после сортировки попадают в DataFrame массив df3.
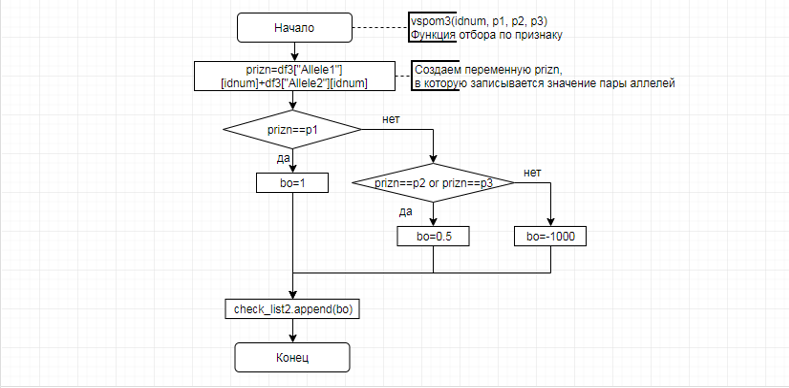
Рисунок 13 – Блок-схема реализации функции vspom3
После инициализации функции и передачи в нее необходимых атрибутов начинается ее работа. В начале в переменную prizn заносится значение сочетание аллелей Allele1 и Allele2 для рассматриваемого признака. Далее прописывается несколько условий. Если сочетание аллелей равно переменной p1, т. е. совпадает с оптимальным сочетанием, то переменной bo присваивается условный коэффициент оптимальности, который в данном случае равен единице.
Если сочетание аллелей не равно значению переменной p1, то проверяется, равен ли оно значению p2 или p3, т. е. совпадает ли с серединным сочетанием. Если обнаружено равенство, то переменной bo присваивается условный коэффициент оптимальности, который в данном случае равен 0,5.
В противном случае, если сочетание аллелей не равно ни одному из сочетаний, переменной bo присваивается значение «-1000», что означает, что для данного образца (или для всего стада) сочетания для анализа по данному признаку.
После анализа сочетания аллелей переменная bo заносится в список check_list2, в котором сохраняются все значения переменных bo для очередного образца. Список check_list2 используется для хранения аналитических коэффициентов для третьего блока «Экономически значимые признаки».
Рассмотрим функцию bol (рисунок 14). Функция предназначена для проведения анализа данных по каждому образцу и занесения полученной информации в массивы dff1 и dff2 для последующей генерации ковра для всего стада.
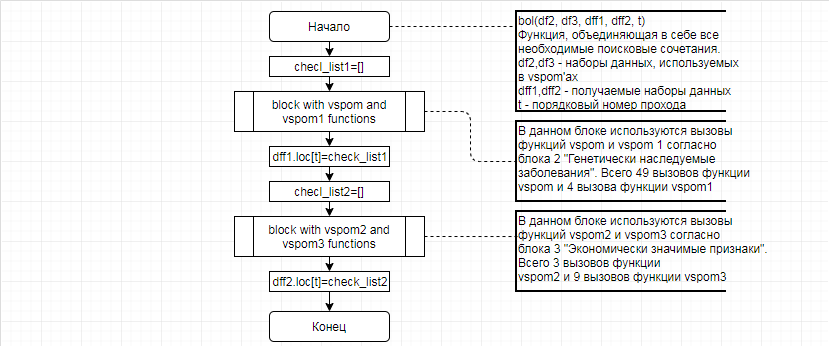
Рисунок 14 – Блок-схема реализации завершения функции bol
Функция получает на вход массивы df2 и df3, в которых содержатся данные о признаках, отсортированные в соответствии с установленным шаблоном, также в функцию передаются массивы dff1 и dff2, которые содержат в себе результаты аналитики в виде коэффициентов для всего стада, причем массив dff1 содержит информацию об анализе блока 53SNP, а dff2 – информацию об анализе блока 12SNP.
Стоит заметить, что признаки находятся в определенной последовательности, так как массив df2 и df3 уже отсортирован в соответствии с установленным шаблоном, поэтому допустимо использовать индексы строк, так как они привязаны к признакам, т. е. вне зависимости от исходного массива, содержащего информацию о стаде, в каждой строке всегда будет один и тот же заранее известный признак.
После инициализации функции и передачи в нее необходимых атрибутов она начинает свою работу. Вначале происходит очистка списка check_list1,
в который будет сохранятся результат анализа для каждой особи. Далее идет блок с вызовами функций vspom и vspom1, которые отвечают за обработку данных второго блока 53SNP. В каждую функцию vspom или vspom1 передается индекс строки, в которой находятся данные о признаке, анализ которого проводится и сочетания аллелей согласно интерпретатору. Всего происходит 49 вызовов функции vspom и четыре вызова функции vspom1, в итоге все 53 признака в блоке анализируются. После анализа список check_list1 добавляется
в итоговый массив dff1 в строку под индексом t, где t – это номер прохода (очередного образца).
Далее происходит очистка списка check_list2, в который будет сохранятся результат анализа для каждой особи. Далее идет блок с вызовами функций vspom2 и vspom3, которые отвечают за обработку данных второго блока 12SNP. В каждую функцию vspom2 или vspom3 передается индекс строки, в которой находятся данные о признаке, анализ которого проводится и сочетания аллелей согласно интерпретатору. Всего происходит три вызова функции vspom2
и девять вызовов функции vspom3, в итоге все 12 признаков в блоке анализируются. После анализа список check_list2 добавляется в итоговый массив dff2
в строку под индексом t, где t – это номер прохода (очередного образца).
После этого функция завершает свою работу.
Рассмотрим работу вспомогательной функции sort (рисунок 15), которая отвечает за сортировку данных каждого конкретного образца по блокам, при этом в каждом блоке происходит распределение по порядку, который необходим для занесения этих данных в результирующий документ.
Функция получает на вход идентификатор очередного образца (inpdata),
а также DataFrame массивы dff1, dff2, dff3, которые содержат информацию
о результатах анализа для всего стада и переменную t, содержащую информацию о номере итерации, происходящей в главной функции. Далее происходит инициализация переменных shabl2, shabl3, в которые заносятся пути расположения шаблонов, согласно которым происходит сортировка данных. Далее совершается чтение временного файла, в котором находятся данные для очередного образца, представляющие собой массив из 265 строк в произвольном порядке, так как прибор для секвенирования заранее не упорядочивает их. Данные из временного файла заносятся в переменную dfdata. После этого происходит чтение файлов шаблонов dfshabl2, dfshabl3 для второго и третьего блоков соответственно.
Далее происходит служебное дублирование переменных dff1, dff2 и t, необходимое для дальнейшей корректной работы программы на этапе вызова функции.
После получения всех необходимых данных, в переменные b, d заносятся размеры блоков (53 и 12 строк соответственно), а в переменную b заносится размер всего массива данных (265 строк). После этого можно приступать
к циклам сортировки.
Оба алгоритма сортировки работают аналогичным способом, поэтому будет описан принцип работы алгоритма для сортировки и заполнения массива второго шаблона, в остальных циклах меняются лишь некоторые переменные.
Внешний цикл алгоритма отвечает за последовательный построчный перебор DataFrame-массива первого шаблона. Перебирается i от 0 до 264, при этом на каждом проходе переменной ind присваивается значение, соответствующее i-ому наименованию маркера первого шаблона.
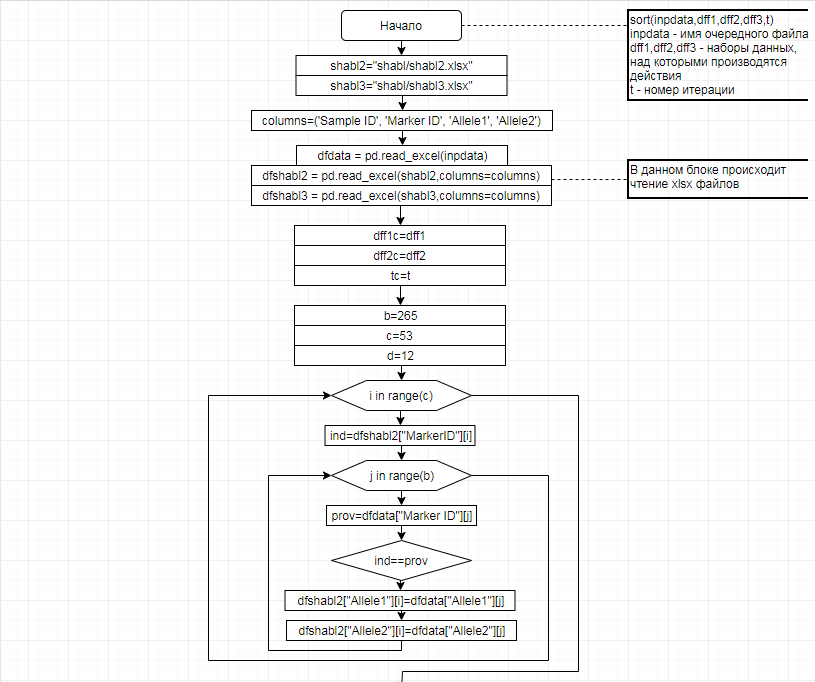
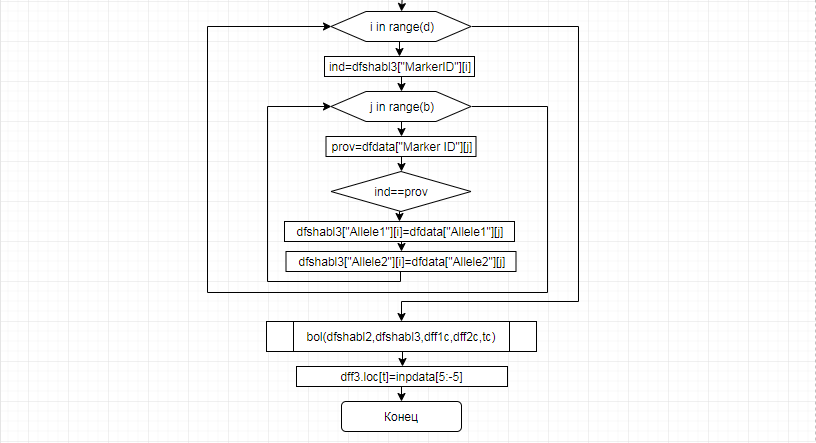
Рисунок 15 – Блок-схема реализации функции sort
Внутри этого цикла происходит построчный перебор исходного массива данных, притом при каждом проходе переменной prov присваивается значение, соответствующее j-ому наименованию маркера исходного набора данных. Далее проверяется, равна ли переменная ind переменной prov, и если да, то в массив шаблона с очередным наименованием маркера вносятся значения соответствующего маркера из исходного массива данных. Цикл устроен таким образом, что на каждом очередном проходе для строки шаблона подыскивается соответствующая строка в исходном массиве, таким образом, на выходе получается
DF-массив dfshabl2, который уже упорядочен, так как за основу его взят массив из шаблона, и заполнен соответствующими данными из исходного массива.
Для второго цикла меняются лишь величина внешних проходов согласно размерности шаблонных массивов (d вместо c) и сам шаблонный массив (вместо dfshabl2 подставляется dfshabl3).
Далее происходит вызов функции bol, суть и цели которой описаны выше,
в которую передаются полученные заполненные массивы dfshabl2, dfshabl3, результирующие массивы dff1 и dff2 и переменную t, содержащую номер итерации.
В массив dff3 заносится имя очередного образца, данный массив далее будет необходим при переносе данных ковра на Excel-лист.
Далее рассмотрим главную функцию программы. Блок-схема этой функции представлена на рисунках 16а–16в. При запуске программы происходит инициализация данной функции, другими словами, данная функция срабатывает при запуске программы, и в ней уже происходит вызов вспомогательных функций, рассмотренных выше. В программе, отвечающей за генерацию паспортов, используются следующие библиотеки: Pandas, Openpyxl, os, Datetame.
Pandas – программная библиотека на языке Python для обработки и анализа данных. Работа Pandas с данными строится поверх библиотеки NumPy, являющейся инструментом более низкого уровня. Предоставляет специальные структуры данных, такие как DataFrame, и операции для манипулирования ими. Основной областью применения библиотеки является обеспечение работы в рамках языка программирования Python в целях сбора (очистки), анализа и моделирования данных, без необходимости перехода на более ориентированные для данных целей языки, такие, как R и Octave.
В данной библиотеке одной из ключевых особенностей является использование объектов DataFrame, необходимых для манипулирования индексированными массивами двумерных данных. В концепции рассматриваемой программы объекты DataFrame играют ведущую роль, соответственно, по этой причине использование библиотеки Pandas является необходимым.
Также данная библиотека позволяет реализовать удобную и малозатратную с точки зрения ресурсов работу с DataFrame-объектами, что позволяет быстро и качественно интегрировать данные объекты на Excel-листы.
Не стоит забывать, что данная библиотека также помогает преобразовывать CSV-данные в DataFrame-массивы и обеспечивает дальнейшую корректную работу с ними.
Библиотека Openpyxl необходима для того, чтобы читать и редактировать файлы Excel-форматов, таких как «.xlsx», «.xlsm», «.xltx» и др.
Целью программы является создание результирующих документов в формате «.xlsx», причем по определенному шаблону, таким образом, для выполнения этих целей была выбрана именно библиотека Openpyxl, так как она наиболее точно подходила под выдвигаемые требования.
Данная библиотека отвечает за загрузку в память программы файла-шаблона и последующее заполнение шаблона необходимыми данными, чтобы на выходе получить заполненный паспорт для каждого образца в соответствии с требованиями.
Библиотека os включает в себя множество функций для работы с операционной системой, причем их поведение, как правило, не зависит от ОС, что позволяет сделать программу универсальной и переносимой. В данной программе библиотека os отвечает за работу с директориями, а конкретно за удаление при необходимости временных файлов, в которых хранятся неупорядоченные данные по каждому конкретному образцу.
После инициализации главной функции и подключения необходимых библиотек, создаются три пустых DataFrame-массива, которые впоследствии будут нести в себе информацию об анализе для стада в целом и использоваться для построения ковра.
После этого пользователю предлагается в консоли ввести имя «.csv» файла, который он получил в результате работы прибора для секвенирования. Важно заметить, что для обеспечения корректности работы программы имя файла вводится без расширения и сам файл должен находится в корневой папке с исполняемым «.py» файлом.
Далее в переменной on происходит преобразование введенного пользователем имени файла в необходимы для дальнейшей работы формат, а после этого происходит открытие файла и запись данных из него в переменную df.
Вследствии чего мы имеем DataFrame массив, хранимый в переменной df, с которым уже и будет производиться дальнейшая обработка.
В переменной filename инициализируется имя первого временного файла,
в который будут занесены данные о первом образце. Далее инициализируется список filenames, который включает в себя информацию обо всех временных файлах, сгенерированных программой. В данный список заносится имя первого файла, хранимое в переменной filename.
В переменной last хранится индекс последней строки, или, другими словами, строковая размерность массива df. Далее инициализируется переменная listzn, в которую заносится пустой объект DataFrame. Данный объект будет использоваться для накопления данных о каждом очередном образце, с целью последующего занесения этих данных в соответствующий временный файл. Вначале в listzn заносится значение первой строки из переменной df. Этот шаг необходим для корректной работы цикла в дальнейшем. Далее создается переменная t, которая также необходима для корректной работы цикла.
После этого в цикле, который поочередно отрабатывает все строки массива df, происходит сортировка данных по образцам и сохранение их во временных файлах. Алгоритм сортировки в цикле построен по принципу сравнения
с предыдущим (именно поэтому были манипуляции с listzn и создание переменной t). Если очередная строка массива df равна предыдущей, то эта строка заносится в массив listzn в строку с номером t_1, где t_1 является счетчиком. Если же очередная строка не совпадает с предыдущей, это означает, что получена вся информация по данному образцу. При этом массив listzn сохраняется во временный файл с необходимым именем, после чего массив обнуляется и на место его первой строки заносится не совпавшая строка, таким образом, происходит формирование массива, содержащего информацию о следующем образце. Также происходит присвоение нового значения переменной filename, так как это уже будет новый временный файл с данными о следующем образце. Новое значение filename добавляется в список filenames. После завершения работы цикла записывается последний временный файл с соответствующим именем.
Далее происходит преобразование каждого временного файла с помощью функции sort, в которую поочередно передаются все файлы, которые сгенерировала программа. Имена данных файлов располагаются в списке filenames.
После разбиения исходного массива df на временные файлы можно приступать к анализу данных. Для обеспечения вывода информации о прогрессе работы программы объявляются переменные prog и rg. В качестве счетчика объявляется переменная nmb.
Далее следует цикл, который обрабатывает поштучно каждый временный файл, вызывает функцию, анализирующую полученную информацию из этого файла, и графически выводит прогресс выполнения.
Работа алгоритма заключается в том, что он извлекает имя очередного временного файла, передает это имя, а также массивы df1, df2, df3 и значение счетчика в функцию sort, принцип работы которой описан выше. После этого
к счетчику прибавляется единица, выводится в консоль состояние прогресса анализа и удаляется очередной, уже отработанный, временный файл.
Следующим шагом работы программы является открытие шаблона ковра посредством функции openpyxl и указание листа, на который будут заносиьтся данные.
После выполнения цикла анализа в программе имеется три итоговых массива df1, df2 и df3, которые далее следует перенести на лист результирующего документа.
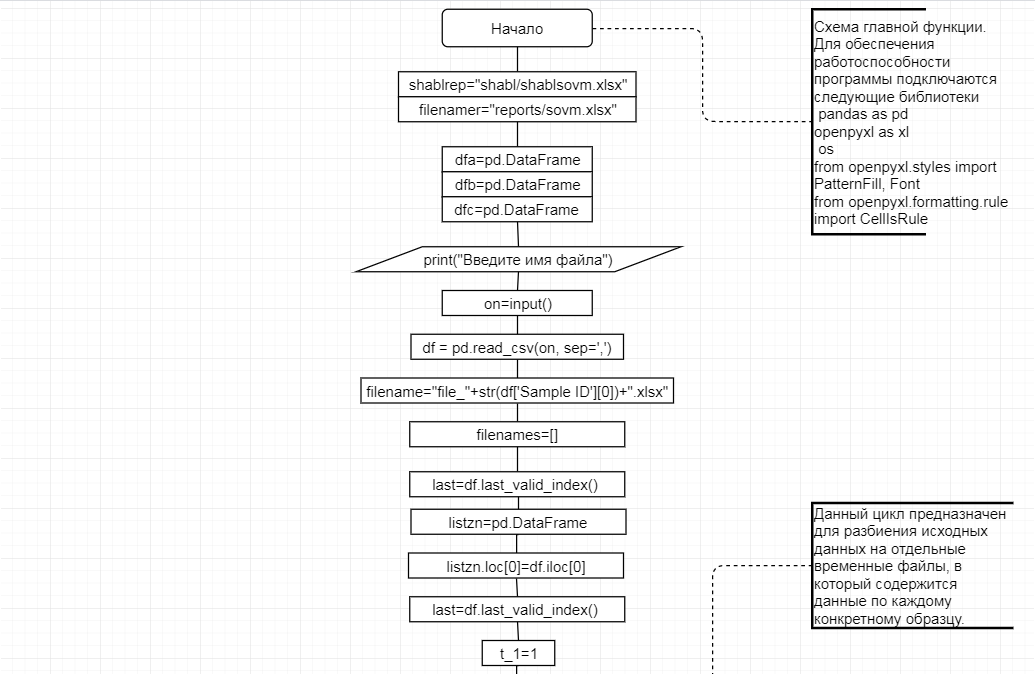
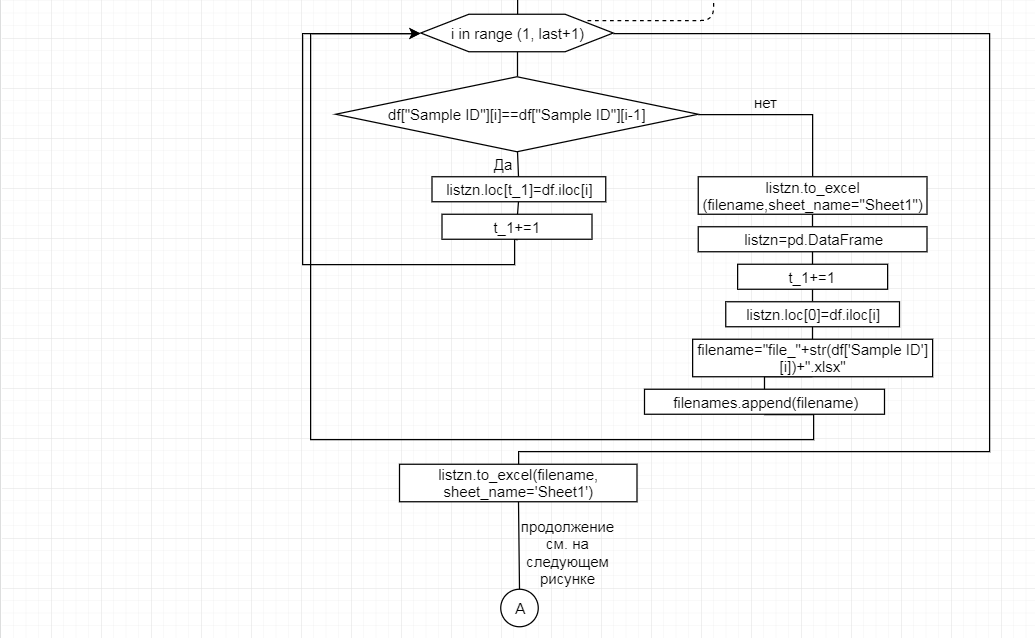
Рисунок 16а – Блок-схема реализации главной функции программы Covers
для генерации цветной 2D-диаграммы («ковра»)
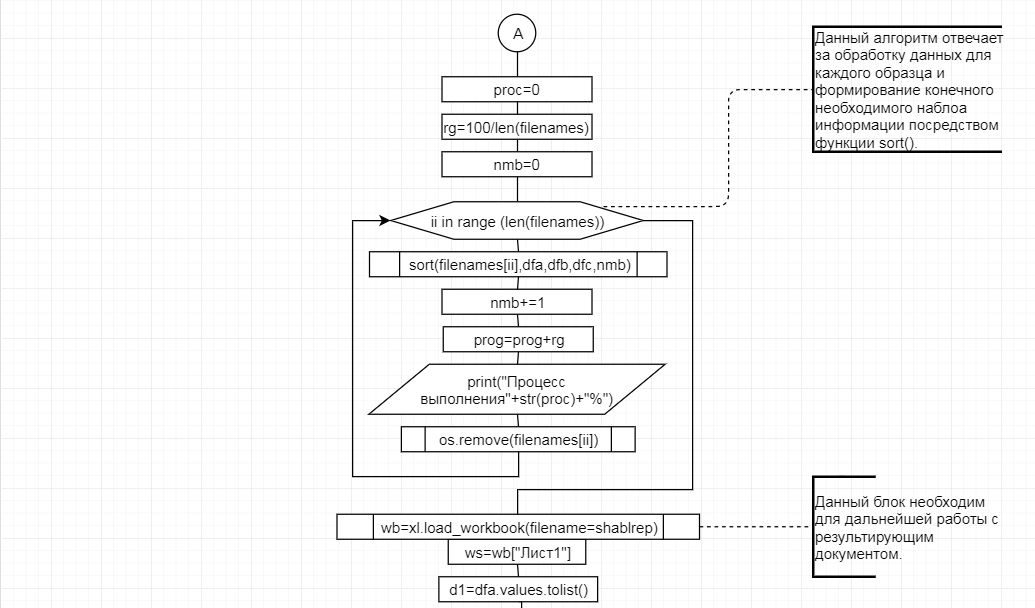
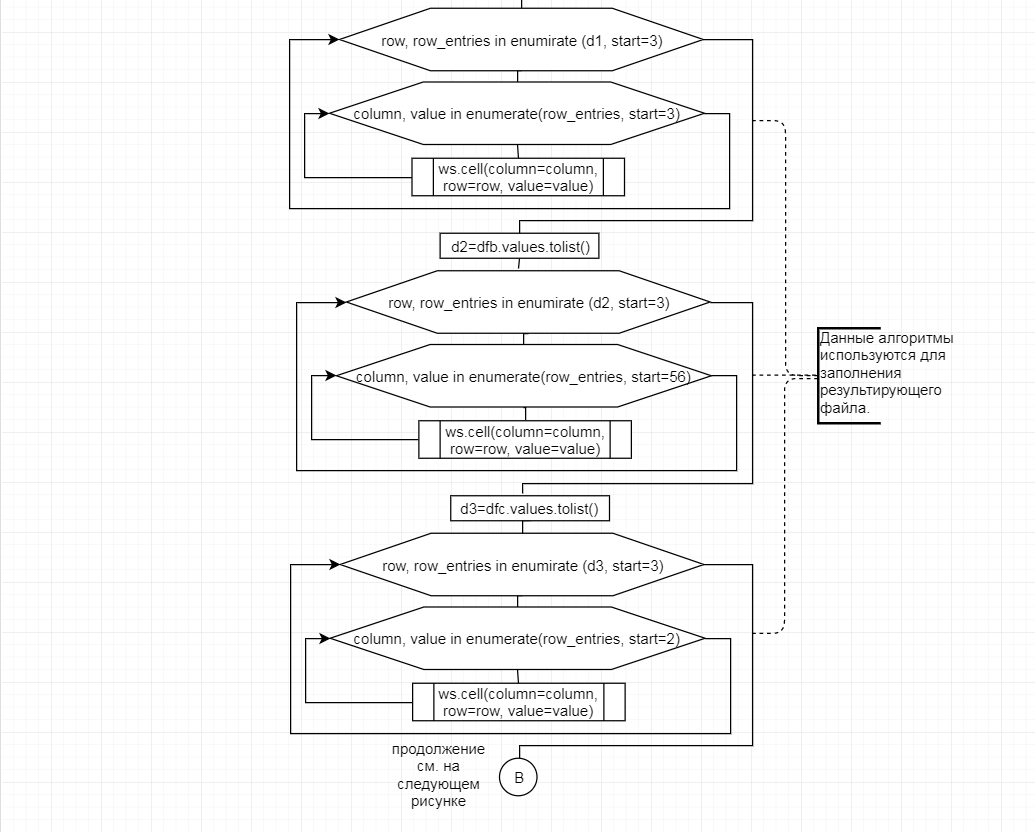
Рисунок 16б (продолжение) – Блок-схема реализации главной функции
программы Covers для генерации цветной 2D-диаграммы («ковра»)
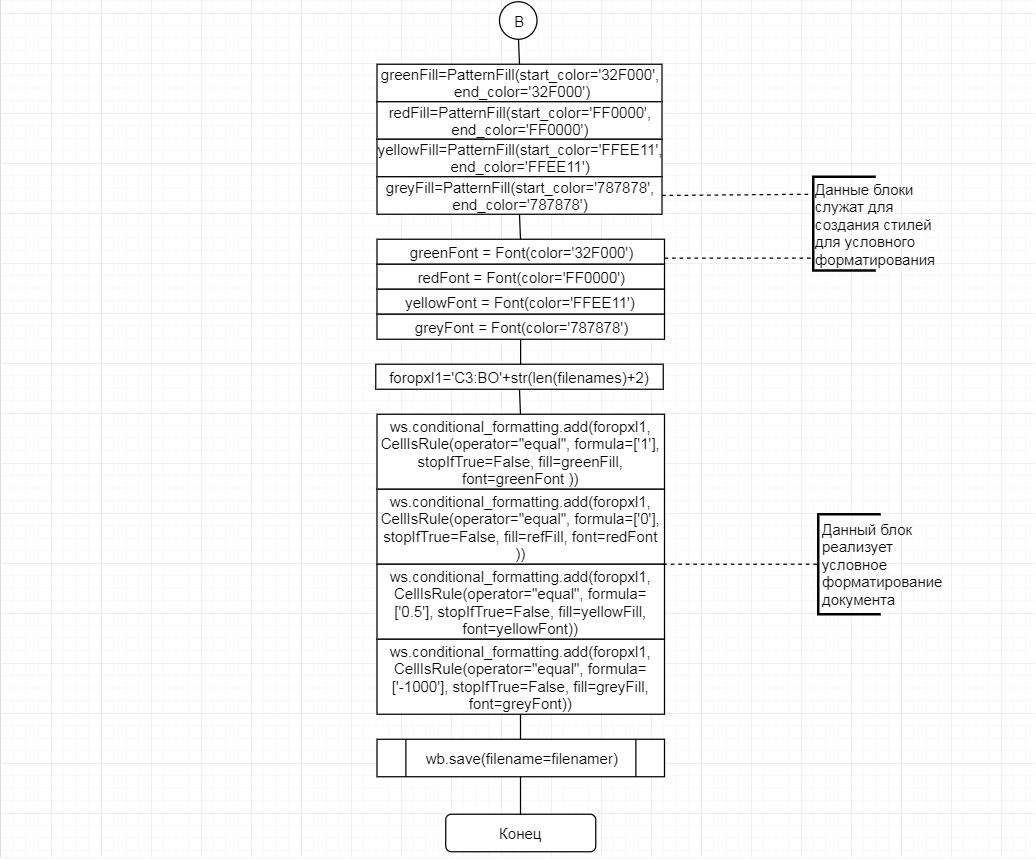
Рисунок 16в (продолжение) – Блок-схема реализации главной функции
программы Covers для генерации цветной 2D-диаграммы («ковра»)
Для этого используются три идентичных цикла, в которых изменяются лишь имена и значения некоторых переменных, опишем первый цикл.
Для начала переменной d1 присваиваются значения df1-масиива с указанием того, что эти значения будут занесены на лист. Далее из переменной d1 построчно извлекается информация, затем строки делятся на ячейки (по четыре ячейки в каждой строке) и значения этих ячеек заносятся на Excel-лист в строку, начиная со стартовой (в данном случае с третьей) и в столбец, начиная со стартового (в данном случае третий).
В остальных циклах меняется соответственно исходные массивы и переменные, в которые они загружаются для занесения (d2 для df2, d3 для df3),
а также стартовая строка для массива d2.
Далее в результирующем документе необходимо выделить строки с соответствующим значением требуемыми цветами согласно техническому заданию. Для этого в библиотеке openpyxl предусмотрены модули, позволяющие внутри программы указать стили и условия для условного форматирования.
Для этого необходимо создать стили заливки и текста, которые будут далее применяться при условном форматировании, за что и отвечают два блока создания стилей для заливки (четыре функции PatternFill) и цвета текста (четыре функции Font), в которых указываются необходимые атрибуты.
Далее указывается область, к которой будет применяться условное форматирование, причем область может быть с любым размером строк согласно исходному количеству образов, что сделано для более гибкой работы программы с разными размерами стада.
После этого применяется условное форматирование для указанной области согласно указанных стилей.
Для ячеек, где сочетание аллелей положительно, т. е. их значение равно единице, используется зеленый цвет заливки (код цвета 32F000).
Для ячеек, где сочетание аллелей отрицательно, т. е. их значение равно нулю, используется красный цвет заливки (код цвета FF0000).
Для ячеек, где сочетание аллелей нейтрально, т. е. их значение равно 0,5, используется желтый цвет заливки (код цвета FFEE11).
Для ячеек, где сочетание аллелей не определенно, используется серый цвет заливки (код цвета 787878).
После выполнения условного форматирования результирующий документ сохраняется, и программа завершает свою работу.
4.2.3 Обеспечение диалога программ Just Append Cows и Covers с пользователем
4.2.3.1 Обеспечение диалога программы Just Append Cows (генерация генетических паспортов)
Для обеспечения диалога с пользователем в программе предусмотрен вывод графической информации. Для начала программа запрашивает у пользователя имя файла, в котором содержится исходные результаты секвенирования для стада. Имя файла должно вводиться без расширения. В данном случае исходным будет файл с именем «group1.csv» (также предусмотрен вариант программы для работы с «.xlsx» файлами). Пример запроса имени файла представлен на рисунке 17.

Рисунок 17 – Пример запроса имени файла
Далее программа с помощью консольного интерфейса будет уведомлять пользователя о состоянии выполнения обработки данных и создания временных файлов для каждого конкретного образца (рисунок 18).

Рисунок 18 – Пример уведомления пользователя о состоянии выполнения
обработки данных и создания временных файлов для каждого
конкретного образца
После разбиения исходного файла по образцам и создания временных файлов программа приступает к сортировке данных и созданию паспортов. Так как для заполнения паспорта необходимы помимо результатов секвенирования такие данные, как порода, возраст, половозрастная группа и идентификационный номер, то пользователю будет предложено ввести требуемые данные последовательно для каждого указанного образца (рисунок 19).

Рисунок 19 – Окно заполнения сопутствующих данных по животному
После генерации результирующих документов по каждому образцу пользователю будет предложено удалить временные файлы. Допустимые варианты ответа: Да, Нет (без учета регистра) (рисунок 20).
![]()
Рисунок 20 – Диалоговое окно выбора решения о удалении
или сохранении временных файлов
В конце работы программа уведомить пользователя о своем состоянии,
а также о месте расположении результирующих документов (рисунок 21).
![]()
Рисунок 21 – Финальное диалоговое окно программы Just Append Cows
4.2.3.2 Обеспечение диалога программы Covers («Ковра»)
(генерация цветной 2D-диаграммы)
Для обеспечения диалога с пользователем в программе предусмотрен вывод графической информации. Для начала программа запрашивает у пользователя имя файла, в котором содержится исходные результаты секвенирования для стада. Имя файла должно вводится без расширения. В данном случае исходным будет файл с именем group1.csv (также предусмотрен вариант программы для работы с .xlsx файлами). Пример запроса имени файла представлен на рисунке 22.

Рисунок 22 – Начальное диалоговое окно программы Covers
Далее программа с помощью консольного интерфейса будет уведомлять пользователя о состоянии выполнения обработки данных и создания временных файлов для каждого конкретного образца (рисунок 23).

Рисунок 23 – Диалоговое окно уведомления о состоянии выполнения
обработки данных и создания временных файлов для каждого
конкретного образца
После этого программа приступает к анализу полученных данных и занесению его результатов в конечный массив. Для удобства использования также предусмотрено уведомление пользователя о ходе выполнения анализа (рисунок 24).
Рисунок 24 – Диалоговое окно уведомления о ходе выполнения анализа
После заполнения результирующих массивов, происходит запись их в результирующий файл, и применяется к нему условное форматирование. После выполнения этих действий программа уведомляет пользователя о завершении своей работы, а также указывает расположение конечного файла (рисунок 25).
![]()
Рисунок 25 – Диалоговое окно уведомления о завершении своей работы,
а также указывает расположение конечного файла
4.2.3.3 Описание исходного Excel-файла для программы Covers
С целью унификации результирующих документов было принято решение разработать общий шаблон для генерации 2D-диаграмм («генетического ковра»), в котором бы содержалась информация о результатах анализа всего стада, а также некоторая справочная информация для обеспечения более удобной дальнейшей работы с документом.
В итоге был реализован шаблонный документ в формате Excel-книги, который включает в себя два листа, на одном из которых отображаются визуализированные цветами данные, а на втором содержится таблица с интерпретацией и кратким описанием каждого признака.
Информация на первом листе представлена в виде таблицы. Все признаки были сгруппированы по блокам и упорядочены. В шапке таблицы присутствуют подписи блоков. Для каждого признака выделен свой столбец, для удобства реализованы ссылки на лист со справочной информацией, т. е. при клике на любой из признаков, пользователю будет предоставлена справочная информация по конкретному признаку, которая включает в себя его порядковый номер, идентификатор SNP, краткое описание, а также сочетания пар аллелей и их расшифровка (является ли сочетание позитивным, нейтральным или негативным).
Каждому образцу соответствует отдельная строка, в которой указывается его номер, а также результаты его анализа.
С целью более удобного и наглядного представления данных к таблице внутри программы применяется условное форматирование, согласно которому ячейки с позитивным прогностическим сочетанием аллелей выделяются зеленым цветом, ячейки с нейтральным прогностическим сочетанием аллелей – желтым цветом, ячейки с негативным прогностическим сочетанием аллелей – красным цветом, ячейки с неопределенным прогностическим сочетанием аллелей – серым цветом.
Пример ковра для 15 образцов можно увидеть на рисунке ниже (рисунок 26).
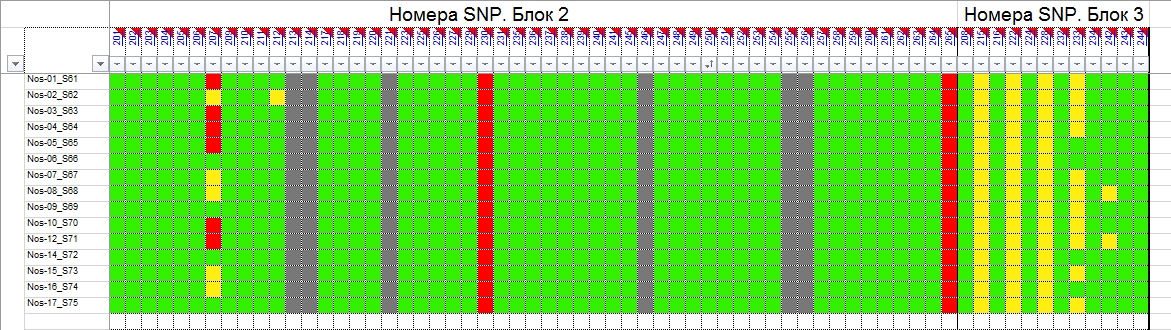
Рисунок 26 – Пример 2D-диаграммы («ковра») для 15 образцов (животных)
ВЫВОДЫ
1. Методом таргетного секвенирования на приборе MiSeq проведено исследование более 300 образцов (цельная кровь и спермодозы) биоптантов КРС с целью определения конкретных значений SNP из списка входящих в коммерчески доступную панель TruSeq Bovine Parentage Sequencing Panel Illumina (США).
2. Создана и проверена на материале первичных экспериментальных данных секвенирования работоспособная программа (программный продукт)
Just Append Cows – для генерации генетического паспорта животного.
3. Создана и проверена на материале первичных экспериментальных данных секвенирования работоспособная программа (программный продукт) Covers («Ковер») – для генерации цветной 2D-диаграммы, описывающей распределение гомозигот и гетерозигот в аллельном спектре произвольной экспериментальной группы животных. Данная программа может быть использована для сопоставления данных представленности аллелей у родительской пары
и подбора оптимального донора для искуственного осеменения.
4. С применением программы Just Append Cows проведена паспортизация экспериментальныхгруппы животных общей численностью более 300.
5. На полученном оригинальном экспериментальном материале продемонстрирована применимость программы Covers для генерации цветной 2D-диаграммы и контроля аллельного спектра групп, а также оптимизации сравнительного подбора спермодоз быка определенного генотипа для конкретной коровы при искусственном осеменении.
СПИСОК ИСПОЛЬЗОВАННОЙ ЛИТЕРАТУРЫ
- Алтухов, Ю. П. Полиморфизм ДНК в популяционной генетике /
Ю. П. Алтухов, Е. А. Салменкова // Генетика. – 2002. – Т. 38. – С. 1173–1195. - Бек, К. Экстремальное программирование: разработка через тестирование / К. Бек. – СПб. : Питер, 2003. – 224 с.
- Вандер Плас, Дж. Python для сложных задач. Наука о данных и машинное обучение / Дж. Вандер Плас. – СПб. : Питер, 2018. – 576 с.
- Веллер, Дж. И. Геномная селекция животных / Дж. И. Веллер. – СПб. : Изд. «Проспект Науки», 2018. – 208 с.
- ГОСТ 19.701-90 ЕСПД (ISO 5807:1985). Схемы алгоритмов программ, данных и систем. – 1992.
- Дасгупта, С. Алгоритмы / С. Дасгупта, Х. Пападимитриу, У. Вазирани; пер. с англ.; под ред. А. Шеня. – М. : МЦНМО, 2014. – 320 с.
- Клименко, И. С. Теория систем и системный анализ / И. С. Клименко. – М. : Российский новый университет, 2014. – 264 c.
- Кофиади, И. А. Методы детекции однонуклеотидных полиморфизмов: аллель-специфичная ПЦР и гибридизация с олигонуклеотидной пробой /
И. А. Кофиади, Д. В. Ребриков // Генетика. – 2006. – Т. 42. – С. 22–32. - Мартин, Р. Чистый код. Создание, анализ и рефакторинг / Р. Мартин; пер. с англ. Е. А. Матвеев. – СПб. : Питер, 2019.
- Мировые тенденции в селекции молочного скота / К. И. Лукъянов, В. А. Солошенко, И. И. Клименюк, Н. С. Юдин // Генетика и разведение животных. – 2015. – Т. 3. – С. 63–69.
- Мюллер, А. Введение в машинное обучение с помощью Python. Руководство для специалистов по работе с данными / А. Мюллер, С. Гвид; пер.
с англ. – СПб. : ООО «Альфа-книга», 2017. - Попов, В. П. Теория и анализ систем / В. П. Попов, И. В. Крайнюченко. – Пятигорск : ПГГТУ, 2012. – 236 с.
- Приемы объектно-ориентированного программирования: паттерны проектирования / Э. Гамма, Р. Хелм, Р. Джонсон, Дж. Влиссидес; пер. с англ.
А. Слинкин. – СПб. : Питер, 2001. - Рамальо, Л. Python. К вершинам мастерства / Л. Рамальо; пер. с англ. – М. : ДМК-Пресс, 2016.
- Рейтц, К. Автостопом по Python / К. Рейтц, Т. Шлюссер; пер. с англ.
Е. Зазноба. – СПб. : Питер, 2017. - Ресурс с документацией по Python [Электронный ресурс]. – URL : https://www.python.org/doc/.
- Ресурс с документацией по библиотеке Openpyxl [Электронный ресурс]. – URL : https://openpyxl.readthedocs.io/en/stable/index.html.
- Ресурс с документацией по библиотеке Pandas [Электронный ресурс]. – URL : https://pandas.pydata.org/pandas-docs/stable/.
- Ресурс с информацией о работе с библиотекой DateTime [Электронный ресурс]. – URL : https://python-scripts.com/datetime-time-python.
- Ресурс с информацией о работе с модулем os [Электронный ресурс]. – URL : https://pythonworld.ru/moduli/modul-os.html.
- Рыбчин, В. Н. Основы генетической инженерии : учеб. пособие для вузов / В. Н. Рыбчин, В. С. Гайцхоки. – Минск : Выш. шк., 1986. – 186 с.
- Саммерфилд, М. Программирование на Python 3. Подробное руководство / М. Саммерфилд; пер. с англ. – М. : Символ-Плюс, 2009. – 608 с.
- Смарагдов, М. Г. Генетическое картирование локусов, ответственных за качественные показатели молока у крупного рогатого скота / М. Г. Смарагдов // Генетика. – 2006. – Т. 42. – С. 5–21.
- Смарагдов, М. Г. Тотальная геномная селекция с помощью SNP как возможный ускоритель традиционной селекции / М. Г. Смарагдов // Генетика. – 2009. – Т. 45. – С. 725–728.
- Статья о интеграции Python и Excel [Электронный ресурс]. – URL : https://habr.com/ru/company/otus/blog/331998/.
- Фаулер, М. Архитектура корпоративных программных приложений / М. Фаулер; пер. с англ. – Вильямс, 2004.
- Фаулер, М. Рефакторинг. Улучшение существующего кода / М. Фаулер, К. Бек; пер. с англ. – М. : Символ-Плюс, 2008.
- Хейдт, М. Изучаем pandas. Высокопроизводительная обработка и анализ данных в Python / М. Хейдт, А. Груздев; пер. с англ. – М. : ДМК-Пресс, 2019.
- Шевелуха, В. С. Сельскохозяйственная биотехнология и биоинженерия / В. С. Шевелуха. – Изд. 4, знач. перераб. и доп. – URSS, 2015. – 704 с.
- Щелкунов, С. Н. Генетическая инженерия / С. Н. Щелкунов. – Новосибирск : Изд-во Новосиб. ун-та, 1997.
- Эрнст, Л. К. Биологические проблемы животноводства в XXI веке / Л. К. Эрнст, Н. А. Зиновьева. – М. : РАСХН, 2008. – 508 с.
- A genome-wide scan for signatures of selection in Azeri and Khuzestani buffalo breeds / M. Mokhber, M. Moradi-Shahrbarak, M. Sadeghi, H. Moradi-Shahrbarak, A. Stella, E. Nicolzzi, J. Rahmaninia, J. L. Williams // BMC Genomics. – 2018. – DOI 10.1186/s12864-018-4759-x.
- Ammendrup, S. Legislative requirements for the identification and traceability of farm animals within the European Union / S. Ammendrup, A. E. Fussel // Revue Scientifique et Technique. – 2001. – Vol. 20. – P. 437–444.
- Animal Passports and Identification / W. Buick // Defra Veterinary Journal. – 2004. – Vol. 15. – P. 20–26.
- Apaf-1 deficiency and neural tube closure defects are found in fog mice /
N. Honarpour, S. L. Gilbert, B. T. Lahn, X. Wang, J. Herz // Proceeding of the National Academy of Sciences USA. – 2001. – Vol. 98. – P. 9683–9687. - Assessing genetic architecture and signatures of selection of dual purpose Gir cattle populations using genomic information / A. Maiorano, D. Lourenco,
S. Tsuruta, A. Ospina, N. Stafuzza, Y. Masuda, A. Filho, J. Cyrillo, R. Curi, J. Silva // PLOS One. – 2018. – Vol. 13. – e0200694. - Bovine exome sequence analysis and targeted SNP genotyping of recessive fertility defects BH1, HH2, and HH3 reveal a putative causative mutation in SMC2 for HH3 / M. C. McClure, D. Bickhart, D. Null, P. Vanraden, L. Xu, G. Wiggans,
G. Liu, S. Schroeder, J. Glasscock, J. Armstrong, J. B. Cole, C. P. Van Tassell,
T. S. Sonstegard // PLOS One. – 2014. – Vol. 9. – P. e92769. - Brookes, A. J. The essence of SNPs / A. J. Brookes // Gene. – 1999. –
Vol. 234. – P. 177–186. - Brookes, A. J. The essence of SNPs / A. J. Brookes // Gene. – 1999. – Vol. 234. – P. 177–186.
- Commission of the European Communities 2000, Regulation (EC) No 1760/2000 of the European Parliament and of the Council of 17 July 2000: Establishing a system for the identification and registration of bovine animals and regarding the labelling of beef and beef products and repealing Council Regulation (EC) No. 820/97. Off J Eur Communities L 204:1-10.
- Comparision of different imputation methods from low- to high-density panels using Chinese Holstein cattle / Z. Weng, Zh. Zhang, Q. Zhang, S. He, X. Ding // Animal. – 2013. – Vol. 7. – P. 729–735.
- Comparison of the effectiveness of microsatellites and SNP panels for genetic identification, traceability and assessment of parentage in an inbred Angus herd / M. E. Fernández [et al.] // Genet and Mol Biol. – 2013. – Vol. 36(2). – P. 185–191.
- Compilation of a panel of informative single nucleotide polymorphisms for bovine identification in the Northern Irish cattle population / A. R. Allen [et al.] // BMC Genet. – 2010. – Vol. 11. – P. 5.
- Complex vertebral malformation in Holstein calves / J. S. Agerholm,
C. Bendixen, O. Andersen, J. Arnbjerg // The Journal of Veterinary Diagnostic Investigation. – 2001. – Vol. 13. – P. 283–289. - Council Regulation (EC) No 178/2002 of the European Parliament and of the Council of 28 January 2002: Laying down the general principles and requirements of food law, establishing the European Food Safety Authority and laying down procedures in matters of food safety. Off J Eur Communities L 31:1-24.
- Cunningham, E. P. Biological Identification Systems: genetic markers /
E. P. Cunningham, C. M. Meghen // Revue Scientifique et Technique. – 2001. –
Vol. 20. – P. 491–499. - Current trends in microsatellite genotyping / E. Guichoux, L. Lagache,
S. Wagner, P. Chaumell, P. Leger, O. Lepais, C. Lepoittevin, T. Malausa, E. Revardel, F. Salin, R. J. Petit // Molecular Ecology Resources. – 2011. – Vol. 11. – P. 591–611. - De Vries, A. Economic value of pregnancy in dairy cattle / A. De Vries // Joural of Dairy Science. – 2006. – Vol. 89. – P. 3876–3885.
- Detection and characterization of SNPs useful for identity control and parentage testing in major European dairy breeds / F. A. O. Werner, G. Durstewitz,
F. A. Habermann, G. Thaller, W. Kramer, S. Kollers, J. Buitkamp, M. Georges,
G. Brem, J. Mosner, R. Fries // Animal Genetics. – 2004. – Vol. 35. – P. 44–49. - Detection of haplotypes associated with prenatal death in dairy cattle and identification of deleterious mutations in GART, SHBG and SLC37A2 S./ Fritz,
A. Capitan, A. Djari, S. C. Rodriguez, A. Barbat, A. Baur, C. Grohs, B. Weiss,
M. Boussaha, D. Esquerré, C. Klopp, D. Rocha, D. Boichard // PLOS One. – 2013. – Vol. 8. – P. e65550. - Ellegren, H. Microsatellites: simple sequences with complex evolution /
H. Ellegren // Nat. Rev. Genet. – 2004. – Vol. 5. – P. 435–445. - Exclusion probabilities of 22 bovine microsatellite markers in fluorescent multiplexes for semi automated parentage testing / D. W. Heyen, J. E. Beever, Y. Da, R. E. Evert, C. Green, S. R. Bates, J. S. Ziegle, H. A. Lewin // Animal Genetics. – 1997. – Vol. 28. – P. 21–27.
- Factors associated with frequency of abortions recorded through Dairy Herd Improvement test plans / H. D. Norman, R. H. Miller, J. R. Wright, J. L. Hutchison, K. M. Olson // Journal of Dairy Science. – 2012. – Vol. 95. – P. 4074–4084.
- Fine mapping of a quantitative trait locus for twinning rate using combined linkage and linkage disequilibrium mapping / T. H. Meuwissen, A. Karlsen, S. Lien, I. Olsaker, M. E. Goddard // Genetics. – 2002. – Vol. 161. – P. 373–379.
- Fosgate, G. T. Ear-tag retention and identification methods for extensively managed water buffalo (Bubalus bubalis) in Trinidad / G. T. Fosgate,
A. A. Adesiyun, D. W. Hird // Preventive Veterinary Medicine. – 2006. – Vol. 73. – P. 287–296. - Genetic Control of Conventional Labelling through the Bovine Meat Production Chain by Single Nucleotide Polymorphisms Using Real Time PCR /
R. Capoferri, G. Bongioni, A. Galli, R. Aleandri // Journal of Food Protection. – 2006. – Vol. 69. – P. 1971–1977. - Genomic evaluation and identification of a haplotype affecting fertility for Ayrshire dairy cattle / T. A. Cooper, G. R. Wiggans, D. J. Null, J. L. Hutchison // Journal of Dairy Science. – 2013. – Vol. 96 (E-Suppl. 1). – P. 74.
- Genomic evaluations with many more genotypes / P. M. VanRaden,
J. R. O’Connell, G. R. Wiggans, K. A. Weigel // Genetics Selection Evolution. – 2011. – Vol. 43. – P. 10. - Genomic Selection in Dairy Cattle: The USDA Experience / G. R. Wiggans, J. B. Cole, S. M. Hubbard, T. S. Sonstegard // Annu. Rev. Anim. Biosci. – 2017. – Vol. 5. – P. 13.1–13.19.
- Glusman, G. Whole-genome haplotyping approaches and genomic medicine / G. Glusman, H. C. Cox, J. C. Roach // Genome Medicine. – 2014. – Vol. 6. – P. 73.
- Grover, A. Development and use of molecular markers: past and present /
A. Grover, P. C. Sharma // Critical Rev. Biotech. – 2016. – Vol. 36. – P. 290–302. - Haplotype tests for recessive disorders that affect fertility and other traits /
J. B. Cole, P. M. VanRaden, D. J. Null, J. L. Hutchison, T. A. Cooper, S. M. Hubbard // Aip Research Report Genomics. – 2015. – P. 9–13. - Harmful recessive effects on fertility detected by absence of homozygous haplotypes / P. M. VanRaden, K. M. Olson, D. J. Null, J. L. Hutchison // Journal of Dairy Science. – 2011. – Vol. 94. – P. 6153–6161.
- Hayes, B. The distribution of the effects of genes affecting QTL in livestock / B. Hayes, M. E. Goddard // Genet. Sel. Evol. – 2001. – Vol. 33. – P. 209–229.
- High-throughput genotyping with single nucleotide polymorphism /
K. Ranade, M. S. Chang, G. T. Ting, D. Pei, C. F. Hsiao, M. Olivier, R. Pesich,
J. Hebert, Y.-D. Chen, V. J. Dzau, D. Curb, R. Olshen, N. Rish, D. R. Cox, D. Botstein // Genome Res. – 2001. – Vol. 11. – P. 1262–1268. - Houston, R. A сomputerised database system for bovine traceability /
R. Houston // Revue Scientifique et Technique. – 2001. – Vol. 20. – P. 652–661. - Hovingh, E. Abortions in dairy cattle: I. Common causes of abortions /
E. Hovingh // Virginia Cooperation Extension Publication. – 2009. – Vol. 404. – P. 288. - IDEA: a large scale project on electronic identification of livestock / O. Ribo, C. Korn, U. Meloni, M. Cropper, P. De Winne, M. Cuypers // Revue Scientifique et Technique. – 2001. – Vol. 20. – P. 426–436.
- Identification of a nonsense mutation in APAF1 that is likely causal for a decrease in reproductive efficiency in Holstein dairy cattle / H. A. Adams,
T. S. Sonstegard, P. M. VanRaden, D. J. Null, C. P. Van Tassell, D. M. Larkin,
H. A. Lewin // Journal of Dairy Science. – 2016. – Vol. 99. – P. 6693–6701. - Idris, I. Python Data Analysis / I. Idris. – Packt Publishing Ltd, 2014.
- Importance of the traceability of animal products in epidemiology / V. Caporale, A. Giovannini, C. Di Francesco, P. Calistri // Revue Scientifique et Technique. – 2001. – Vol. 20. – P. 372–378.
- ISAG cattle core and additional SNP panel 2013. International Society for Animal Genetics (ISAG) Web site. – URL : www.isag.us/committees.asp?autotry=true&ULnotkn=true. Accessed June 13, 2016.
- Kim, S. SNP genotyping: technologies and biomedical applications / S. Kim, A. Misra // Annual Review of Biomedical Engineering. – 2007. – Vol. 9. – P. 289–320.
- Krawczak, M. Informativity assessment for bi-allelic single nucleotide polymorphisms / M. Krawczak // Electrophoresis. – 1999. – Vol. 20. – P. 1676–1681.
- Kruglyak, L. The use of a genetic map of biallelic markers in linakage studies / L. Kruglyak // Nature Genetics. – 1997. – Vol. 17. – P. 21–24.
- Lachance, J. SNP ascertainment bias in population genetic analyses: why
it is important, and how to correct it / J. Lachance, S. A. Tishkoff // BioEssays. – 2013. – Vol. 35. – P. 780–786. - Localization of Apaf1 gene expression in the early development of the mouse by means of in situ reverse transcriptase-polymerase chain reaction / M. Müller, J. Berger, N. Gersdorff, F. Cecconi, R. Herken, F. Quondamatteo // Developmental Dynamics. – 2005. – Vol. 234. – P. 215–221.
- Marrero, Y. P. B. Python Tutorial / Y. P. B. Marrero. – Lean Publishing, 2014.
- Mason, A. S. SSR Genotyping / A. S. Mason // Methods Mol. Biol. – 2015. – Vol. 1245. – DOI 10.1007/978-1-4939-1966-6_6.
- McGrann, J. Animal traceability across national frontiers in the European Union / J. McGrann, H. Wiseman // Revue Scientifique et Technique. – 2001. –
Vol. 20. – P. 406–412. - McKean, J. D. The importance of traceability for public health and consumer protection / J. D. McKean // Revue Scientifique et Technique. – 2001. – Vol. 20. – P. 363–371.
- Methylation of 12S rRNA is necessary for in vivo stability of the small subunit of the mammalian mitochondrial ribosome / M. D. Metodiev, N. Lesko,
C. B. Park, Y. Camara, Y. Shi, R. Wibom, K. Hultenby, C. M. Gustafsson,
N. G. Larsson // Cell Metabolism. – 2009. – Vol. 9. – P. 386–397. - Meuwissen, T. H. Mapping multiple QTL using linkage disequilibrium and linkage analysis information and multitrait data / T. H. Meuwissen, M. E. Goddard // Genet. Sel. Evol. – 2004. – Vol. 36. – P. 261–279.
- Meuwissen, T. H. Prediction of total genetic value using genome –wide dense marker maps / T. H. Meuwissen, B. J. Hayes, M. E. Goddart // Genetics. – 2001. – Vol. 157. – P. 1819–1829.
- Micorsatellite based parentage control in cattle / M. L. Glowatzki-Mullis,
C. Gaillard, G. Wigger, R. Fries // Animal Genetics. – 1995. – Vol. 26. – P. 7–12. - Miller, R. D. Regions of Low Single Nucleotide Polymorphism Incidence in Human and Orangutan Xq: Deserts and Recent Coalescences / R. D. Miller,
P. Taillon-Miller, P.-Y. Kwok // Genomics. – 2001. – Vol. 1. – P. 78–88. - Performance of different SNP panels for parentage testing in two East Asian cattle breeds / E. Strucken, B. Gudex, M. Ferdosi, H. Lee, K. Song, J. Gibson,
M. Kelly, E. Piper, L. Porto-Neto, S. H. Lee, C. Gondro // Animal Genetics. –
2014. – Vol. 45. – P. 572–575. - Peter, A. T. Abortions in dairy cows: New insights and economic impact /
A. T. Peter // Advances in Dairy Technology. – 2000. – Vol. 12. – P. 233–244. - Pettitt, R. G. Traceability in the food animal industry and supermarket chains / R. G. Pettitt // Revue Scientifique et Technique. – 2001. – Vol. 20. – P. 584–597.
- Rance, D. Necaise. Data structures and algorithm using Python / D. Rance. – Wiley, 2010.
- Raspor, P. Bio-markers: traceability in food safety issues / P. Raspor // Acta Biochimica Polonica. – 2004. – Vol. 52. – P. 659–664.
- Report From The Commission To The Council And The European Parliament: On the possibility of introduction of electronic identification for bovine animals. – 2005. – URL : http://eur-lex.europa.eu/LexUriServ/LexUriServ.do? uri=COM:2005:0009:FIN:EN:PDF.
- Resolution of quantitative traits into Mendelian factors by using a complete linkage map of restriction fragments length polymorphism / A. H. Paterson,
E. S. Lander, J. D. Hewitt, S. Paterson, S. E. Lincoln, S. D. Tanksley // Nature. – 1988. – Vol. 335. – P. 721–726. - Roles of Mis18a in epigenetic regulation of centromeric chromatin and CENP-A loading / I. S. Kim, M. Lee, K. C. Park, Y. Jeon, J. H. Park, E. J. Hwang,
T. I. Jeon, S. Ko, H. Lee, S. H. Baek, K. I. Kim // Molecular Cell. – 2012. –
Vol. 46. – P. 260–273. - Schütz, E. Analytical and statistical consideration on the use of the ISAG-ICAR-SNP bovine panel for parentage control, using the Illumina BeadChip technology: example on the German Holstein population / E. Schütz, B. Brenig // Genet Sel Evol. – 2015. – Vol. 47(1). – P. 3.
- Selection and use of SNP markers for animal identification and paternity analysis in U.S. beef cattle / M. P. Heaton, G. P. Harhay, G. L. Bennett, R. T. Stone, W. M. Grosse, E. Casas, J. W. Keele, T. P. L. Smith, C. G. Chitkon-McKown,
W. W. Laegreid // Mammalian Genome. – 2002. – Vol. 13. – P. 272–281. - The Holstein Friesian lethal haplotype 5 (HH5) results from a complete deletion of TFB1M and cholesterol deficiency (CDH) from an ERV-(LTR) insertion into the coding region of APOB / E. Schütz, C. Wehrhahn, M. Wanjek, R. Bortfeld,
W. E. Wemheuer, J. Beck, B. Brenig // PLOS One. – 2016. – Vol. 11. – P. e0154602. - The Mouse Genome Database (MGD): Premier model organism resource for mammalian genomics and genetics / J. A. Blake, C. J. Bult, J. A. Kadin,
J. E. Richardson, J. T. Eppig // Nucleic Acids Research. – 2011. – Vol. 39. –
P. D842–D848. - UniProt: The Universal Protein knowledgebase / R. Apweiler, A. Bairoch, C. H. Wu, W. C. Barker, B. Boeckmann, S. Ferro, E. Gasteiger, H. Huang, R. Lopez, M. Magrane, M. J. Martin, D. A. Natale, C. O’Donovan, N. Redaschi, L. S. Yeh // Nucleic Acids Research. – 2004. – Vol. 32. – P. D115–D119.
- Weber, J. L. Abudant class of human DNA polymorphism which can be typed using the polymerase chain reaction / J. L. Weber, P. E. May // American J. Human Genet. – 1989. – Vol. 44. – P. 388–396.
- Whole genome linkage disequilibrium maps in cattle / S. D. McKay,
R. D. Schnabel, B. M. Murdoch, L. Matukumalli, J. Aerts, W. Coppieters, D. Crews, E. Neto, C. Gill, C. Gao, H. Mannen, P. Stothard, Z. Wang, C. Tassell, J. Williams,
J. Taylor, S. Moore // BMC Genetics. – 2007. – Vol. 8. – P. 1–12. - Whole-genome sequencing of 234 bulls facilitates mapping of monogenic and complex traits in cattle / H. D. Daetwyler, A. Capitan, H. Pausch, P. Stothard,
R. van Binsbergen, R. F. Brøndum, X. Liao, A. Djari, S. C. Rodriguez, C. Grohs,
D. Esquerré, O. Bouchez, M. N. Rossignol, C. Klopp, D. Rocha, S. Fritz, A. Eggen, P. J. Bowman, D. Coote, A. J. Chamberlain, C. Anderson, C. P. Van Tassell, I. Hulsegge, M. E. Goddard, B. Guldbrandtsen, M. S. Lund, R. F. Veerkamp, D. A. Boichard, R. Fries, B. J. Hayes // Nature Genetics. – 2014. – Vol. 46. – P. 858–865. - Zane, L. Strategies for microsatellite isolation: a review / L. Zane,
L. Bargelloni, T. Patarnello // Mol. Ecology. – 2002. – Vol. 11. – P. 1–16. - Абельсон, Х. Структура и интерпретация компьютерных программ /
Х. Абельсон, Д. Сассман; пер. с англ. Г. К. Бронников. – 2-е изд. – М. : Добросвет, 2018.

