Титульный лист и исполнители
РЕФЕРАТ
Отчет 180 с., 3 ч., 6 табл., 35 рис., 209 источников литературы.
ВИНОГРАД, АБОРИГЕННЫЕ СОРТА, ДНК, ГИБРИДЫ, СЕЛЕКЦИЯ, ДНК-МАРКЕРЫ, ГЕНЫ, VVMYBA1, ИНТРОДУЦИРОВАННЫЕ СОРТА, КЛОНЫ, ПРОДУКТИВНОСТЬ.
Объект исследований – гены VvMybA1 и VvMybA2, детерминирующие окраску у сортов и клонов перспективных аборигенных и интродуцированных сортов винограда, отобранных в условиях Юга Российской Федерации и содержащихся в национальной ампелографической коллекции Анапской зональной опытной станции СКЗНИИСиВ.
В работе представлены результаты исследований молекулярного строения генов VvMybA1 и VvMybA2, влияние на фенотипическую проявляемость признака, представлены особи содержащие гомо- и гетерозиготное состояние изученных аллелей; сделаны выводы о потенциальном влиянии строения аллелей на конечный признак; в результате было построено три кластерных древа для изучения филогенетических взаимодействий изученных генотипов (по каждому гены у отдельности и оба вместе); сделаны предположения о происхождении некоторых сортов и их родстве.
Выявлены родственные взаимоотношения между аборигенными и интродуцированными сортами по средством обнаружения сходства строения исследованных генов с известными аборигенными и интродуцированными генотипами; определены гомо- и гетерозиготные генотипы.
Данные выводы могут быть использованы как при подборе пар для скрещивания с целью селекции новых высокопродуктивных и адаптивных сортов для выращивания на Юге Российской Федерации.
ВЕДЕНИЕ
Культурный виноград Vitis vinifera subsp. sativa D. C. является одной из наиболее ценных плодово-ягодных культур в мире по экономической эффективности и площади выращивания. Литературные, археологические и палео-ботанические источники позволяют понять распространение виноградарства в Европе и, в частности, в Российской Федерации, начиная с территории Кавказа [1, 9].
Сорта винограда, которые возделываются веками в традиционных винодельческих регионах, все чаще заменяются более высокопродуктивными клонами. Генетическое разнообразие, накопившееся за столетия культивирования традиционных сортов, крайне важно для улучшения их генома. Аборигенные сорта, вытесняемые новыми, представляют собой огромный, еще не раскрытый пласт знаний и значительный источник ценных генов для селекционной науки. Поэтому так важна молекулярно-генетическая идентификация коллекции АЗОСВиВ (Анапская зональная опытная станция виноградарства и виноделия), позволяющая не только узнать ее биологическое разнообразие, но также установить сортосоответствие в сравнении с другими известными мировыми коллекциями. Описание аборигенных сортов винограда по широкому спектру генетических признаков позволит не только создать ДНК-паспорта генотипов, но и использовать эти данные для установления родственных связей между ними
и при поисках их родителей, что станет неоценимой помощью селекционерам во время подбора родительских пар для скрещиваний.
Обширная исследовательская программа проводится во всех странах развитого виноградарства на распространенных известных сортах для отбора «чистокровных» высокоурожайных и высококачественных генотипов, обладающих различным спектром полезных для сельского хозяйства и востребованных потребителем признаков. И конечной целью проведения этих исследований является обеспечение материальной и теоретической базы для обоснования совершенствования клоновой селекции и повышения ее результативности [74, 169].
Как известно, с развитием молекулярной биологии возникла возможность ускорения селекционного процесса на базе использования молекулярно-генетических методов, в результате чего появилась маркерная селекция.
Сортовой состав популяций Краснодарского края и, в целом, Юга Российской Федерации крайне многообразен. Распространение современной методологии маркер опосредованной селекции выявило перспективность использования молекулярно-генетических методов анализа, что повысило ее эффективность, особенно после открытия наличия генов детерминирующих антоциановую окраску ягод винограда, набор которых не только специфичен для каждого сорта винограда, но и может отличаться по своему строению как от сорта к сорту, так и от клона к клону.
Первые результаты по идентификации клонов молекулярными маркерами показали, что такие НИР ограничиваются лишь набором использованных локусов, в которые могут входить различные маркерные признаки, такие как, например, микросателлитные маркеры, iPBS-маркеры, IRAP, ISSR и пр. Таким образом, при использовании большого количества маркеров вероятность найти различающиеся аллели повышается [158, 159].
Целью исследования было изучить молекулярно-генетическое разнообразие и строение генов VvmybA1 и VvmybA2 в семействе Vitaceae.
Для выполнения этой цели были поставлены следующие задачи:
- Сбор образцов. Выполнен в несколько этапов на нескольких коллекциях: ампелографическая коллекция АЗОС ВиВ и ампелографическая коллекция Крымской ОСС.
- Пробоподготовка и выделение ДНК. Пробоподготовка образцов листьев к выделению ДНК проводилась путем высушивания их в лиофильной сушке Martin Christ BETA 2-8 LDplus, имеющейся на кафедре биотехнологии, биохимии и биофизики Кубанского ГАУ. Дробление высушенных листьев производилось на кафедре на вибрационной мельнице GT200. Выделение ДНК из дробленых листьев осуществлялось при помощи DNeasy Plant Mini Kit и ЦТАБ-методом.
- ПЦР анализ. Праймерные пары и параметры амплификации уже разработаны и доступны в открытых литературных источниках.
- Очистка и клонирование амплифицированных фрагментов. Пробоподготовка к секвенированию проводилась путем очистки амплифицированных фрагментов при помощи QIAquick Gel Extraction Kit.
- Секвенирование. Секвенирование амплифицированных фрагментов двух генов проводилось в ЗАО «Евроген».
- Анализ полученых данных. Проводили in silico с использованием программ: ClustalO, NCBI BLAST, NCBI CD-search, MEGA7 и др.
1 ОБЗОР СОСТОЯНИЯ ИЗУЧЕННОСТИ ВОПРОСА
1.1 Основы ДНК-маркирования генотипов винограда
Внутривидовая гибридизация применяется в течение многих веков. Результатами ее являются многие сорта сельскохозяйственных культур, в том числе винограда. Анализ литературных источников показывает, что полученные результаты в этом направлении, хотя и значительные, но не решают всех проблем виноградарской отрасли.
Основная причина невысокой эффективности селекции винограда – недостаток знаний о биологических особенностях сортов и генетически обусловленных механизмах передачи признаков и свойств потомству. Это, конечно, не означает, что возможности межсортовой гибридизации исчерпаны и что от нее следует отказаться как от устаревшего и непродуктивного метода. В последнее время получено немало сортов, обладающих комплексом ценных свойств, которые были выделены методами как традиционной гибридизации, так и клоновой селекции, они регистрируются и обогащают промышленный сортимент
[8, 13, 45, 56].
Генетическая изменчивость сортов винограда, выраженная в возникновении мутаций и неоднородности исходного материала, проявляется в фенотипическом варьировании морфологических признаков, например, темпом роста, формирования органов, физиологических свойствах, изменяющих жизнеспособность организмов [28, 36, 43, 46]. Или фенотипически не выражается, например, в случаях изменения биохимических свойств, регулирующих синтез химических веществ [16, 24, 33]. Поэтому отбор можно вести по всем вышеперечисленным признакам и свойствам, особенно выделяя из них хозяйственно ценные. Однако морфологические, физиологические и биохимические признаки находятся в корреляции с генотипом и изменяются вместе с ним [14, 49, 53, 55]. Эти изменения могут быть вызваны влиянием каких-либо внешних условий, поэтому установить причину варьирования не всегда удается с достаточной точностью [19, 32, 42, 57]. Для этого необходимо использовать новые методы исследования сортов, такие как ДНК-маркирование [21, 35, 40]. В частности, направленных на идентификацию конкретных генов, несущих важное сельскохозяйственное значение.
ДНК-маркеры – характеристика генотипа, не зависящая от фенотипа, что обеспечивает богатство полиморфизмов, позволяющих идентифицировать сорта и строить точные генетические карты во многих высших растениях [12, 15, 30]. Высокое генетическое разнообразие у винограда, распространенного по всему миру, вызывает интерес к оценке генеалогии внутри рода Vitis, а также необходимость улучшения распознающих систем, пригодных для идентификации виноградных сортов [41, 58, 59]. Это поощряет попытки внедрения и использования данных о строении генов и генотипов в целом, из которых можно создавать сортовой резерв для улучшения существующего ампелографического разнообразия [4, 31, 44, 48, 135].
На данный момент, генетический резерв винограда – это смесь стародавних и недавно выведенных сортов [3, 9]. Перед тем как новые сорта полностью заменят старые, важно идентифицировать ценные древние генотипы и редкие формы. В настоящее время сорта идентифицируются различными молекулярными маркерами, такими как микросателлиты. Они представляют собой короткие последовательности ДНК, повторяющиеся несколько раз при каждом локусе. В связи
с ошибками прочтения, полимеразы, во время репликации ДНК, создали множество аллелей, которые различны по длине из-за разного количества повторов последовательности. В растениях микросателлиты встречаются часто – один на каждые 1–2,4 тыс. нуклеотидов в таких видах, как арабидопсис, рис, соя, кукуруза и пшеница. Множество SSRs было обнаружено в винограде, а также выявлена высокая гетерозиготность, что повышает полиморфизм микросателлитных маркеров и ведет к решению многих проблем, связанных с клоновой и сортовой идентификацией, а также анализом генеалогии [25, 26, 27, 209].
Молекулярные маркеры имеют огромный потенциал по поиску генетических различий между генотипами и определению разнообразия между клонами. Среди молекулярных маркеров, SSR-маркеры являются наиболее подходящими из-за их кодоминантности, огромного числа повторов и высокой частоты в селективно нейтральной области. Высоко насыщенные карты сцепления на основе одних только микросателлитных маркеров, а также их совместное использование с другими типами маркеров доступных в винограде. Микросателлитные маркеры оказались полезными для установления «личности» генотипов, фингерпринтинга и анализа разнообразия подвоев и сортов, а также межвидовых гибридов [100, 119].
В последние годы создание ДНК-отпечатков показало себя очень полезным для картирования геномов и изучения биоразнообразия. Достаточно высокий полиморфизм позволяет получать большое разнообразие генетических «отпечатков» при известном наборе генетических маркеров. Они доказали свое преимущество перед биохимическими и фенотипическими маркерами, которые имеют низкую величину полиморфизма. В основном, ДНК методы основаны на обнаружении полиморфных геномных последовательностей для внутренних сайтов рестрикции путем гибридизации клонированных зондов с ДНК. Например, длина полиморфных рестрикционных фрагментов (AFLP), случайно амплифицированная полиморфная ДНК (RAPD) или амплификация между простыми повторами последовательностей или микросателитов (SSR). Метод амплификации полиморфных фрагментов опирается на полиморфизм в сайтах узнавания для ферментов рестрикции, но с начала использования праймеров есть необходимость в анализе последовательности ДНК для обнаружения полиморфизмов.
1.2 Методы исследования полиморфизма ДНК
На сегодняшний день существует огромное количество методов исследования ДНК. Каждый из них предоставляет большое количество возможностей получить ответы на бесконечное количество вопросов. В данном разделе мы рассмотрим только те, которые использовались в процессе работы по исследованию полиморфизма генетических ресурсов винограда.
Наиболее простой способ выделения ДНК СТАБ-методом был описан еще в 1994 г. в статье «Простой и эффективный способ для выделения ДНК из виноградных растений» [143].
Развитие методов потребовало более «чистую» ДНК. Приведенный ниже метод выделения дает достаточно большой «урожай» ДНК, но имеющей примеси-загрязнители [153, 191]. Поэтому при дальнейшей работе этот метод был улучшен применением бета-меркаптоэтанола и поливинилпропиллидона, которые позволяют очистить ДНК от фенольных соединений и соединений ДНК
с сахаридами [62, 101, 102]. Такой метод называют «модифицированным СТАБ-методом». Он позволяет выделять ДНК не только из свежих листьев, но и из гербарных и мумифицированных, т. е. дает возможность изучать очень старые препараты листьев, что крайне важно при изучении происхождения видов и филогенеза [17, 18, 111].
Наличие загрязнителей сильно влияет на использование ДНК в исследованиях из-за ингибирования процесса реакции в целом и снижения активности Taq-полимеразы, а также из-за плохой очистки ДНК может меньше храниться из-за нестабильности. Поэтому так важно не только выделить большое количество ДНК, но и правильно и хорошо ее очистить [87, 125, 167].
В статьях, приведенных выше, описывается метод, дающий высокое количество чистой ДНК, которая долго хранится, но этот способ выделения имеет ряд отрицательных факторов. Во-первых, тратится много времени, во-вторых, используемые вещества, такие как хлороформ, изоамиловый спирт, бета-меркаптоэтанол являются крайне вредными для организма человека.
Такие чувства, как страх за свое здоровье, желание сделать больше за одинаковое количество времени, создали наборы для выделения нуклеиновых кислот различными методами. В настоящее время это направление активно развивается, создаются «наборы», позволяющие делать больше, быстрее и безопаснее, что сказывается не только на здоровье и работоспособности рабочих, но
и на финансовом положении лаборатории, так как исключаются затраты на защитное снаряжение лаборантов.
К примеру, выделение ДНК с использованием PeqGOLD PLANT DNA mini KIT – это дорогой, но очень эффективный способ выделения и очистки ДНК. Если при использовании вышеописанных методик медленно, но верно выделялось 12 образцов, то при применении данного набора и двух центрифуг выделялось 24 биотипа сразу [34, 129].
ПЦР – полимеразная цепная реакция. Этот этап был разработан Кэри Мюллисом, за что в 1993 г. он был удостоен Нобелевской премии по химии [157].
Следует упомянуть, что для более эффективной работы разработаны наборы и для ПЦР-реакций, тот же Peqlab-Kit. Результатом этого явилась унификация параметров реакции, так теперь не надо подбирать для каждого праймера свою температуру отжига [151]. Тем не менее, данный набор удобно использовать при мультиплексной ПЦР, когда применяется большое число ДНК-маркеров одновременно. В случае же, когда используются маркеры на конкретные генетические признаки, применяются специализированные под каждый маркер параметры, а они, в свою очередь, применяются отдельно друг от друга.
На сегодня разработано огромное количество праймеров, используемых
в работе. Они применяются как для генотипирования различных участков ДНК, как связанных, так и не связанных с кодирующими последовательностями.
И, конечно же, разработаны маркеры, помогающие идентифицировать гены устойчивости к вредителям и болезням, которые обычно имеются не только
в литературных источниках, но и в базах данных [146, 176].
Как уже было сказано, для большего упрощения и убыстрения работы были разработаны мультиплексы – подборка групп праймеров, гибридизирующихся с цепью исследуемой ДНК одновременно. Их подбирают таким образом, чтобы размер получаемых амплифицированных аллелей на перекрывал друг друга. Это крайне важно при анализе, дабы не возникло путаницы при дифференциации продуктов амплификации. При использовании мультиплексов
в гель-электрофорезе используют Touch Down PCR. Это такая разновидность полимеразной цепной реакции, где в первых циклах температура отжига праймера выше необходимой, а в последующих она снижается до оптимальной. Таким образом, убираются неспецифичные элементы отжига, мешающие различать продукты амплификации. Следует заметить, что в капиллярном электрофорезе аллели могут накладываться друг на друга, но при отображении в программе GeneMapper прекрасно различимы по цветам, поэтому они могут применяться вместе [130].
Существует много видов ПЦР-реакции. На наш взгляд, следует обратить внимание на ПЦР в реальном времени. Это далеко не дешевый метод, но он позволяет определить, где и сколько ПЦР продукта амплифицировалось, что избавляет от проверки наличия результатов на любом электрофорезе.
В данном методе используют флюоресцентно-меченые праймеры или ДНК-зонды. Также используется краситель SybrGreen 1, он обеспечивает достаточно простой и экономичный способ детекции, без необходимости применения зондов или специальных праймеров [130].
Очень важно, что qPCR (количественная ПЦР) можно комбинировать
с ОТ-ПЦР (обратно-транскрипционная ПЦР) для измерения малых количеств мРНК, что позволяет исследовать количественную информацию о содержании мРНК в клетке и, следовательно, позволяет судить о величине экспрессии гена в клетке или ткани.
Все полученные данные выводятся на экран компьютера и обрабатываются в реальном времени.
После амплификации следует визуализировать полученные ПЦР-продукты.
Визуализация результатов ПЦР. Как экспериментальный метод электрофорез был разработан отечественными учеными, профессорами Московского университета П. И. Стаховым и Ф. Ф. Рейссом в 1809 г.
На данный момент существует большое количество публикаций, описывающих конкретно электрофорез в полиакриламидном геле или же статьи, повествующие о работах, в которых гель-электрофорез применялся как основной метод разделения продуктов амплификации или же белков [83, 122, 190].
Самый распространенный в мире метод оценки длины амплифицированных фрагментов – это электрофорез в полиакриламидном геле. Но с этим методом связано большое количество проблем и трудностей в использовании. Например, плохая полимеризация акриламидного геля, когда с виду он без изъянов, а после просвечивания ультрафиолетом оказывается, что образцы уползли в край спейсера. Также возможно, что ДНК просто растеклась по акриламиду во время вымачивании в растворе бромистого этидия. Или же произошло отключение электроэнергии и после четырех часов электрофореза, оказывается, что все образцы выстроились в одну линию, где невозможно что-либо идентифицировать [20, 23].
В 1960-х гг. был предложен метод секвенирования биополимеров – установление их нуклеотидной или аминокислотной последовательности. В результате чего получают строение первичной структуры молекулы. Обычно, размеры секвенируемых участков ДНК 100 пар нуклеотидов (при использовании методов next-generation sequencing) и 1000 пар (при секвенировании по Сенгеру). В итоге получают последовательности участков генов, всю мРНК и даже целые геномы [2].
При секвенировании обычно применяют методы Эдмана, Сэнгера и др. До начала сиквенса производят амплификацию участка ДНК при помощи ПЦР, последовательность которого нужно установить.
Метод определения первичной последовательности пептидов разработан
в 1950–1956 гг. Виктором Эдманом. Обрабатывается исследуемый пептид специальным набором реагентов, это приводит к отщеплению одной аминокислоты с N-конца. Циклическое повторение реакции и анализ получаемой информации дают знания об исследуемом пептиде. В настоящее время он практически не применяется из-за неколичественного протекания реакции и побочных продуктов [37].
Другой метод секвенирования был открыт Фредериком Сенгером в 1977 г., за что он был удостоен Нобелевской премии по химии в 1980 г. Также метод известен как «метод обрыва цепи». Принцип метода заключается в том, что одна из цепочек ДНК является матрицей для синтеза второй [163].
Капиллярный электрофорез. Если в вышеописанных методах разделение происходит в полиакриламидной пластине, то здесь уже происходит разделение ионов по заряду, в тонком капилляре, заполненном электролитом.
Детектирование с помощью флуоресценции используют для образцов, имеющих собственную флуоресценцию или же модифицированных химическими метками. Данный способ обеспечивает высокую чувствительность. Также используют детектирование флуоресценции, вызванное лазером, подобные системы могут детектировать в пределах 10–18–10–21 моль [171]. При этом важно сказать, что именно данный метод обычно и используется для таргетного секвенирования выбранных участков геномов, полученных в результате амплификации
с ДНК-маркерами, при использовании прямого и обратного праймеров.
1.3 Ампело-генетический анализ сортов и клонов винограда
Комбинированные маркерные системы являются отличными дескрипторами, описывающими сорта винограда. Использование их совместно с фенотипическими характеристиками дает наиболее полную картину о состоянии совокупности внешних и внутренних признаков виноградной лозы.
В настоящее время изучение генофонда и фенотипического разнообразия винограда – приоритетная цель для ученых многих стран мира. Этот процесс происходит по признакам, принятым на вооружение под эгидой Международной организации винограда и вина. По большей части, это фенотипические признаки, такие как окраска и форма ягод, цвет и вкус вина, наличие антоциана, биохимические свойства, но большинство исследователей склоняются к тому, что следует использовать молекулярно-генетические маркеры, которые способны не только выявить отличия, но и показать скрытый потенциал у гетерозиготных особей.
В целом и частности, проблема дифференциации сортов и клонов винограда, отбор протоклонов и выделение из них самостоятельных сортов существует давно.
В практике виноградарства известны случаи изменчивости отдельных свойств и признаков. Древнеримские ученые Палладий и Колумелла, наблюдая такие факты, советовали использовать их в практически полезных для человека целях. Колумелла, например, писал, что следует черенковать и использовать
в производстве самые их плодородные части. Использовать клоновую селекцию советовали такие виноградари Швейцарии, Германии и Франции как Казенава, Гюйо, Каррьер, Сарториус, Мюллер-Тургау. В нашей стране основоположниками ее были С. А. Мельник, А. С. Мержаниан, М. П. Цебрия и др. Настойчиво рекомендовал применять отбор наиболее продуктивных растений И. В. Мичурин: «Тщательной селекцией (отбором) черенков, повторением отводки лучших частей лозы, сравнительно короткой обрезкой и посадкой на лучшую почву следует способствовать развитию лучших качеств» – писал он [7].
На данный момент, клоновой селекции винограда посвящено множество работ, признанных мировой научной элитой. Если говорить в целом, то работа по дифференциации клонов винограда включает в себя изучение фенотипической и генотипической изменчивости. И, как известно, иногда бывает, что фенотипа недостаточно, чтобы отличить сорта, тогда на помощь приходят молекулярно-генетические маркеры [15, 22, 29, 39].
В нашей стране методология проведения клоновой селекции винограда разработана профессором А. С. Мержанианом. На основании многолетних опытов и литературных данных о сортах Каберне-Совиньон, Рислинг, Алиготе, он пришел к выводу, что отбор клонов надо вести учитывая соотношение хозяйственно-ценных и морфологических признаков [38, 52]. Их он разделил на три группы: мутации, модификации (не передаются вегетативному потомству) и длительные модификации (передаются вегетативному потомству). Далее, этот метод был улучшен применением использования многомерного статистического анализа и технологии in vitro, что позволило поднять результативность клоновой селекции [51, 54].
Наиболее традиционным считается метод оценки полиморфизма по морфологическим признакам листьев среднего яруса кроны [10, 11]. В ампелографии полиморфизм – это явление биоразнообразия, одновременное существование нескольких форм растений, отличающихся друг от друга генотипически
и фенотипически и, таким образом, наличие клоновой изменчивости [1, 5, 6]. Гетерогенный полиморфизм сортов и клонов имеет большое практическое применение, так как они обеспечивают высокую экономическую отдачу при правильном их использовании. И ранее уже проводились исследования различных групп фенотипов сортов Пино и Рислинг – показано различие фенотипов групп винограда 231 учетного куста по 10 листовым признакам [47]. Помимо этого, также была проведена оценка полиморфизма сортогруппы Мерло на базе учебного хозяйства «Кубань» Кубанского государственного аграрного университета. В процессе работы авторами установлено, что суммарно незначительное отличие по каждому признаку, в комплексном анализе дает достаточно большую разницу при отборе клонов [50]. Таким образом, это позволило отобрать протоклоны сортов для дальнейшего исследования. В целом, как
в нашей, так и в других странах, этому направлению исследований отводятся отдельные темы научно-исследовательских работ [89, 194, 207].
Появление ДНК-маркеров произвело взрыв в клоновой селекции. Возможности ДНК-фингерпринтинга практически безграничны, они позволяют не только отличить друг от друга виноградные растения по каким-либо признакам, будь то гены или микросателлитные последовательности, но и создавать эталоны, с которыми можно сравнивать новые сорта и клоны.
Высокая изменчивость и генов и микросателлитных последовательностей дает возможность дифференцировать клоны внутри популяции [72, 110]. Применение этого метода привело к ускорению селекционного процесса. Вместе с идентификацией фенотипа этот метод дает огромное преимущество [134, 174, 180].
Например, описанным способом шестьдесят два иранских сорта были проанализированы по девяти микросателлитным маркерам. Было обнаружено наличие от 4 до 16 аллелей на локус в группах генотипов. Фенограммы, созданные для поиска различий среди сортов и клонов, выделили три группы, в которые были собраны все выбранные образцы. Среди иранских столовых сортов были обнаружены синонимы и омонимы [94].
В северо-западной части Испании, на астурианских виноградниках, выращивается много автохтонных сортов винограда. В связи со старостью виноградников, они характеризуются сортосмесью. С целью различения этих сортов и отбора перспективных протоклонов винограда, были проведены исследования по генотипированию 62 клонов виноградной лозы. По итогам отбора 62 генотипа были введены в селекционный процесс [132].
Достаточно большая работа была проведена Министерством сельского хозяйства США по генотипированию 3600 генотипов 35 видов винограда. Для исследования использовали 550 праймерных комбинаций. Было обнаружено от 16 до 38 различных аллелей на локус. Отсутствие аллелей не было обнаружено.
В результате исследования были созданы ДНК-паспорта выбранных клонов, также обнаружены два сорта, показавшие одинаковые аллели, а также выявлена высокая полезность SSR маркирования для винограда [197].
Созданы карты сцепления признаков при помощи SSR и SNP маркеров основных популяций винограда, таких как Сирах, Пино нуар, Гренаш, Каберне-Совиньон и Рислинг. В общей сложности для создания карты было использовано 1134 молекулярных маркера. Данные карты могут служить отличным инструментом в селекции виноградной лозы [169].
В Хорватии проводится постоянная работа по выведению новых сортов
и отбору протоклонов из старых образцов. Так, в соответствии с международной программой GrapeGen 06 на факультете агрономии в университете Загреба при помощи совместных методов фенотипического и генотипического анализа уже отобрано сто сортов. Была запущена не только клоновая селекция по отбору продуктивных клонов, но и фитосанитарная селекция наиболее устойчивых сортов к вредителям и болезням [149].
В Турции ведется активная работа по генотипированию сортов и клонов винограда. Например, 23 гибрида были отобраны для селекции по их сортовым характеристикам. Их исследовали в два этапа: по морфо-физиологическим
и генетическим параметрам. Фенотипирование проводили в соответствии со стандартной методикой OIV. В генотипировании использовали 20 микросателлитных маркеров, которые показали размер аллелей от 88 до 294 пар оснований [71].
Также странами-участницами программы GeneRes являются Молдова
и Румыния. В их задачу входит изучение сортов винограда бассейна Черного моря. Данная программа финансируется не только Европейским союзом, но
и персонально правительством Люксембурга, что подчеркивает ее значимость для мирового научного сообщества [131].
В Республике Словения широко выращивают технические сорта винограда. Более 50 редких традиционных сортов используется в производстве. Методика их сохранности как генотипов, а также идентификация их по внешним
и внутренним признакам была разработана на факультете биотехнологии Университета Любляна [161].
Наука не стоит на месте и поэтому произошло создание так называемых REMAP и IRAP техник для картирования генома винограда с помощью ретротранспозонных и интро-SSR-маркеров. Данные методы показывают еще большую вариабельность признаков не только между клоном и контролем, но
и между клонами в популяции. Это позволяет поднять на новый уровень клоновую селекцию. Совместное использование этой техники с ампелографическими данными и SSR-маркерами, позволяет получить наиболее полное представление об изучаемом клоне [75, 118, 165].
Мобильные генетические элементы были найдены почти во всех живых организмах в гетеро- и эухроматиновых областях генома. Последствия движения этих элементов в геноме может спровоцировать расширение повторяющихся некодирующих регионов, нарушение функции гена дикого типа, ведущее
к статусу псевдогена, модифицирование гена функционирования, что приводит к искажению выбираемых фенотипов и хромосомным перестройкам, приводящим к видообразованию [82. 119]. В европейском винограде наиболее известны три таких элемента Gret-1, Tvv-1 и Vine-1. Интересно, что Gret-1 и Vine-1 впервые были определены по их вставкам в регуляторные последовательности функциональных генов. В настоящее время только Gret-1 был полностью секвенирован, а также раскрыта его связь с мутациями, в результате чего в темно окрашенных сортах появляются светло окрашенные ягоды из-за его включения в промоутер VvMybA1 транскрипционного фактора, управляющего биосинтезом антоцианов в процессе созревания винограда [73, 78, 139].
Ретротранспозоны – это главные двигатели генетического разнообразия
и инструменты для обнаружения изменений в геноме, возникших под их влиянием. После демонстрации того, что они часто встречаются в растительных кариотипах, были созданы различные маркерные системы для детекции полиморфизма, вызванного ими, а также анализа генетического разнообразия в популяциях [116].
Ретротранспозоны являются основным агентом эволюции генома. Были разработаны различные системы молекулярных маркеров, которые используют метод анализа этих генетических элементов и их стабильную интеграцию
в дисперсные хромосомные локусы, которые полиморфны внутри каждого вида. Ключевыми методами SSAP, IRAP, REMAP, RBIP и ISBP обнаруживают сайты, в которых ретротранспозоны ДНК сохраняются между семьями элементов, интегрированых в геном. Маркерные системы, использующие эти методы, могут быть легко разработаны и применимы в отсутствии обширных данных
о последовательностях генома. Они предлагают доступ к динамическим и полиморфным частям генома и дополняют методы, такие как SNPs, которые нацелены, в первую очередь, на разделение генов [166].
Ретротранспозоны могут быть использованы в качестве маркеров, потому что их интеграция создает новые соединения между геномной ДНК и их консервативными концами. Для обнаружения полиморфизмов вставки ретротранспозона маркерные системы обычно полагаются на ПЦР-амплификации между этими концами и некоторые фланговые компоненты геномной ДНК. В этой связи были разработаны два метода: анализ ретротранспозона-микросателлит (REMAP) и внутренний полиморфизм ретротранспозона (IRAP), которые не требуют рестрикции ферментами. IRAP продукты, полученные от двух близлежащих ретротранспозонов, используются для амплификации последовательностей, стоящих перед праймерами. В REMAP амплификация между ретротранспозонными последовательностями простых повторов (микросателлитов) производит маркерные полосы [117, 118, 120].
В Испании были исследованы клоны сорта Темпоранилло с совместным использованием микросателлитных и ретротранспозонных маркеров. Всего было описано 28 клонов, из которых только один смог пройти отбор по генетическим маркерам. Учитывая, что в исследовании использовался всего один ретротранспозонный маркер, можно сделать вывод о их высоком полиморфизме [84, 196, 199, 202].
В Японии было проведено исследование по изучению влияния ретротранспозонов на окраску кожицы ягод винограда. По результатам был сделан вывод, что мутации в изменении окраски ягод могут быть вызваны ретротранспозонами, что было подтверждено молекулярно-генетическими исследованиями с применением маркеров на гены VvmybA1 и VlmybA1-1 [124]. Окраска ягоды является одной из самых главных характеристик сортов винограда. Это морфологическое свойство различает сорта друг от друга в момент полной зрелости [85, 192, 195]. Дополнение формы листа, грозди и окраски побега может быть достаточно, чтобы идентифицировать сорт [77, 175]. Сорта с окрашенной ягодой содержат антоцианы в эпидермисе. Количество и концентрация красящего вещества определяет их типичную окраску. VvmybA1 (Myb-связанный транскрипционный фактор) регулирует синтез антоцианов и экспрессию гена, ответственного за окраску ягоды [182, 186, 187]. Наличие ретротранспозона Gret-1 рядом с кодирующей последовательностью блокирует его экспрессию, вызывая осветление окраски кожицы [160, 178, 181].
В Италии были описаны 62 сорта по 30 SSR, 4 REMAP и 1 IRAP маркерам.
В результате выявили 58 маркер-специфичных аллелей, 22 генотип-специфичные аллели и четыре REMAP и IRAP частных аллели [203]. После чего в выборке сортов были найдены синонимы и омонимы. Также был сделан вывод, что совместное использование различных маркеров показывает себя наиболее эффективно и делает идентификацию генотипов наиболее легкой [204, 206, 208].
1.4 Анализ аллельного состояния локусов VvMybA1 и VvMybA2 у сортов и клонов винограда
Европейский виноград Vitis vinifera L. является одной из наиболее широко распространенных и экономически важных сельскохозяйственных культур. Такой признак как окраска ягоды – одна из важнейших характеристик при описании существующих сортов и в создании новых форм. И, как известно, окраска (как, впрочем, и некоторые другие признаки), зависит от количества и состава антоцианинов. Очевидно, что окрашенные сорта накапливают их в ягодах, в то время как не окрашенные – нет и, следовательно, не синтезируют такие пигменты [79]. Несмотря на то, что это одна из старейших многолетних одомашненных плодовых культур в мире, выбор метода культивации и переработки урожая виноградной лозы зависит от того, какой гибрид, клон и подвой используются в производстве, от его ампелографических свойств [173]. Очевидно, что вегетативное размножение позволило сохранить желаемые признаки, но в значительной степени повлияло на частоту спонтанных соматических мутаций, наблюдаемых в полевых условиях [65, 97]. Из этого следует, что многие признаки были отобраны не только в результате скрещиваний, но и клоновой селекции, например: урожайность с куста, форма и компактность грозди, размер и, конечно, цвет ягод [76, 112, 155]. В свою очередь, именно как результат гибридизации и мутаций мы можем видеть сорта, которые не только имеют или не имеют окраску ягод, но также формируют и промежуточные признаки окраски: розовые, красные, желтые и пр. Конечно же, важным фактором, влияющим на созревание и формирование ягод, является окружающая среда, в которой произрастает виноградное растение [60, 103, 107, 183]. Но все же окраска детерминируется генами, которые содержит тот или иной генотип. Изначально, метаболические пути антоцианового окраса регулируются группой генов Myb [99, 108, 124, 145, 200].
Изменения в семействе виноградных транскрипционных факторов VvMybA ответственны за содержание антоцианов в ягодах культурного и дикого винограда. Предыдущие исследования показали, что, например, неокрашенный виноград возник благодаря мутации двух соседних генов: инсерции ретроэлемента в VvMybA1 [141] и мутации однонуклеотидного полиморфизма
в VvMybA2 [66, 113]. Таким образом, исследования генетических основ данного признака показали, что кластер генов, расположенный во второй хромосоме, ответственен за большую часть изменения цвета, и, что фенотип обусловлен совместной работой генов VvMybA [156]. Данный локус состоит из трех генов типа MYB, среди которых, достоверно известно, что VvMybA1
и VvMybA2 функционально вовлечены в пигментацию ягод [145, 200]. И, на их примере, было продемонстрировано, что появление генотипа, характерного для белоягодных сортов, зависит именно от VvMybA1 и VvMybA2 [99, 145, 200]. Если, в частности, то блокирование функционирования гена VvMybA1 происходит из-за вставки транспозона Gret1 [141, 201], в то время как VvMybA2 может иметь однонуклеотидный полиморфизм (SNP) K980 в кодирующей последовательности, который модифицирует предполагаемую
α-спираль домена узнавания R2R3, и, также, приводит к потере функциональсти аллели [93, 200].
Таким образом, одним из наиболее важных генов, отвечающих за данный признак, является VvMybA1, которые имеет несколько вариативных аллелей [99]. Как было показано ранее, до этого целью проведенных исследований было изучение хорошо известных западных сортов-интродуцентов для нашей зоны. В то время как аборигенные сорта, бассейна Черного моря, обладающие огромным селекционным потенциалом для выведения новых гибридов и отбора клонов, остаются не изученными. Тем более, не изученными остаются
и структуры их конкретных генов, например, VvMybA1, изучение которых важно для выявления уникального генетического строения аллелей, понимания частных случаев фенотипического разнообразия и для изучения эволюции семейства Виноградовых в целом.
Исследование биологического разнообразия – одно из важнейших направлений современной ботаники. Как известно, изучение и описание растений проводится различными методами, которые, в основном, объединяют внешние и внутренние признаки: от строения листа до устройства генома. Не смотря на кажущуюся общность этих методов, они являются также и видоспецифичными, в виду того, что строения различных растений является уникальным и, соответственно, требует частного подхода.
Одним из таких уникальных и наиболее активно изучаемых растений является виноград культурный (Vitis vinifera L.). Для описания этого вида были разработаны специальные методики, утвержденные международным научным сообществом, в частности, организацией Office International de la Vigne et du Vin (IPGRI, 1997). В набор дескрипторов [64, 208] были включены, как и внешние признаки (форма и окраска побега, листа, ягод), так и генетические (микросателлитные последовательности в геноме винограда), которые остаются актуальными по сей день и используются во всех ампелографических коллекциях мира. В свою очередь, полученные данные объединяются в специализированные базы данных, что позволяет сравнивать изучаемые формы с установленными генотипами и определять, например, их чистосортность [90, 128].
Что касаемо морфологических дескрипторов, то все они характеризуют именно состояние признака. Это удобно при описании генотипов, но не при изучении фундаментальных причин, детерминирующих эти признаки, и определении филогенетических связей. Если брать во внимание общепринятые генетические дескрипторы, то установлено, что микросателлитные последовательности являются нейтральными и не обуславливают никаких признаков, но отлично подходят для филогенетического анализа, установления происхождения сортов или же изучения структуры генетических популяций [104]. Также,
в селекционной практике различных сельскохозяйственных культур используются и маркеры, которые позволяют выявлять хозяйственно ценные признаки или же маркеры, которые могут быть ассоциированы с какими-либо признаками и выявлять потенциальные значимые мутации.
Если говорить о виноградном растении, то одной из его отличительных особенностей является склонность к спонтанным мутациям, которые зачастую влияют и на хозяйственно ценные признаки [91, 98, 133, 188]. Поэтому, помимо нейтральных, используются также и маркеры, способные различать между собой клоны различных сортов винограда [121, 142]. Данные маркерные системы показывают в большинстве своем, случайные мутации, поэтому, поиск конкретных изменений, конечно же, производится, в основном, при помощи SNP генотипирования или секвенирования генов, отвечающих за определенные признаки, которые также могут иметь однонуклеотидные замены, влияющие на конечный признак [61, 105, 109].
Одним из таких признаков является накопление антоцианинов в ягодах.
В виноградном растении антоцианы синтезируются в разветвленном метаболическом процессе производства фенилпропанойдов, который на данный момент описан для многих растений [189, 205]. В частности, если говорить о винограде, то окраска ягод и, в целом синтез антоцианинов, является одним из ключевых факторов при отборе и выведении новых сортов и клонов, уже давно известны соматические вариации данного признака у этой экономически важной сельскохозяйственной культуры [185].
Выявлено что окраска ягод у винограда детерминируется кластером генов, расположенных во второй хромосоме генома [150, 201]. И, как было установлено, основную роль в формировании антоциановой окраски у винограда играют два гена: VvMybA1 и VvMybA2. При этом было обнаружено, что по той или иной причине, эти гены (одновременно или по одиночке) деактивированы в геномах белоягодных сортов. Как правило, причин три: 1) встраивание ретротранспозона Gret-1 в ген VvMybA1 [201]; 2) делеция в гене VvMybA1 [70]; 3) нуклеотидная замена в гене VvMybA2 [86]. В целом, доказано, что белоягодные формы и возникли из-за таких мутаций и закрепились в виде популяций, так как данные мутации устойчивы и стабильно передаются как вегетативному, так и генеративному потомству. Несмотря на это, данные мутации не являются необратимыми и известны случаи возвращения к окрашенному состоянию
у белоягодных сортов [88, 205].
На данный момент строение гена VvMybA1 изучено у достаточно многих генотипов винограда, относящихся к сортам и гибридам, выведенных за пределами России. При этом, генетическое разнообразие этого признака в формах винограда из региона Северного Кавказа, бассейна Черного моря и других географических областей России практически не изучено и, поэтому, представляет большой интерес для фундаментальной и прикладной молекулярной генетики
в виду потенциального наличия уникальных аллелей и в целях изучения происхождения аборигенных сортов. В свою очередь, добавление структуры генов Myb к описанию ампелографических признаков позволит более детально изучить биологическое разнообразие аборигенного винограда, и, возможно, выделить перспективные формы для селекции [162, 179].
Как уже было отмечено, окраска ягод винограда является одним из важнейших признаков в селекционной работе. По этой причине важно накапливать и анализировать информацию о строении генов, влияющих на этот признак, что позволит разрабатывать ДНК-маркеры для селекции [106]. Также важно отметить, что идентификация уникальных мутаций важна и для того, чтобы в дальнейшем их использовать с целью трансформации виноградного генома различными методами, исследования международных групп ученых по этой проблеме активно ведутся [92, 147, 154, 177, 184, 193]. В том числе, перенос данных генов находит применение не только для самого винограда, но и других растений [154].
Vitis vinifera L. – одна из наиболее древних и распространенных растительных культур, издавна выращиваемых человеком. И, как известно, чаще всего данная культура используется для дальнейшей переработки урожая
в вино. При этом хорошее вино – это продукт, который обладает балансом множества различных характеристик, например, кислотность, сахаристость, наличие различных аминокислот и т. д. В свою очередь, такие общие показатели как качество и количество вина зависят от еще большего числа факторов, таких как почвенно-климатические условия, наличие полива и наличие вредителей [67, 68]. Тем более, установлено, что цены на вино, произведенное на одной и той же территории, могут варьировать различное количество раз из года в год только из-за годовых погодных условий [67]. С другой стороны, стоимость вина, произведенного в один год, может варьировать из-за области, где оно произведено [67, 69].
Помимо этого, климат и агротехнология выращивания, в совокупности
с внутренними факторами сильно влияют и на продолжительность жизни вина, так как в отличие от остальных продуктов переработки, после открытия бутылки – вкус и качество сильно изменяются с течением времени. И такая возможность сохранения создает еще более высокий и желанный для потребителя класс вин [95, 96, 136, 164].
Но несмотря на это, основной признак, на который обращает внимание потребитель при выборе вина – это его цвет [127]. Очевидно, фактом является то, что цвет выбранного вина играет и важную роль в представлении потребителем возможного вкуса вина и, в общем и целом, всех его ожиданий [148, 170]. Для самого же вина важно, что наличие пигментных компонентов влияет на его состав, например, наличие антиоксидантов в красных винах [152, 198]. В частности, в молодых красных винах окраска усиливается также и содержанием полифенольных соединений, присутствующих в кожице ягод, одними из наиболее распространенных являются антоцианины [80].
Как было установлено, антоциановая окраска ягод винограда контролируется кластером генов, расположенных в хромосоме 2 [88, 137, 156]. Этот локус близкородственен к R2R3-Myb генам и псевдогенам, среди которых функциональны MybA1 и MybA2, являющимися двумя смежными транскрипционными факторами, которые регулируют окраску ягод. Помимо них существуют MybA3 и MybA4, при этом они не показали наличие значительного (MybA3) или какого-либо (MybA4) влияния на синтез антоцианинов [140, 200]. Таким образом, сейчас научным сообществом, в основном, изучаются строение
и экспрессия именно двух генов VvMybA1 и VvMybA2, у которых было
открыто наличие аллелей, различных по своему строению, детерминированию признака и экспрессии, в том числе и возникающих при соматических мутациях [140, 141, 172].
На данный момент описаны различные варианты строения аллелей данных генов как для окрашенных сортов, так и для неокрашенных [123, 141]. При этом важно отметить, что было выявлено не только потеря окраски у мутантных форм [200], но и ее восстановление [88], что, конечно же, является интересным для конечного потребителя. Поэтому, в нашем исследовании мы поставили целью изучение строения аллелей генов VvMybA1 и VvMybA2 у ранее отобранных генотипов.
2 МАТЕРИАЛЫ И МЕТОДЫ
2.1 Растительный материал
В качестве растительного материала, нами были выбраны 105 генотипов сортов, форм и клонов винограда для изучения строения их аллелей генов VvMybA1 и VvMybA2 (таблица 1). При этом, в список вошли: аборигенные сорта, сорта новой селекции и перспективные клоны, отобранные в результате предыдущих работ профессором Л. П. Трошиным.
Таблица 1 – Список генотипов, отобранных для исследования
| 1 | Кумшатский | 28 | Андижанский черный |
| 2 | Варюшкин 9 ряд кл.19 (26) | 29 | Босса |
| 3 | Траминер | 30 | Сиргула |
| 4 | Совиньон Таманский | 31 | Хатми |
| 5 | Профессор Трубилин | 32 | Плавай |
| 6 | Совиньон Таманский 114-35 куст |
33 | Мсхвилтвала Тетри |
| 7 | Сибирьковый 14р. Клет. 19с | 34 | Тита рбили |
| 8 | Шаани | 35 | Тавлинский поздний |
| 9 | Алый терский | 36 | Тавквери |
| 10 | Португальский синий | 37 | Пти Вердо 1 к3 р7 |
| 11 | Кульджинский | 38 | Рислинг Рейнский 1-7 (7) |
| 12 | Чинури 1-7 (18-19) | 39 | Солярис К |
| 13 | Риш баба 1-8 (33-34) | 40 | Шардоне 1-7 (13-14) |
| 14 | Чауш | 41 | Панагия Сумела 2 |
| 15 | Уни белый (92-93) | 42 | Панагия Сумела 11 |
| 16 | Нимранг 1-11 (19-20) | 43 | Жупски баядисер 3куст |
| 17 | Кизил сапак 1-8 (61-62) | 44 | Кумшацкий белый |
| 18 | Буланый 1-36 (63-64) | 45 | Сыпун черный |
| 19 | Хатми 1-11 (76-77) | 46 | Будай шули |
| 20 | Воскеат 1-38 (70-71) | 47 | Гок изюм |
| 21 | Цоликаури 1-36 (49-50) | 48 | Гюляби дагестанский |
| 22 | Шабаш | 49 | Гок ала |
| 23 | Гамэ черный | 50 | Яй изюм розовый |
| 24 | Чиляки 1-23 (35-36) | 51 | Яй изюм белый |
| 25 | Тербаш 1-23 (35-34) | 52 | Рислинг Алькадар 34а |
| 26 | Баян Ширей | 53 | Рислинг 34г |
| 27 | Катта-Курган | 54 | Рислинг К-830 |
| Продолжение таблицы 1 | |||
| 55 | Пино белый 32 | 97 | Долгожданный 6-9 |
| 56 | Рислинг 9-9-1 | 98 | Долгожданный |
| 57 | Рислинг 2-19-6-1 | 99 | Долгожданный Ф |
| 58 | Каберне-Совиньон 337 | 100 | Антоний Великий 30-5 |
| 59 | Рислинг 3-14-11-11-1 | 101 | Антоний Великий 30-6 |
| 60 | Пино белый 31 | 102 | Антоний Великий |
| 61 | Рислинг 3-14-20-9-2 | 103 | Антоний Великий Ф |
| 62 | Рислинг 9-6-4 | 104 | Аркадия розовая 2-5 |
| 63 | Пино белый 46 | 105 | Аркадия розовая 2-6 |
| 64 | Каберне-Совиньон 5А | ||
| 65 | Каберне-Совиньон 169 | ||
| 66 | Рислинг 7-11-18-9-1 | ||
| 67 | Каберне-Совиньон 15КМ | ||
| 68 | Каберне-Совиньон 15КМ | ||
| 69 | Рислинг 3-14-9-9-1 | ||
| 70 | Пино белый 6 | ||
| 71 | Рислинг 7-15-107-7п | ||
| 72 | Рислинг 3-14-4-11-1 | ||
| 73 | Рислинг 7-12-201-15-1 | ||
| 74 | Клон Грамотенко | ||
| 75 | Алиготэк | ||
| 76 | Мерло 14МГ | ||
| 77 | Рислинг контроль | ||
| 78 | Каберне-Совиньон контроль | ||
| 79 | Алиготе контроль | ||
| 80 | Совиньон 1 | ||
| 81 | Совиньон 2 | ||
| 82 | Совиньон 3 | ||
| 83 | Совиньон 4 | ||
| 84 | Совиньон контроль | ||
| 85 | Мерло контроль | ||
| 86 | Анюта 5-5 | ||
| 87 | Анюта | ||
| 88 | Анюта Ф | ||
| 89 | Ливия 14-6 | ||
| 90 | Ливия 14-5 | ||
| 91 | Ливия | ||
| 92 | Ливия Ф | ||
| 93 | Гелиос 50-5 | ||
| 94 | Гелиос 50-6 | ||
| 95 | Академический К | ||
| 96 | Долгожданный 6-8 | ||
Помимо этих сортов, также в опыт были добавлены и другие, при невозможности идентифицировать какую-либо из аллелей у выбранных сортов.
2.2 Выделение ДНК
Выделение ДНК проводилось двумя способами: ЦТАБ-методом и при помощи коммерческого набора DNeasy Plant Mini Kit фирмы QIAGEN. Перед выделением ДНК из листьев, отобранные растительные ткани высушивались
в лиофильной сушке Martin Christ BETA 2-8 LDplus в течение 24 ч. После этого, производилось их дробление при помощи металлических шариков в пробирках с пробами в вибрационной мельнице GT200. Ниже для каждого метода приводятся протоколы.
2.2.1 Выделение ДНК ЦТАБ-методом
Протокол выделения ЦТАБ-методом:
- К дробленой растительной ткани добавляли 1–2 % поливинилплоипиролидина и 810 мкл ЦТАБ-буфера. Полученный гомогенизат перемещали на инкубацию в термостат на 65 °С на 30 мин. В процессе инкубации перемешивали содержимое пробирки на вортексе каждые 5 мин.
- После инкубации охлаждали пробирки до комнатной температуры
и добавить в каждую пробу по 810 мкл раствора хлороформа и изоамилового спирта, смешанного в соотношении 24 : 1. - Далее перемешивали на вортексе, после чего центрифугировали 15 мин со скоростью 13000 об./мин.
- Получившийся супернатант переносили в новую пробирку объемом 1,5 мл.
- К нему добавляли 50 мкл 3М ацетата натрия, 150 мкл 4М хлорида натрия и 540 мкл холодного изопропанола.
- Далее, перемешивали на вортексе и инкубировали при температуре минус 20 °С в течение 30 мин.
- После чего центрифугировали в течении 15 мин со скоростью
13000 об./мин. - Промывание образовавшегося осадка выполняли 70%-м этиловым спиртом объемом 500 мкл. Для этого удаляли из пробирки жидкую фазу, приливали к осадку спирт, перемешивали на вортексе, после чего спирт удаляли пипеткой.
- Следующим этапом – в термостат помещали пробирки с открытыми крышками и высушивали осадок при температуре 37 °С в течение 20 мин.
- После чего добавляли к осадку 300 мкл ТЕ буфера и 1 мкл РНКазы.
- Данную смесь перемешивали на вортексе и инкубировали на термостате один час при температуре 37 °С.
- Потом добавляли 100 мкл 10М ацетата аммония и 1000 мкл холодного этилового спирта.
- После перемешали на вортексе и инкубировали при температуре минус 80 °С в течение 5 мин.
- После чего центрифугировали 10 мин при 13000 об./мин.
- Жидкую фазу удаляли и промывали 70%-м этиловым спиртом объемом 500 мкл.
- Потом осадок высушивали в пробирках с открытыми крышками в термостате 20 мин при температуре 37 °С до полного испарения этанола.
- В конце к осадку приливали 50 мкл ТЕ. Для лучшего растворения ДНК, пробирки инкубировали при плюс 3 °С в течение 12 ч.
2.2.2 Выделение ДНК с использованием набора DNeasy Plant Mini Kit
Протокол выделения ДНК коммерческим набором DNeasy Plant Mini Kit компании QIAGEN:
- К раздробленным тканям листьев добавляли 400 мкл буфера AP1
и 4 мкл РНКазы А. Перемешивали на вортексе и инкубировали в течение 10 мин при 65 °С. Перемешивали содержимое пробирки 2–3 раза во время инкубации. - Приливали 130 мкл буфера P3. Перемешивали и инкубировали в течение 5 мин на льду.
- После чего центрифугировали лизат в течение 5 мин на скорости
20000 об./мин. - Затем переносили верхнюю фазу в спин-колонку QIAshredder, находящуюся в прозрачной пробирке для сбора без крышки объемом 2 мл. Центрифугировали в течение 2 мин на скорости 20000 об./мин.
- Аккуратно переносили жидкость, образовавшуюся под колонкой, в новую пробирку, не допуская встряхивания. К жидкости добавляли 1,5 объема буфера AW1 и перемешивали на вортексе.
- Переносили 650 мкл смеси в спин-колонку DNeasy Mini, помещенную
в пробирку объема 2 мл. Центрифугировали в течение 1 мин на скорости
≥ 6000 об./мин. Повторяли этот шаг с оставшимся образцом необходимое количество раз. - Помещали спин-колонку в новую пробирку объемом 2 мл. Добавляли 500 мкл буфера AW2, центрифугировали в течение 1 мин на скорости
≥ 6000 об./мин. Удаляли жидкость, образовавшуюся под колонкой в прозрачной пробирке. - После добавляли еще 500 мкл буфера AW2. Центрифугировали в течение 2 мин на скорости 20000 об./мин.
- Переносили спин-колонку в новую пробирку объемом 1,5 мл или 2 мл.
Примечание: извлекать спин-колонку из пробирки следует осторожно, не допуская контакта с жидкостью, находящейся под ней.
- Затем добавляли 100 мкл буфера AE для элюции и инкубировали в течение 5 мин при комнатной температуре (15–25 °C). После инкубации центрифугировали в течение 1 мин на скорости 6000 об./мин
- Еще раз повторяли 11 шаг для увеличения количества полученной ДНК.
2.2.3 Оценка качества и количества выделенной ДНК
Оценку качества и количества ДНК, выделенной из высушенных листьев, проводили двумя методами: 1) при помощи агарозного гель-электрофореза;
2) при помощи спектрофотометра Implen NanoPhotometer NP80.
После проведения измерений, тотальная ДНК разбавлялась в деионизированной воде до концентрации 20 нг/мкл.
2.3 Проведение полимеразной цепной реакции
Полимеразная цепная реакция проводилась в амплификаторе ДТ-322, компании «ДНК-Технология». Для проведения ПЦР были выбраны праймеры, созданные с целью идентификации в генотипах винограда генов VvMybA1
и VvMybA2 и, соответственно, ранее опубликованные в статьях [63, 201]. Последовательности праймеров приведены в таблице 2.
Таблица 2 – Нуклеотидные последовательности, использованные
в работе праймеров
| Название | Нуклеотидная последовательность |
| VvMybA1_F | GACGTAAAAAAATGGTTGCACGTG |
| VvMybA1_R | GAACCTTCTTTTTGAAGTGGTGACT |
| VvMybA2_F | GTGAGGAGAGTACATTGTAGGA |
| VvMybA2_R | GAACCTTCTTTTTCAGGTGGTGACC |
При проведении ПЦР использовались следующие параметры амплификации:
95 °С – начальная денатурация, 5 мин;
далее 30 циклов:
95 °С –денатурация, 30 с;
55 °С – отжиг праймеров, 30 с;
72 °С – элонгация, 1 мин;
после чего дополнительный шаг:
72 °С – финальная элонгация, 5 мин.
2.4 Проверка наличия амплифицированных фрагментов (ДНК-бендов)
Проверка наличия амплифицированных фрагментов в результате ПЦР проводилась в камере для горизонтального электрофореза компании «Helicon», в 2%-м ТАЕ-агарозном геле. Параметры гель-электрофореза были выбраны следующие: 150V, 150А, в течение 20 мин. Далее агарозные пластины фотографировались в ультрафиолетовом свете и неудачные образцы ПЦР отмечались для повторного проведения амплификации.
2.5 Выделение и очистка амплифицированных фрагментов
Полученные фрагменты вырезали из гелевой пластины скальпелем и выделяли при помощи набора Cleanup Standart, компании ЗАО «Евроген» (http://evrogen.ru/kit-user-manuals/BC022.pdf). Далее приводится протокол выделения и очистки ПЦР-фрагментов на колонке с незначительными изменениями, примененными во время работы.
Объемы растворов в наборе рассчитаны на средний вес фрагментов геля 150 мг.
Протокол выделения и очистки ПЦР-фрагментов:
- Фрагмент геля с целевой ДНК вырезали скальпелем в слабом ультрафиолетовом свете.
- После чего гель помещали в микроцентрифужную пробирку объемом 2 мл.
- К гелю добавляли три объема «связывающего раствора» (но не менее 350 мкл). Тем не менее, в связи с рекомендацией, при использовании гелей
с концентрацией агарозы 1,8 % и выше, количество «связывающего раствора» было увеличено до четырех–пяти объемов. - Далее смесь инкубировали при 50–55 °C на твердотельном термостате «Термит» (компания «ДНК-Технология») до полного растворения геля. Для ускорения растворения раствор перемешивали путем встряхивания пробирки
на вортексе каждые 5 мин. - После чего необходимо перейти к пункту «II. Выделение ДНК на колонке».
Протокол выделения ДНК на колонке:
- Поместить спин-колонку в собирательную пробирку.
- Перенести пробу в колонку и центрифугировать 30 с. Удалить фильтрат из собирательной пробирки, расположенной снизу. В связи с тем, что объем пробы был 1000 мкл, ее разделяли на несколько нанесений на колонку. После каждого нанесения аликвоты колонку центрифугировали.
- После чего добавляли 700 мкл «промывочного раствора» в колонку
и центрифугировали 30 с. Фильтрат, собранный снизу, удаляли. - Повторить пункт 3.
- Пустую колонку центрифугировали 1 мин для полного удаления промывочного раствора.
- Колонку переставляли в новую пробирку на 1,5 мл.
- После чего оставляли при комнатной температуре на 10 мин для испарения остатка спирта.
- В центр мембраны приливали 50 мкл элюирующего раствора и оставляли на 5 мин.
- Центрифугировали 30 с.
- Элюирующий раствор (50 мкл) повторно наносили на колонку, оставляли на 5 мин и центрифугировали 30 с для увеличения выхода ДНК.
Очищенную ДНК хранили при минус 20 °C.
Концентрация образца ПЦР измерялась нанофотометром Implen NanoPhotometer NP80 и доведилась деионизированной водой до объема 6 мкл, содержащего очищенный фрагмент ПЦР в количестве 30 нг/мкл.
2.6 Секвенирование амплифицированных фрагментов
Секвенирование проводилось компанией ЗАО «Евроген» (http://evrogen.ru/
services/sequencing/service-sequencing.shtml) методом Сэнгера, с использованием прямого и обратного ПЦР праймеров (таблица 2). Такой метод секвенирования был выбран для достижения наиболее достоверного результата, так как некоторые из изучаемых генотипов были геторозиготны, а также сравнение прямой и обратной последовательности позволяло избежать ошибок при секвенировании, которые могли возникнуть при амплификации Taq-полимеразой
в процессе ПЦР.
2.7 Анализ нуклеотидных последовательностей
Анализ на соответствие ожидаемому результату проводился посредством поиска последовательности в базе данных NCBI, используя BLAST, blastx
и CD-search [138].
Выравнивание полученных ДНК и протеиновых последовательностей проводили стандартными параметрами в программе ClustalO, форматами VIENNA (для выравнивая в fasta формате) и ClustalW (для дальнейшего анализа) [168]. Анализ выравненных последовательностей проводили при помощи интерфейса MView [81].
Филогенетические отношения между изучаемыми последовательностями аминокислот устанавливали с использованием программы MEGA7 [126] методом Maximum Likelihood [144], с моделью Jones-Taylor-Thornton [115]
и 999 бутстрепами. Координатный анализ строился при помощи программы Jalview [114].
3 РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЙ
В результате исследований были получены последовательности исследованных генов у 105 генотипов. Данные сиквенсы были сравнены между собой
и выявлены полиморфные участки. В итоге, для получения понятия о родстве генотипов были использованы методы кластеризации изученных ДНК-последовательностей.
3.1 Изучение аллелей гена VvMybA1 у аборигенных сортов
В результате амплификации ДНК-праймерами VvMybA1_F и VvMybA1_R (таблица 3), нами были выявлено наличие искомых аллелей гена VvMybA1
у всех генотипов. Результаты разделения продуктов амплификации в 2%-м агарозном геле отображены на рисунках 1–6.
Таблица 3 – Результаты поиска в базе данных NCBI
| Сорт | Результат/процент идентичности | Источник |
| 1 | 2 | 3 |
| Яй изюм белый |
Vitis vinifera cultivar Sirch MybA1 gene / 100 % | Roshani-Bakhsh et al., 2017, не опубликовано |
| Гюляби дагестанский |
Vitis vinifera VvMybA3 gene for myb-related transcription factor / Vitis vinifera DNA, retrotransposon Gret1, complete sequence and VvMybA1 gene for Myb-related transcription factor / 99,84 % | Azuma et al., 2009 |
| Воскеат | Vitis vinifera DNA, retrotransposon Gret1, complete sequence and VvMybA1 gene for Myb-related transcription factor / 100 % | Azuma et al., 2009 |
| Чиляки | Vitis vinifera VvMybA3 gene for Myb-related transcription factor / Vitis vinifera DNA, retrotransposon Gret1, complete sequence and VvMybA1 gene for Myb-related transcription factor / 100 % | Azuma et al., 2009 |
| Шабаш | Vitis vinifera DNA, retrotransposon Gret1, complete sequence and VvMybA1 gene for Myb-related transcription factor / 100 % | Azuma et al., 2009 |
| Цоликаури | Vitis vinifera DNA, retrotransposon Gret1, complete sequence and VvMybA1 gene for Myb-related transcription factor / 99,84 % | Azuma et al., 2009 |
| Чауш | Vitis vinifera DNA, retrotransposon Gret1, complete sequence and VvMybA1 gene for Myb-related transcription factor / 100 % | Azuma et al., 2009 |
| Продолжение таблицы 3 | ||
| 1 | 2 | 3 |
| Совиньон Таманский |
Vitis vinifera DNA, retrotransposon Gret1, complete sequence and VvMybA1 gene for Myb-related transcription factor / 100 % | Azuma et al., 2009 |
| Риш баба | Vitis vinifera DNA, retrotransposon Gret1, complete sequence and VvMybA1 gene for Myb-related transcription factor / 100 % | Azuma et al., 2009 |
| Шаани | Vitis vinifera DNA, retrotransposon Gret1, complete sequence and VvMybA1 gene for Myb-related transcription factor / 100 % | Azuma et al., 2009 |
| Хатми | Vitis vinifera DNA, retrotransposon Gret1, complete sequence and VvMybA1 gene for Myb-related transcription factor / 100 % | Azuma et al., 2009 |
| Плавай | Vitis vinifera DNA, retrotransposon Gret1, complete sequence and VvMybA1 gene for Myb-related transcription factor / 100 % | Azuma et al., 2009 |
| Баян Ширей |
Vitis vinifera DNA, retrotransposon Gret1, complete sequence and VvMybA1 gene for Myb-related transcription factor / 100 % | Azuma et al., 2009 |
| Мсихлтвала Тетри |
Vitis vinifera DNA, retrotransposon Gret1, complete sequence and VvMybA1 gene for Myb-related transcription factor / 100 % | Azuma et al., 2009 |
| Катта-Курган | Vitis vinifera DNA, retrotransposon Gret1, complete sequence and VvMybA1 gene for Myb-related transcription factor / 100 % | Azuma et al., 2009 |
| Тита Рбили | Vitis vinifera DNA, retrotransposon Gret1, complete sequence and VvMybA1 gene for Myb-related transcription factor / 100 % | Azuma et al., 2009 |
| Уни белый | Vitis vinifera DNA, retrotransposon Gret1, complete sequence and VvMybA1 gene for Myb-related transcription factor / 100 % | Azuma et al., 2009 |
| Хатми | Vitis vinifera DNA, retrotransposon Gret1, complete sequence and VvMybA1 gene for Myb-related transcription factor / 100 % | Azuma et al., 2009 |
| Тербаш | Vitis vinifera DNA, retrotransposon Gret1, complete sequence and VvMybA1 gene for Myb-related transcription factor / 100 % | Azuma et al., 2009 |
| Сиргула | Vitis vinifera DNA, retrotransposon Gret1, complete sequence and VvMybA1 gene for Myb-related transcription factor / 100 % | Azuma et al., 2009 |
| Чинури | Vitis vinifera DNA, retrotransposon Gret1, complete sequence and VvMybA1 gene for Myb-related transcription factor / 100 % | Azuma et al., 2009 |
| Босса | Vitis vinifera DNA, retrotransposon Gret1, complete sequence and VvMybA1 gene for Myb-related transcription factor / 99,84 % | Azuma et al., 2009 |
| Академик Трубилин |
Vitis vinifera Pinot noir transcription factor MYBA3 (MybA3) gene, MybA3-2 allele / 99,83 % | McCallum et al., 2009, не опубликовано |
| Продолжение таблицы 3 | ||
| 1 | 2 | 3 |
| Гамэ черный |
Vitis hybrid cultivar Grand Noir Chateau des Charmes transcription factor MYBA1 (MybA1) gene, MybA1-1 allele / 100 % | McCallum et al., 2009, не опубликовано |
| Гок ала | Vitis vinifera cultivar Alphonse Lavalle MYBA1 (MybA1) gene, mybA1-ALF allele / 99,84 % | Lijavetzky et al., 2006 |
| Тавквери | Vitis vinifera cultivar Panje Arus MybA1 gene / 99,84 % | Roshani-Bakhsh et al., 2017, не опубликовано |
| Андижанский черный | Vitis vinifera cultivar Panje Arus MybA1 gene / 100 % | Roshani-Bakhsh et al., 2017, не опубликовано |
| Тавлинский поздний | Vitis vinifera cultivar Panje Arus MybA1 gene / 100 % | Roshani-Bakhsh et al., 2017, не опубликовано |
| Кумшацкий черный | Vitis vinifera cultivar Panje Arus MybA1 gene / 100 % | Roshani-Bakhsh et al., 2017, не опубликовано |
| Буланый | Vitis vinifera cultivar Panje Arus MybA1 gene / 100 % | Roshani-Bakhsh et al., 2017, не опубликовано |
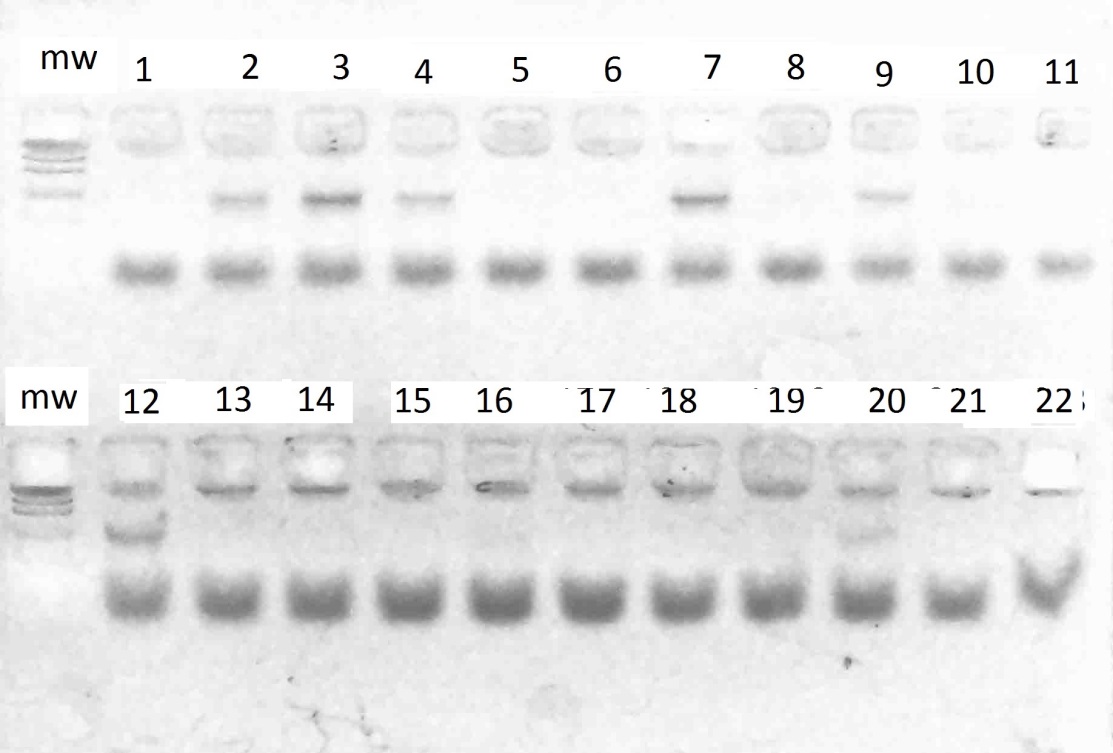
Рисунок 1 – Разделение продуктов амплификации ДНК аборигенных сортов
с праймерами VvMybA1_F и VvMybA1_R

Рисунок 2 – Разделение продуктов амплификации ДНК аборигенных сортов
с праймерами VvMybA1_F и VvMybA1_R
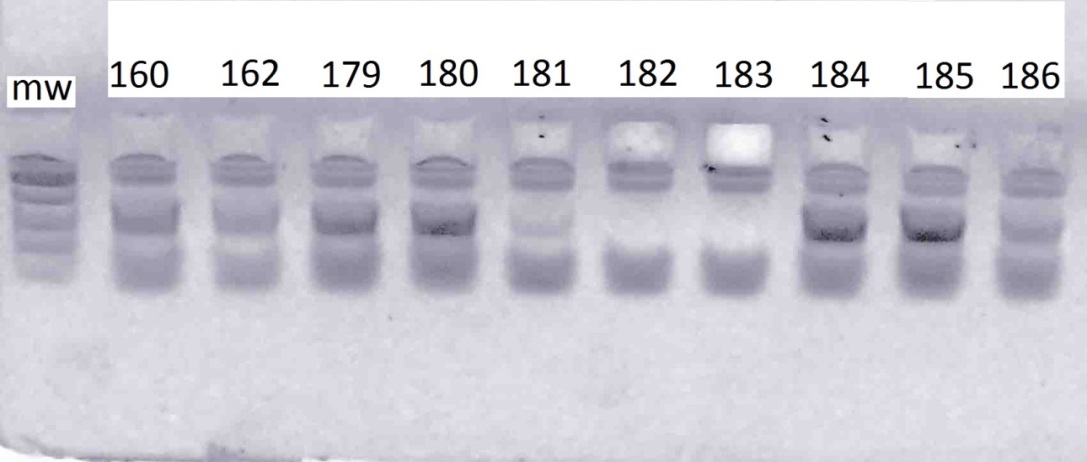
Рисунок 3 – Разделение продуктов амплификации ДНК аборигенных сортов
с праймерами VvMybA1_F и VvMybA1_R
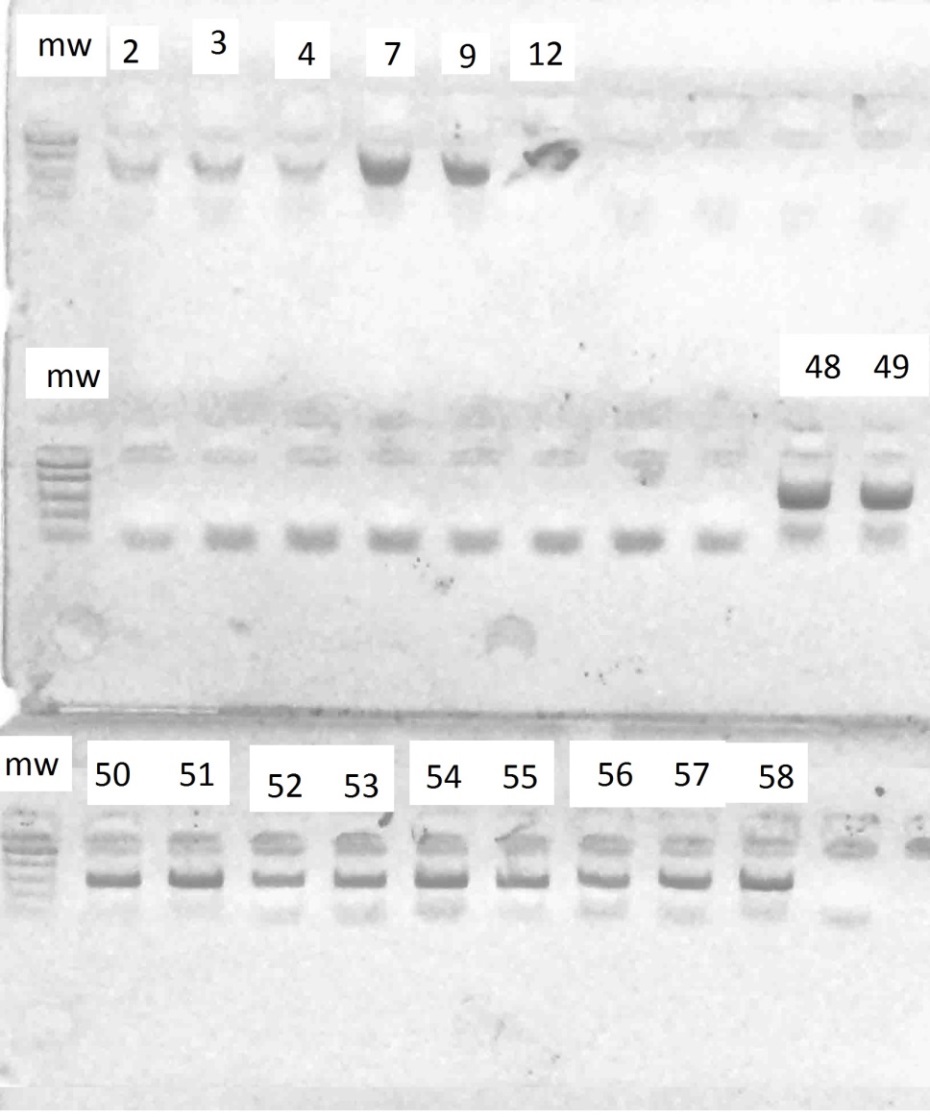
Рисунок 4 – Разделение продуктов амплификации ДНК аборигенных сортов
с праймерами VvMybA1_F и VvMybA1_R
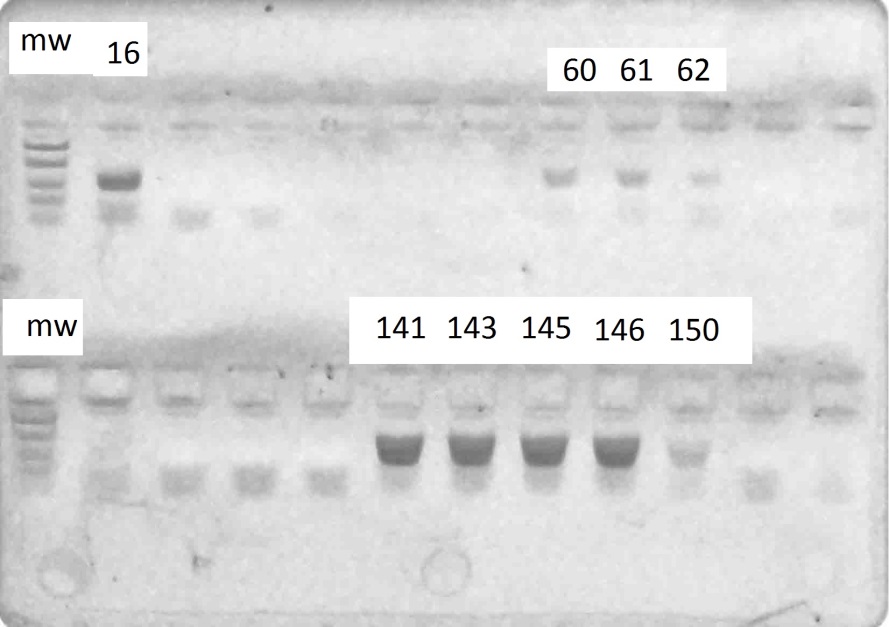
Рисунок 5 – Разделение продуктов амплификации ДНК аборигенных сортов
с праймерами VvMybA1_F и VvMybA1_R

Рисунок 6 – Разделение продуктов амплификации ДНК аборигенных сортов
с праймерами VvMybA1_F и VvMybA1_R
Стоит отметить, что выявленные аллели были визуально одинакового размера (примерно 800 п.н.) Такой результат является вполне ожидаемым, в связи с тем, что указанные маркеры дают возможность детектировать аллели как
у окрашенных, так и у неокрашенных сортов, однако последовательности нуклеотидов данных аллелей могут быть полиморфны. Поэтому, нами было проведено секвенирование с последующим выравниванием данных сиквенсов при помощи программы Clustal Omega с целью анализа их строения (рисунок 7).
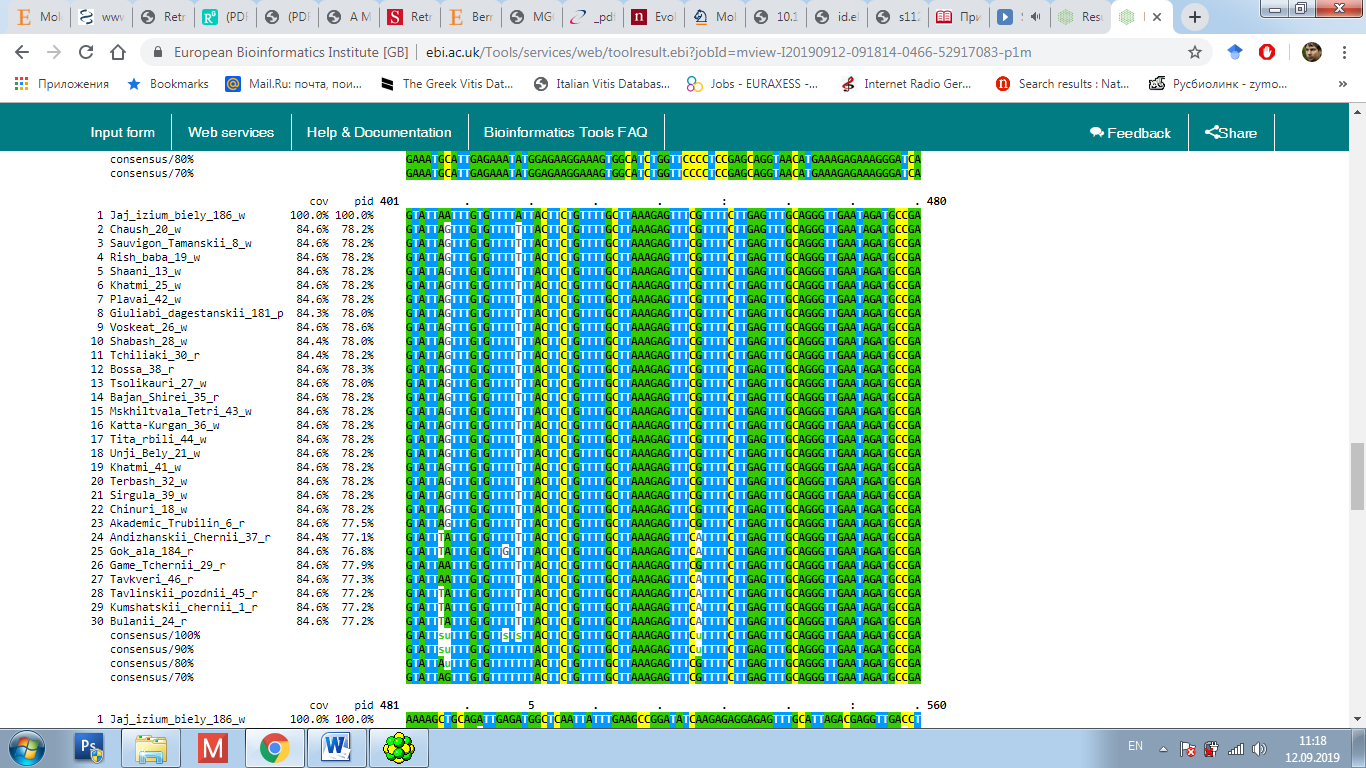
Рисунок 7 – Выравнивание нуклеотидных последовательностей
в программе Clustal Omega
Выравнивание данных последовательностей аллелей изученных сортов показало полиморфное строение у многих образцов (см. рисунок 7). При этом наиболее интересным результатом было обнаружение у генотипа Яй изюм белый вставки, длинной 110 п.н. Это интересно в виду того, что на данный момент известна функциональная аллель mybA1-SUB, имеющая вставку почти
в раза длиннее – 188 п.н. Предполагается, что данная вставка имеет схожее происхождение у сорта Яй изюм белый. Помимо этого, также предполагается, что она присутствует и у таких родственных генотипов как Яй изюм черный
и Яй изюм розовый. Помимо этого, в результате секвенирования у всех изученных генотипов были выявлены как уникальные, так и общие нуклеотидные замены. Наибольшее количество однонуклеотидных полиморфизмов было выявлено у сорта Яй изюм белый, при этом, качество сиквенса данного генотипа было достаточно высоким (рисунок 8).
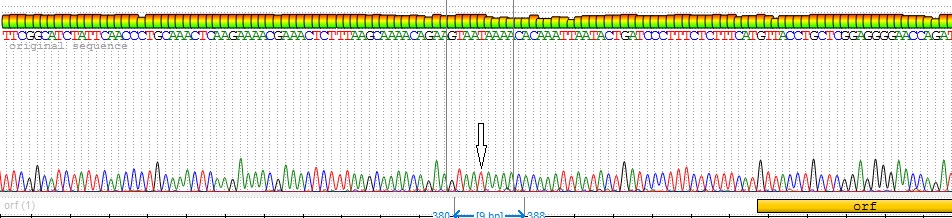
Рисунок 8 – Нуклеотидная замена, выявленная у сорта Яй изюм белый
Как один из примеров, на рисунке 8 мы можем видеть нуклеотидную замену у сорта Яй изюм белый в 418 нуклеотидной позиции. При этом, у всех остальных сортов встречается только один нуклеотид, т. е. данная мутация является уникальной для изученной выборки аборигенных сортов.
Для того, чтобы проверить имеются ли еще генотипы и, соответственно, аллели с такими же нуклеотидными заменами и насколько высока схожесть
у изученных нами сортов с уже описанными, мы провели поиск в базе данных NCBI (см. таблицу 3). В целом, итоги, полученные при проверке совпадения
в базе данных NCBI, дали вполне ожидаемый результат: наличие экспрессируемой аллели у красноягодных сортов и не функциональной у белоягодных (см. таблицу 3). Тем не менее, в виду того, что нами использовалось секвенирование с использованием прямого и обратного праймеров, у некоторых сортов было выявлено гетерозиготное состояние локуса. Это не является особенностью, так как подобные случаи уже были известны ранее. Тем не менее, были выявлены как процентные, так и качественные различия у изученных генотипов, в частности, у таких сортов как: Яй изюм белый, Гюляби дагестанский, Цоликаури, Босса, Академик Трубилин, Гамэ черный, Гок ала и Тавквери, в сравнении
с референсными (см. таблицу 3).
Как показано в таблице 3, у некоторых сортов было выявлено наличие различий в строении изученных аллелей при сравнении с референсными сортами. Не смотря на это, при анализе результатов было уточнено, что обнаружены не столько различия между нуклеотидными последовательностями, сколько гетерогенность, очевидно вызванная одновременным наличием нескольких аллелей в генотипах и, соответственно, их сложным селекционным происхождением, которое до конца не установлено из-за того, что данные сорта являются стародавними.
Такая гетерогенность является крайне интересной для последующего изучения и, вполне возможно, станет одним из посылов к дальнейшему изучению истории этих аборигенных сортов и уточнению их происхождения.
Неожиданным же оказалось то, что у дагестанского аборигенного генотипа Яй изюм белый, который имеет неокрашенную ягоду, были найдены 100 % совпадения с сортами Сирх, Сира, Ягуты, Султанина Розада, а также с диким лесным виноградом Vitis vinifera subsp. silvestris. В свою очередь, это указывает на то, что данный аборигенный сорт имеет такую схожую функциональную аллель как MybA1-SUB, которая представлена в сортах с розовой ягодой: Султанина Розада и Кошу. Это же позволяет сделать вывод, что отсутствие окраски
у данного сорта вполне возможно может иметь иную причину, нежели мутация в гене VvMybA1. Помимо этого, такие полученные данные представляют возможность предположить близкое родство данного сорта с диким лесным виноградом. И, возможно, данный генотип унаследовал строение своей аллели именно от сеянцев, которые появились в результате случайного опыления с диким виноградом, достаточно распространенным в Кавказском регионе, тем более что происхождение сорта до сих пор точно неизвестно.
Аборигенный сорт Гюляби дагестанский, имеющий розовую окраску ягод, показал наличие в своем геноме, по-видимому, гетерозиготной аллели. Такой вывод заключается из того факта, что при сравнение с базой данных NCBI было выявлено совпадение его нуклеотидного сиквенса с аллелью сорта Бенитака, который является мутантным окрашенным клоном сорта Италия. Не смотря на это, стоит отметить, что совпадение было выявлено именно с геном, имеющим ретротранспозонную вставку, которая блокирует его нормальную экспрессию. В виду этого факта и того, что в отличие от Бенитаки, происхождение этого аборигенного сорта неизвестно, мы не можем точно утверждать, что в этом случае произошло возвращение окраски из неокрашенной формы. С другой стороны, интересным было также и обнаружение схожей аллели у таких сортов как Чиляки, Баян Ширей и Босса (неокрашенная ягода). Это, в свою очередь, также показывает их гетерогенность и предполагает наличие таких генов в родительских формах.
Происхождение сортов Академик Трубилин и Гамэ черный, а также их принадлежность к группе западно-европейских были подтверждены. Интересным же является то, что было выявлено абсолютное сходство между аллелями Гамэ черный и французского аборигенного сорта Гранд нуар, который имеет ярко выраженную антоцианиновую окраску ягод и вина.
Помимо этого, установлена схожесть строения аллелей аборигенного генотипа Гок ала с сортом Альфонс Лавалле, выведенным во Франции. В частности, наличие у него в генотипе аллели VvMybA1ALF. Данная аллель является функциональной, и поэтому оба сорта имеют интенсивную окраску ягод, и, следовательно, наличие гомозиготной функциональной аллели.
В остальном стоит отметить, что полученные результаты у других аборигенных генотипов соответствовали ожидаемым и подтверждаются фенотипическими проявлениями исследуемого признака. Чтобы проверить влияние строения аллелей на аминокислотную последовательность, мы провели сравнение при помощи алгоритмов blastx (таблица 4).
Таблица 4 – Результаты сравнения аминокислотных последовательностей
изученных сортов винограда
| Сорт | Результат | Источник |
| Яй изюм белый | truncated MybA1 [Vitis vinifera] | Lijavetzky et al., 2006 |
| Гюляби дагестанский |
myb-related transcription factor [Vitis vinifera] | Azuma et al., 2009 |
| Воскеат | myb-related transcription factor [Vitis vinifera] | Azuma et al., 2009 |
| Чиляки | myb-related transcription factor [Vitis vinifera] | Azuma et al., 2009 |
| Шабаш | myb-related transcription factor [Vitis vinifera] | Azuma et al., 2009 |
| Цоликаури | myb-related transcription factor [Vitis vinifera] | Azuma et al., 2009 |
| Чауш | myb-related transcription factor [Vitis vinifera] | Azuma et al., 2009 |
| Совиньон Таманский |
myb-related transcription factor [Vitis vinifera] | Azuma et al., 2009 |
| Риш баба | myb-related transcription factor [Vitis vinifera] | Azuma et al., 2009 |
| Шаани | myb-related transcription factor [Vitis vinifera] | Azuma et al., 2009 |
| Хатми | myb-related transcription factor [Vitis vinifera] | Azuma et al., 2009 |
| Плавай | myb-related transcription factor [Vitis vinifera] | Azuma et al., 2009 |
| Баян Ширей | myb-related transcription factor [Vitis vinifera] | Azuma et al., 2009 |
| Мсихлтвала Тетри |
myb-related transcription factor [Vitis vinifera] | Azuma et al., 2009 |
| Катта-Курган | myb-related transcription factor [Vitis vinifera] | Azuma et al., 2009 |
| Тита Рбили | myb-related transcription factor [Vitis vinifera] | Azuma et al., 2009 |
| Уни белый | myb-related transcription factor [Vitis vinifera] | Azuma et al., 2009 |
| Хатми | myb-related transcription factor [Vitis vinifera] | Azuma et al., 2009 |
| Тербаш | myb-related transcription factor [Vitis vinifera] | Azuma et al., 2009 |
| Сиргула | myb-related transcription factor [Vitis vinifera] | Azuma et al., 2009 |
| Чинури | myb-related transcription factor [Vitis vinifera] | Azuma et al., 2009 |
| Босса | truncated MybA1 [Vitis vinifera] | Lijavetzky et al., 2006 |
| Академик Трубилин |
truncated MybA1 [Vitis vinifera] | Lijavetzky et al., 2006 |
| Гамэ черный | truncated MybA1 [Vitis vinifera] | Lijavetzky et al., 2006 |
| Гок ала | MybA1 [Vitis vinifera] | Lijavetzky et al., 2006 |
| Тавквери | truncated MybA1 [Vitis vinifera] | Lijavetzky et al., 2006 |
| Андижанский черный |
truncated MybA1 [Vitis vinifera] | Lijavetzky et al., 2006 |
| Тавлинский поздний |
truncated MybA1 [Vitis vinifera] | Lijavetzky et al., 2006 |
| Кумшацкий черный |
truncated MybA1 [Vitis vinifera] | Lijavetzky et al., 2006 |
| Буланый | truncated MybA1 [Vitis vinifera] | Lijavetzky et al., 2006 |
Таким образом, результаты показывают нам распределение выявленных аллелей на три основные группы по аминокислотным последовательностям. Но все исследованные сиквенсы показали 100 % схожесть с уже внесенными в базы данных NCBI (см. таблицу 4). Помимо этого, данные аминокислотные последовательности консервативны не только для сортов, но и при межвидовом сравнении. Так, например, сорт Гюляби дагестанский показал совпадение как
с видом Vitis hancockii, так и с видом Vitis pseudoreticulata. Из этого очевидно, что данные виды не могут быть родственными данному сорту в виду их отдаленности по географическому признаку. В целом, сорта показали схожесть
с разными образцами, и, очевидно, имеют различия между собой в аминокислотной последовательности, как это уже было показано для других сортов. Поэтому, для того чтобы выявить особенности их строения, мы провели их выравнивание в ClustalO (рисунок 9).
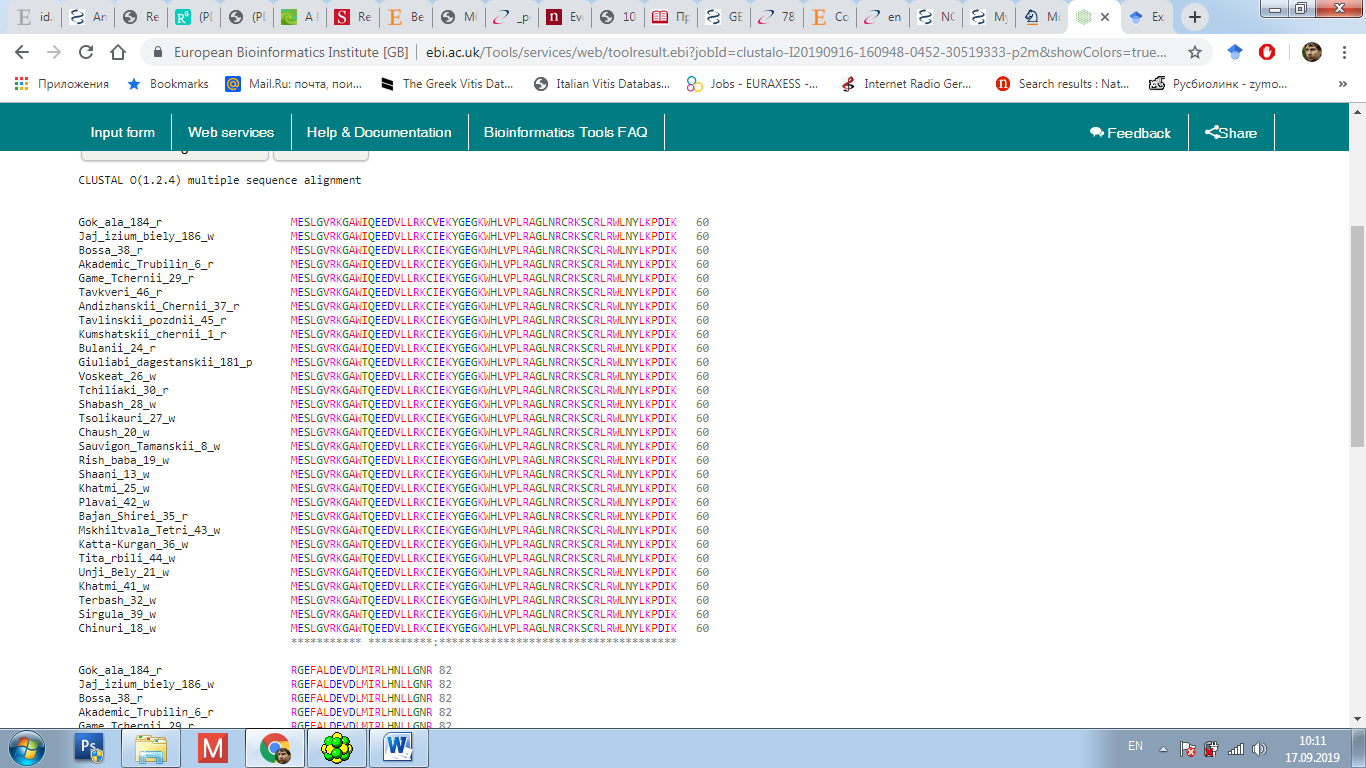
Рисунок 9 – Сравнение аминокислотных последовательностей
аборигенных сортов
Как уже было сказано, изученные сорта сформировали три основных кластера по степени схожести аминокислотного состава. И в результате их выравнивания было выявлено, что в двух позициях (12 и 23) имеются замены.
В частности в 23 – триптофан на изолейцин, в то время как в 23 – изолейцин на валин. При этом, такая замена в нашем исследовании была уникальной и выявлена только у сорта Гок ала. Тем не менее, известно, что данные аминокислоты обладают схожими свойствами и, в виду окраски ягод данного генотипа, по-видимому, данная замена не повлияла на проявление признака.
С другой стороны, интересно отметить корреляцию в большинстве случаев между изменениями аминокислоты под номером 12 и окраской ягод. Как можно увидеть из рисунка 9, большинство сортов, обладающих красной ягодой, имеют в этой позиции изолейцин, в то время как неокрашенные сорта – триптофан. При этом видно, что ClustalO указывает на неравнозначность замены данных аминокислот. При этом, указанные ранее результаты предполагают наличие гетерозиготного состояния у некоторых окрашенных сортов.
Для изучения филогенетических и проверки результатов взаимоотношений, по данным исследованиям аллелей гена VvMybA1 мы провели кластерный анализ изученных генотипов аборигенных сортов винограда с использованием, как ДНК, так и аминокислотных последовательностей для сравнения результатов между собой (рисунок 10).
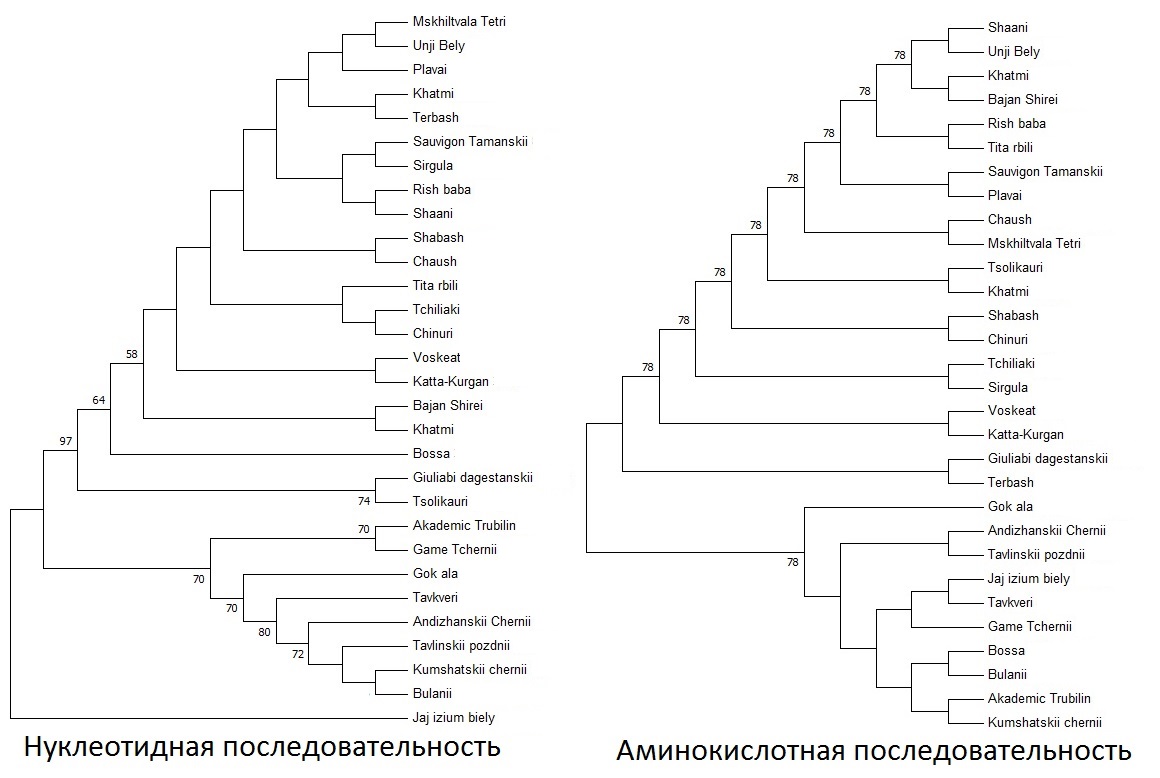
Рисунок 10 – UPGMA кластеризации основанные на нуклеотидных
и аминокислотных последовательностях
Сравнение двух UPGMA кластеризаций, основанных на нуклеотидных
и аминокислотных сиквенсах, выявило, в целом, схожее распределение генотипов. Большинство образцов распределилось между кластерами окрашенных
и неокрашенных генотипов в соответствии с их окраской. Аборигенные сорта, такие как Баян Ширей и Чиляки, имеющие окраску ягод, вошли в кластер неокрашенных сортов, но, как говорилось ранее, это вызвано гетерозиготностью данных сортов.
Однако некоторые сорта не имели строгой привязки к какому-то определенному микро-кластеру. Например, сорт Яй изюм белый был выделен программой в отдельный кластер по результатам анализа ДНК последовательности, в том время как по результатам сиквенса аминокислот он вошел в кластер красноягодных сортов. С одной стороны, это объяснимо наличием уникальной вставки (которая не была обнаружена ни у одного из генотипов), с другой стороны, результат также интересен тем, что сорт обладает белой ягодой, в то время как аминокислотная последовательность его характерна для окрашенных сортов. Таким образом, данный результат поддерживает теорию о том, что потеря окраски у данного сорта имеет другую причину, нежели мутация гена VvMybA1.
Как было установлено, сорт Босса, имеющий интенсивную окраску ягод, по результатам анализа ДНК последовательностей был включен в кластер неокрашенных сортов, в то время как результаты анализа аминокислотного сиквенса были совершенно иными. Данный результат объясним тем, что, не смотря на изменения в строении последовательности нуклеотидов, аминокислоты не были изменены. Однако стоит заметить, что по результатам поиска
с применением NCBI BLAST, данный сорт должен иметь вставку ретротранспозона Gret-1, блокирующую его нормальное функционирование. Этот факт предполагает по себе, что данный сорт народной селекции вполне может оказаться генотипом-ревертантом и, следовательно, был отобран как мутант, произошедший вследствие каких-то вегетативных изменений. В итоге данный сорт, сохранил строение аллели, схожее с изначальной, блокированной мобильным ДНК-элементом.
Таким образом, генетическое разнообразие исследованных сортов представлено несколькими типами известными ранее аллелей: VvMybA1a, VvMybA1c, VvMybA1AFL, VvMybA1SUB и VvMybA1BEN. При этом выявлено, что некоторые сорта гетерозиготны и, очевидно, имеют как функциональную, так
и не экспрессируемую аллели гена VvMybA1. Помимо этого, обнаружены различия в строении генов, которые позволяли, как идентифицировать уникальные генотипы, так и выявить общие области в их строении. Дальнейший анализ при помощи трансляции нуклеотидной в аминокислотную последовательность подтвердил вышеизложенные гипотезы и показал, что имеется и уникальная для данной выборки аминокислотная замена. Таким образом, в целом, аминокислотные сиквенсы имели строение, характерное для групп сортов, которые они представляют по своему фенотипу. Что, в итоге, также было подтверждено
и кластерным анализом аборигенных сортов.
Наиболее интересным результатом исследований было обнаружение у сорта Яй изюм белый 110 п.н. вставки, характерной для аллели mybA1-SUB, которая является функциональной. Тем не менее, так как сорт не имеет выраженной яркой окраски ягод, это дает возможность сделать предположение, что причина отсутствия такого проявления данного признака другая, например, однонуклеотидная мутация в гене VvMybA2. Помимо этого, интересным может оказаться
и то, что такая вставка присутствует в строении аллелей гена антоциановой окраски ягод у близкородственных генотипов Яй изюм черный и Яй изюм розовый, что, конечно же, требует дополнительного изучения. Более того, при сравнении аллелей всех изученных генотипов между собой, у сорта Яй изюм белый выявлено уникальное строение аллели, в связи с тем, что у него было обнаружено наибольшее количество однонуклеотидных замен при сравнении
с другими формами. Тем не менее, строение его аминокислотной последовательности, экспрессируемой геном совпадало с другими сортами, таким образом, наличие нуклеотидных замен, никак не повлияло на аминокислотное строение. По результатам сравнения в ClustalO и MEGA7 данный генотип вошел
в кластер окрашенных сортов, что дает возможность предполагать о иной причине отсутствия окраски ягод сорта (не изменения в гене VvMybA1) и о его родстве с сортами Яй изюм черный и Яй изюм розовый. В то время как открытым же вопросом остается генеалогия этих трех сортов.
Также, мы провели изучение таких известных сортов как Сибирьковый, Шардоне, Сыпун черный и Каберне Кортис. После разделения продуктов амплификации в агарозном геле была визуализированы результаты, которые показали, что все четыре испытуемых образца (два имеющих окраску ягод и два не имеющих) имеют одинаковые по размеру аллели – примерно 850 п.н. Однако данный результат не является уникальным, так как в аналогичных работах, уже была продемонстрирована схожесть изученных ДНК последовательностей, полученных в результате ПЦР, не только по размеру, но и по строению аллелей. Тем не менее, было показано, что одинаковые по размеру области могут быть полиморфны и различаться по структуре, которая влияет и на сам признак окраски. Поэтому, для установления структуры одинаковых по размеру последовательностей аллелей нами было проведено их секвенирование. После чего, участки были сравнены друг с другом в программе ClustalO (рисунок 11) для анализа их полного строения.

Рисунок 11 – Анализ участков изученных аллелей гена VvMybA1
в программе ClustalO
На рисунке 11 демонстрируются некоторые однонуклеотидные полиморфизмы, выявленные по результатам анализа. В итоге, в результате уравнения последовательностей, мы можем видеть, что присутствуют нуклеотидные замены отличающие между собой, в первую очередь, бело- и красноягодные сорта, например 4–10 нуклеотидные позиции. Помимо этого, результаты подтверждаются и методом поиска среди последовательностей, имеющихся в базе данных NCBI, так как при сравнении с ними была выявлена 100 % схожесть, за исключением аборигенного сорта Сыпун черный.
Таким образом, ДНК последовательности алелей выявленные у сортов Шардоне и Сибирьковый оказались идентичными и показали совпадение с аллелью, характерной для сортов, не имеющих окраску ягод. Эти же сорта имеют вставку транспозона Gret-1, который блокирует его работу. Таким образом, мы можем сделать вывод, что именно наличие такой транспозонной вставки в данном гене блокирует экспрессию гена, отвечающего за антоциановый окрас ягод у сорта Сибирьковый.
Помимо этого, не смотря на то, что у сорта Сыпун черный в 290 нуклеотиде была выявлена уникальная мутация, в целом, строение его аллели (так же как и у сорта Каберне Кортис) характерно для окрашенных сортов (в частности: KY406230.1, GU145121.1 и GU145120.1). Интересным же фактом является то, что схожесть выявлена конкретно с аллелью VvMybA1SUB, которая, в свою очередь, иногда может быть выявлена и не в экспрессируемом состоянии у таких неокрашенных сортов и клонов как Султанина, Пировано 166А и др., а, следовательно, не является здесь играющей основную роль в формировании окраски ягод.
После установления принадлежности сиквенсов к искомым аллелям, мы произвели поиск уже среди аминокислотных последовательностей, используя алгоритмы blastx. Таким образом, для всех четырех ДНК-сиквенсов была выявлена предположительная аминокислотной последовательность (рисунок 12).
Результаты поиска аминокислотных последовательностей с использованием blastx показали полное совпадение строения у всех анализируемых образцов за исключением крупной вставки в середине сиквенса, которая является трансляцией интронной последовательности в аминокислоты. По какой-то причине она не была исключена системой.

Рисунок 12 – Результаты поиска аминокислотных последовательностей
при помощи алгоритмов blastx
Посел выявления однонуклеотидных полиморфизмов, мы произвели выравнивание белковых последовательностей в ClustalO (рисунок 13), для поиска различий и в их строении.
Рисунок 13 раскрывает общие и уникальные особенности строения предполагаемых аминокислотных сиквенсов. Общие различия между белоягодными и красноягодными сортами были обнаружены в 12, 49 и 62 позициях аминокислот. Как мы видим, уникальная замена изолейцина на валин была выявлена
у сорта Сыпун черный в 23 позиции сиквенса.

Рисунок 13 – Уравнение протеиновых последовательностей
с интронной вставкой (выделена другим цветом)
Помимо этого важным будет заметить, что два из четырех полиморфизмов располагаются именно в кодирующей части. При этом один из них характерен для бело- и красноягодных генотипов, в то время как 23 аминокислота у сорта Сыпун черный, в данном исследовании, является уникальной. В частности потому, что, согласно CD-search, она располагается в консервативном участке. Также, интересным является и тот факт, что при поиске схожих последовательностей в данной позиции обнаружено сходство с сортом Альфонс Лавалле. Оба сорта являются ярко окрашенными, а, следовательно, изменение такой аминокислотной последовательности никак не влияет на экспрессию данного признака. Примечательно, что поиск данного сиквенса в NCBI выявил, его встречаемость у межвидовых гибридов, которые также имеют схожую структуру своих генов, но отличаются от изучаемого генотипа заменой валина на изолейцин, также как и при сравнении с изученными генотипами.
В свою очередь, неожиданным же оказалась схожесть строения аминокислотной последовательности сорта Сибирьковый с такими окрашенными сортами, такими как Бенитака, Каберне Совиньон и Родитис. Тем не менее, это может быть объяснимо тем, что, например, сорт Бенитака является спонтанным мутантом от белоягодного сорта Италия, сорт Каберне Совиньон – это гибрид, несущий обе аллели, так же как и сорт Родитис.
Дабы провести сравнительный анализ последовательностей генов обуславливающих окрашенные и неокрашенные ягоды, мы произвели их кластеризацию с использованием аминокислотных последовательностей. При этом мы ввели дополнительно генотип (Rosa chinensis, XP_024179665.1) из семейства Розоцветных для создания аут-группы (рисунок 14).
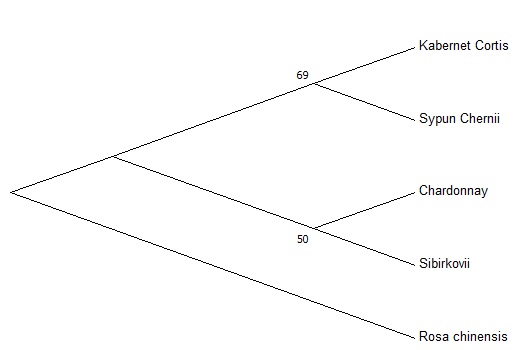
Рисунок 14 – Кластеризация изученных генотипов с использованием
аминокислотных последовательностей (показаны бутстрепы 50 и выше)
При кластеризации, как и ожидалось, генотипы разделились на три группы: аут-группу, окрашенные и неокрашенные сорта. При этом интересным было то, что программа сформировала древо именно таким образом, что белоягодные сорта отделились от основной ветви: располагаются между окрашенными сортами и аут-группой. Эти данные подтверждаются также и другими исследованиями. Так ведь и в самом деле, некоторые сорта с неокрашенной ягодой появились в результате спонтанной мутации, как например, Пино блан.
В это же время существуют также и сорта-ревертанты, например, сорт Бенитака у которого было зафиксировано обратное возвращение окраски, т. е. обратный переход от белого цвета ягод к розовому.
Впервые у некоторых аборигенных сортов России нами были идентифицированы аллели гена VvMybA1. Выравнивание последовательностей показало наличие характерных особенностей строения аллелей для каждого из изученных сортов. Были выявлены уникальные однонуклеотидные замены присущие конкретным генотипам с окрашенной и неокрашенной ягодой. Поиск полученных в результате секвенирования последовательностей в NCBI посредством алгоритмов BLAST показал, что, в целом, все генотипы имеют аллели характерные для бело- и черноягодных сортов.
3.2 Изучение аллелей гена VvMybA1 у клоновых форм распространенных сортов винограда
В результате работы были выявлены аллели (рисунки 15 и 16) и изучено строение последовательностей аллелей гена VvMybA1 у клонов, принадлежащих к распространенным сортотипам винограда. Для определения принадлежности генов к определенным аллелям, а также установления особенностей их строения, мы провели сравнение с уже известными генотипами в базе данных NCBI.
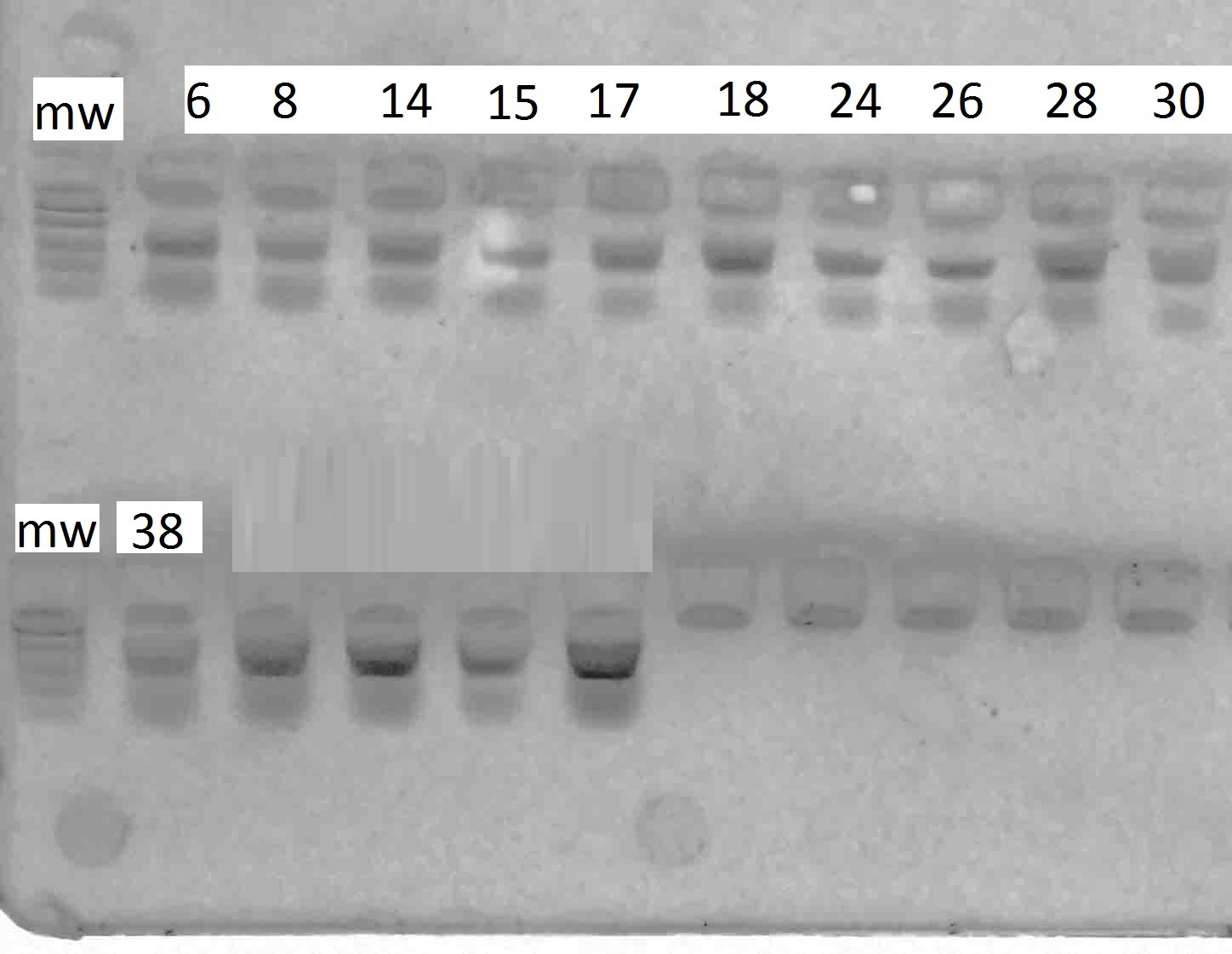
Рисунок 15 – Разделение продуктов амплификации праймеров с сортоклонами
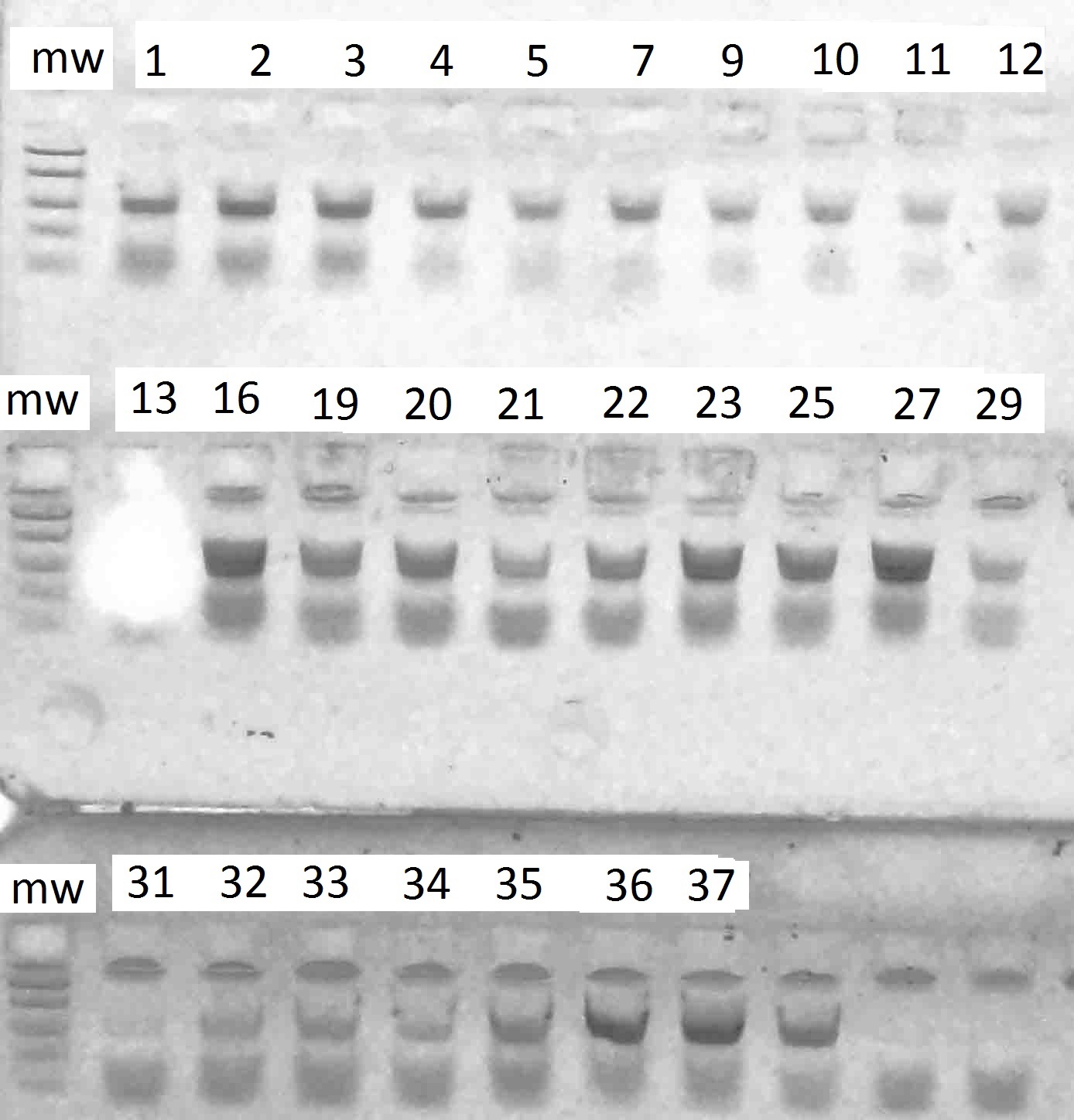
Рисунок 16 – Разделение продуктов амплификации праймеров с сортоклонами
Таким образом, по результатам анализа было выявлено, что сорта имеют аллели, характерные для красноягодных и белоягодных генотипов. В целом
и общем, для клоновых генотипов, относящихся к сортам Рислинг, Пино белый, Совиньон белый и Алиготе, не было обнаружено изменений или какой-либо гетерогенности в строении их аллелей, а последовательности их были характерны для белоягодных сортов. В то же время в сиквенсах таких клонов сортов как Каберне Совиньон и Мерло были выявлены как изменения, так и наличие гетерогенности (рисунок 17).
Как мы можем видеть у некоторых изученных генотипов имеются отличия, выраженные в одно- и динуклеотидных заменах (см. рисунок 17). При этом, интересно, что такие отличия показали именно парные клоны. Например, Каберне Совиньон 15КМ, что повышает вероятность достоверности. Помимо этого, интересно также и отметить, что клон Мерло 14МГ также показал наличие замен, схожих с другими красноягодными генотипами, имеющими такие же мутации. Таким образом, мы провели поиск подобных мест нуклеотидного полиморфизма в сиквенах, при помощи программы Unipro UGENE (рисунки 18 и 19).
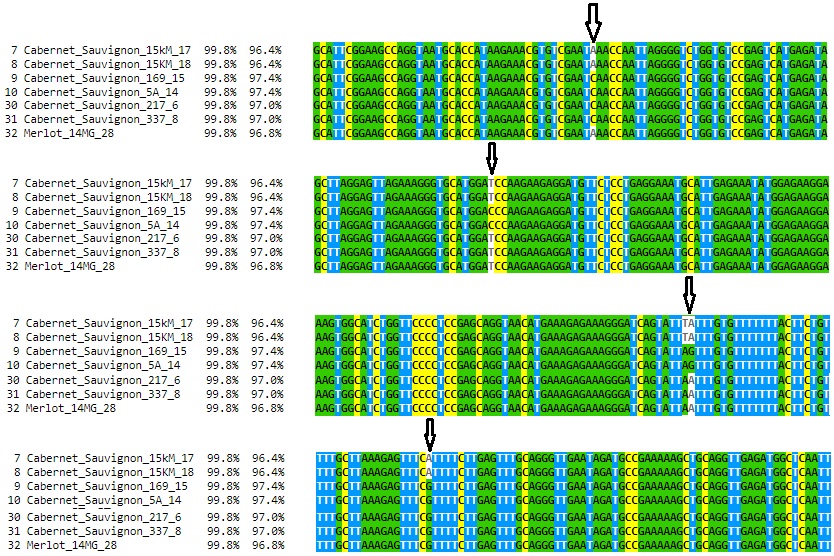
Рисунок 17 – Сравнение последовательностей изученных генотипов
с выявленными различиями в программе ClustalO
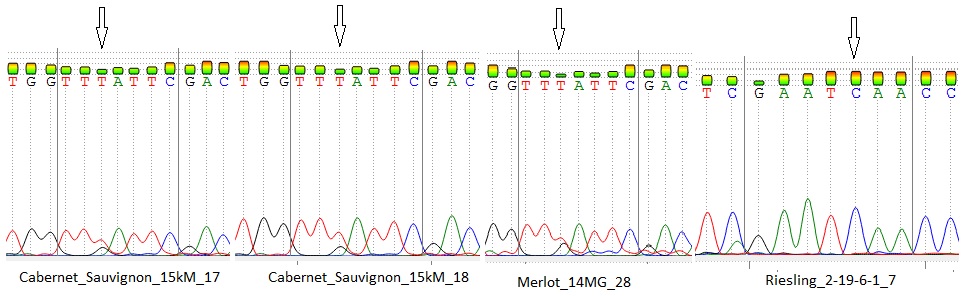
Рисунок 18 – Сравнение последовательностей окрашенных и неокрашенных клоновых генотипов
Как можно увидеть из рисунка 18, изученные окрашенные генотипы имеют отличия в строении последовательности их аллелей. И самое основное из таких отличий заключается в «двоении пиков» при секвенировании аллелей. Это говорит нам о наличии одновременно двух аллелей в генотипе, которые,
в свою очередь, характерны для окрашенных и неокрашенных сортов. Такой вывод мы можем сделать при поиске подобного участка у сорта Рислинг, где нуклеотид аденин заменен на цитозин, в то время как окрашенные сорта, имеют обе аллели, что и выражается в «раздвоении пиков», характеризующих нуклеотиды.
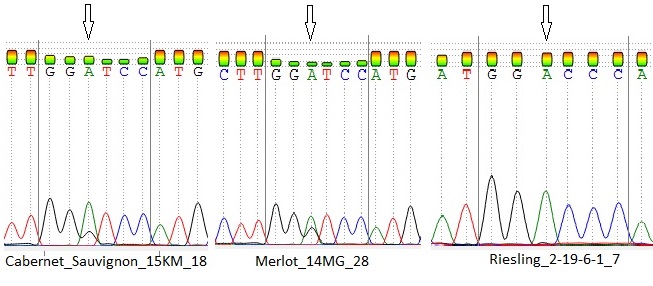
Рисунок 19 – Нуклеотидные замены, выявленные между окрашенными
и неокрашенными сортоклонами
Помимо этого, при секвенировании был выявлен еще один сайт двоения последовательности, отмеченный стрелкой на рисунке. Но, также интересным является и то, что имеется замена и последующего нуклеотида: у окрашенных сортов – тимин, в то время как у неокрашенных – цитозин. Следует также отметить, что такое же различия были выявлены и в 298, 299 и 398 нуклеотидных позициях, где было обнаружено раздвоение сиквенса. Также, у клонов сорта Каберне Совиньон было выявлено наличие аллели имеющей ретротранспозонную вставку, характерную для сорта Совиньон блан и ответственную за экспресии этой аллели и, следовательно, окраски. В свою очередь, очевидно, что она могла быть получена им от одного из своих предков и, поэтому, была обнаружена путем секвенирования, которое показало «двоение» некоторых нуклеотидов в ДНК-последовательности.
Таким образом, проведенные исследования показывают, что изученные нами клоновые генотипы имеют аллели, характерные для этих групп сортов, как обладающих, так и не обладающих окраской ягод. В то же время, исследованные сорта имеют отличия, которые, в свою очередь, характерны для разных агробиологических и эколого-географических групп. Например, для всех белоягодных сортов выявлена схожесть с сортами и клонами, изученными ранее, что говорит об их возможности использования в дальнейшей сравнительной геномике. То же самое можно сказать и об окрашенных сортах, в связи с тем, что у них было подтверждено наличие искомых аллелей, позволяющих иметь антоциановую окраску ягод. Не смотря на это, для поиска различий между клонами, конечно же, очевидным будет рекомендовать использование более вариабельных участков.
Если же говорить о генах, контролирующих антоциановый окрас ягод, то наиболее вероятным представляется промотерная последовательность гена VvMybA2, в котором было выявлено девятнадцать полиморфных сайтов. Поэтому, мы можем заключить, что, хотя клоны и не имеют между собой отличий в изученных генах, но обладают аллелями, характерными для сортов имеющих и не имеющих окраску. Это, в свою очередь, позволяет использовать их в дальнейших исследованиях как референсные сорта при сравнении с другими неизвестными сортами.
3.3 Изучение аллелей гена VvMybA1 у родственных клоновых форм новых сортов винограда
С целью изучения сортов новой селекции мы провели амплификацию праймеров с ДНК новых сортогрупп. В результате работы были выявлены аллели (рисунок 20) и изучено строение последовательностей аллелей гена VvMybA1 у клонов сортов, принадлежащих к новым сортотипам. Для определения принадлежности генов к определенным аллелям и установления особенностей строения их последовательностей, мы провели сравнение с уже известными генотипами в базе данных NCBI.
Как видно из рисунка 20, все выявленные аллели обладали схожими размерами в п.н. Данное положение может быть объяснено несколькими причинами: 1) работа маркеров, рассчитанных на одну и ту же область; 2) отсутствие вставок в изученных аллелях; и 3) близкое родство некоторых из генотипов.
Поэтому, для того чтобы провести изучение строения аллелей и выявить их особенности было проведено секвенирование и выравнивание в программе Clustal Omega (рисунок 21).
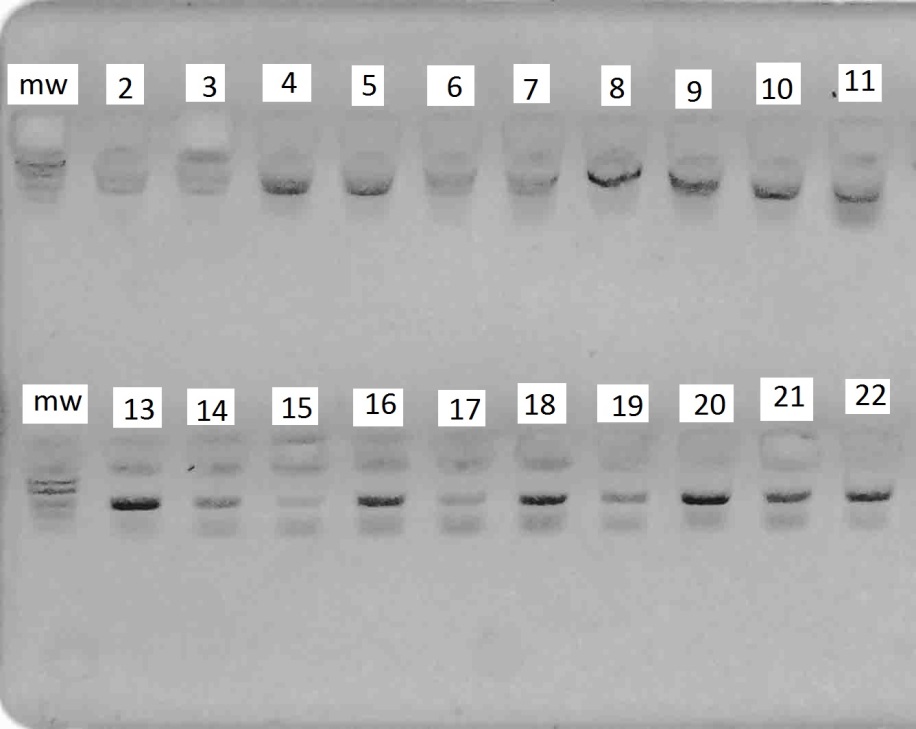
Рисунок 20 – Результаты амплификации ДНК сортов новой селекции
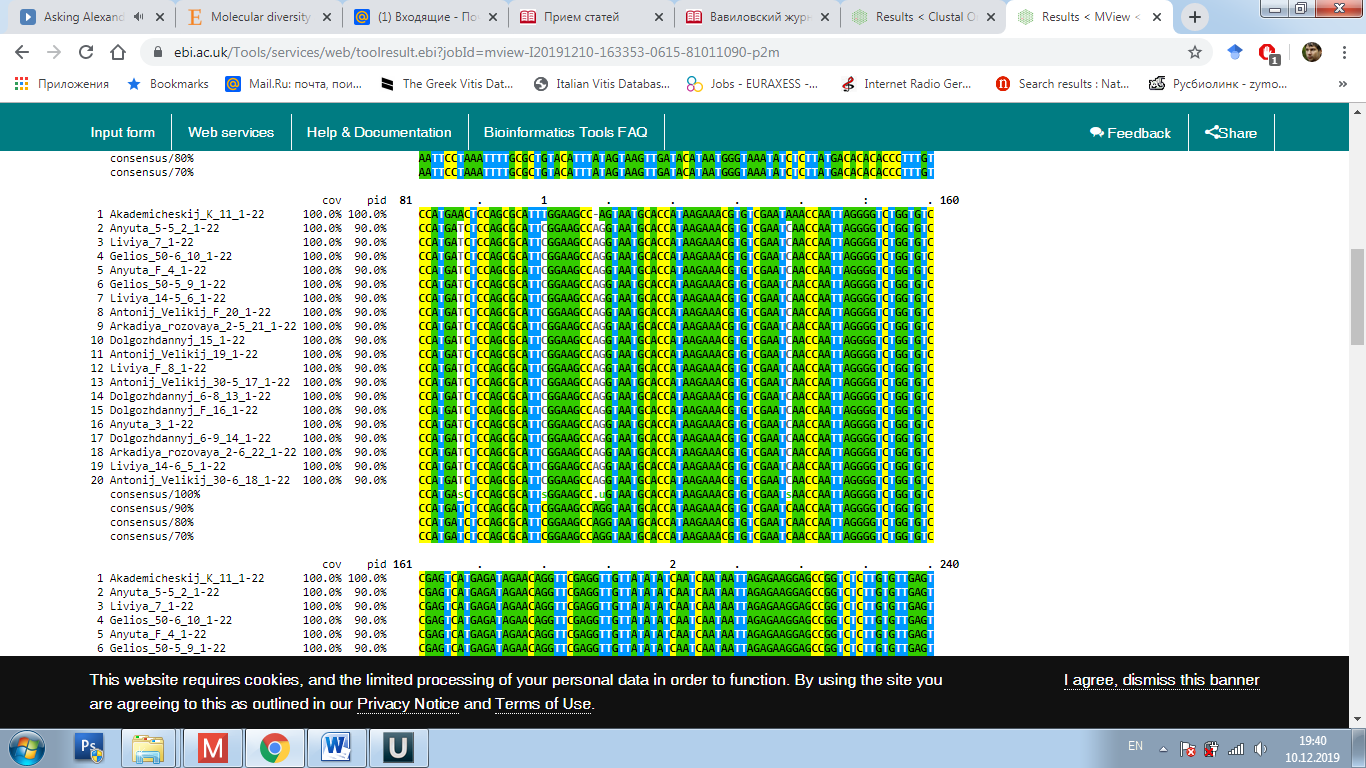
Рисунок 21 – Выявление схожих последовательностей среди сиквенсов
изученных аллелей
Как мы можем видеть, в большинстве своем изученные аллели были похожи друг на друга за исключением сорта Академический. Также, в отличие от остальных изученных генотипов, у сорта Академический была выявлена более короткая аллель в сравнении с другими образцами (рисунок 22).
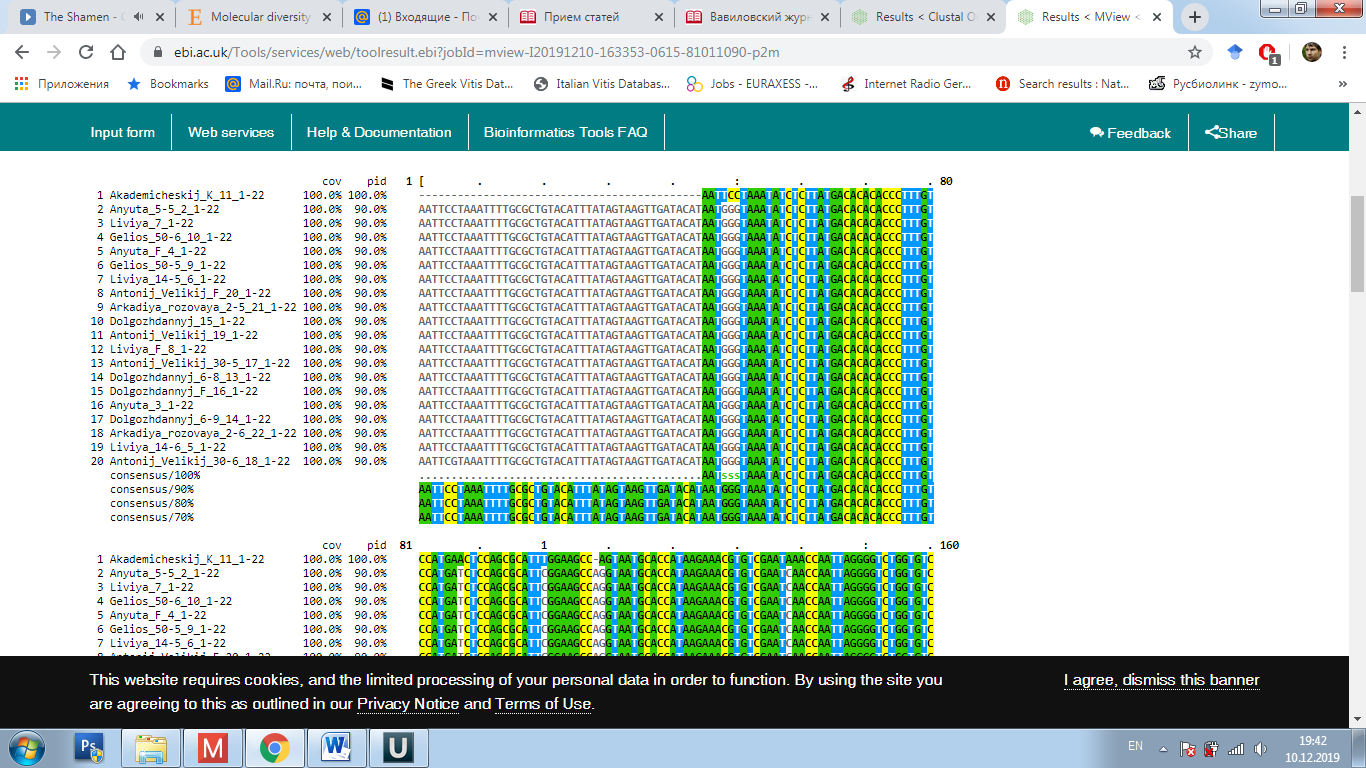
Рисунок 22 – Укороченная аллель генотипа Академический
Тем не менее, чтобы произвести сравнение образцов между собой, нами была произведена кластеризация изученных генотипов. Как мы видим, близкородственные сорта не отличались друг от друга. Тем не менее, это: 1) подтверждает их происхождение; 2) говорит о высоком качестве полученных последовательностей. В свою очередь, сравнение полученных последовательностей
с базой данных NCBI выявило, что изучаемые результаты были ожидаемыми. Таким образом, нами были получены сиквенсы близкородственных сортов новой селекции, которые могут использоваться в дальнейшем как основа для сравнения с сортами, выведенными с использованием таких генотипов как: Талисман, Кишмиш лучистый, Аркадия и Кишмиш находка.
3.4 Изучение строения гена VvMybA2 у сортов винограда
С целью изучения сортов новой селекции мы провели амплификацию праймеров с ДНК аборигенных сортогрупп. В результате работы были выявлены аллели (рисунки 23–29) и изучено строение последовательностей аллелей гена VvMybA2 у аборигенных сортов, принадлежащих к различным сортотипам. Для определения принадлежности генов к определенным аллелям и установления особенностей строения их последовательностей, мы провели сравнение с уже известными генотипами в базе данных NCBI.
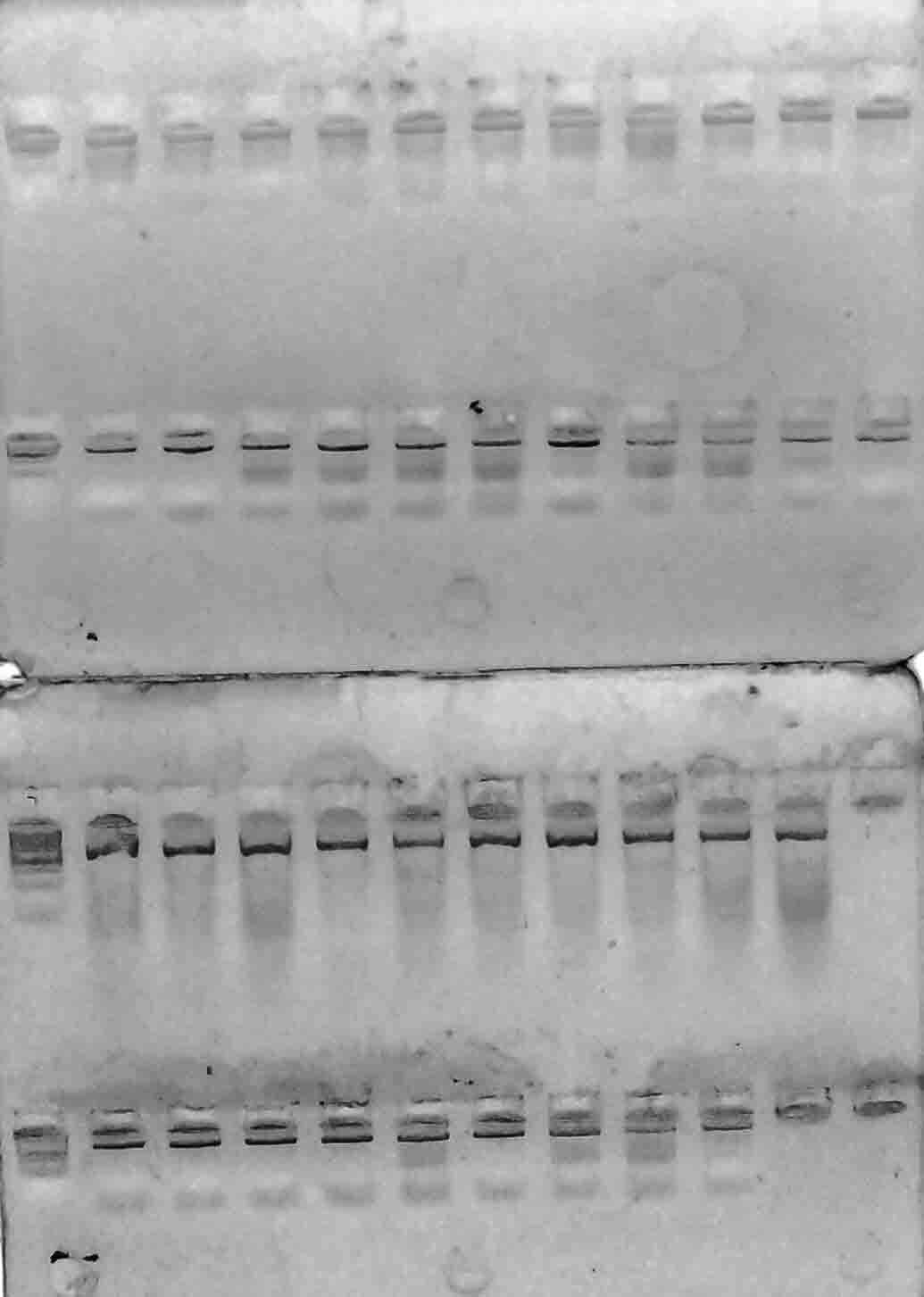
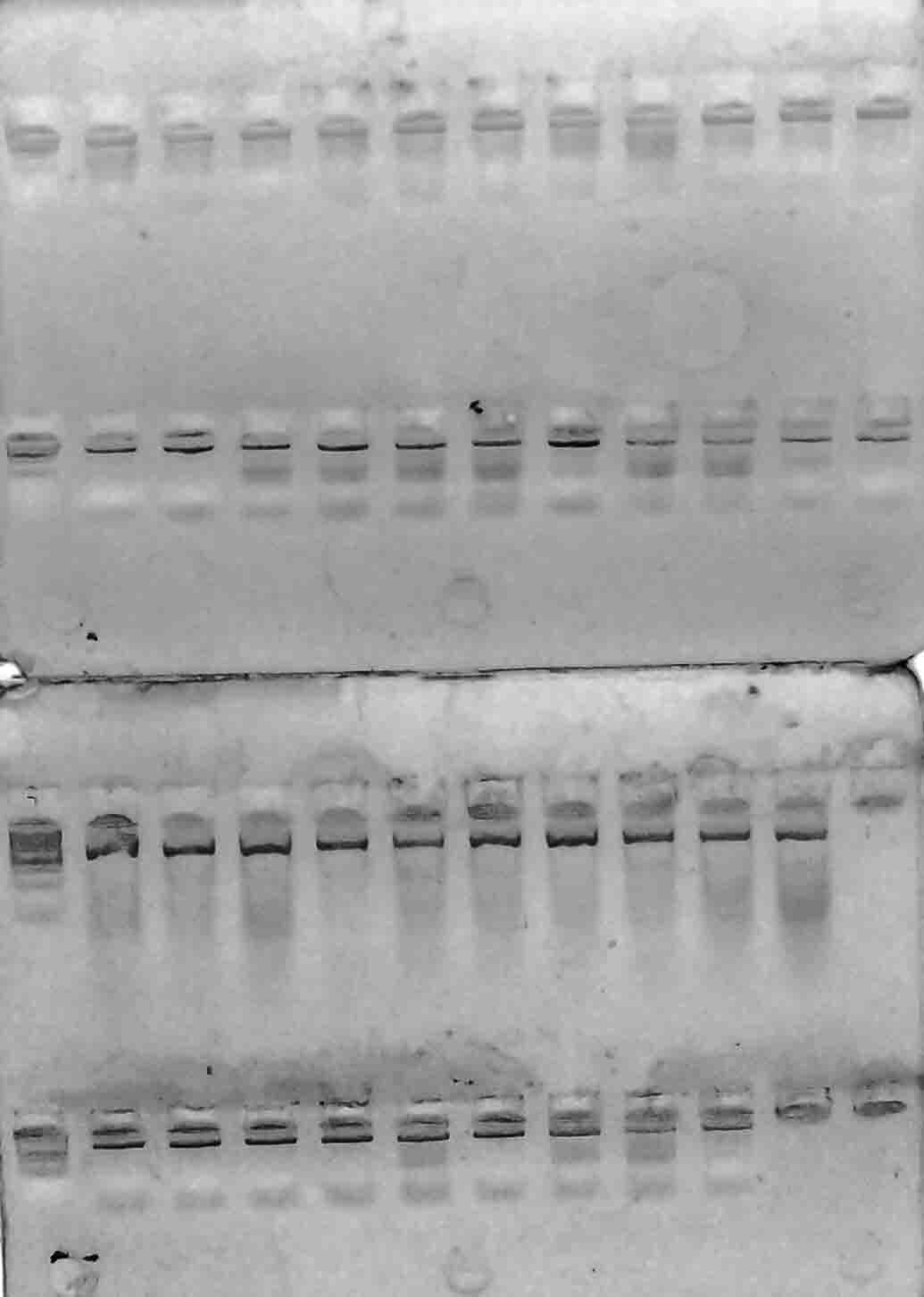
Рисунок 23 – Разделение продуктов амплификации аллелей гена VvMybA2
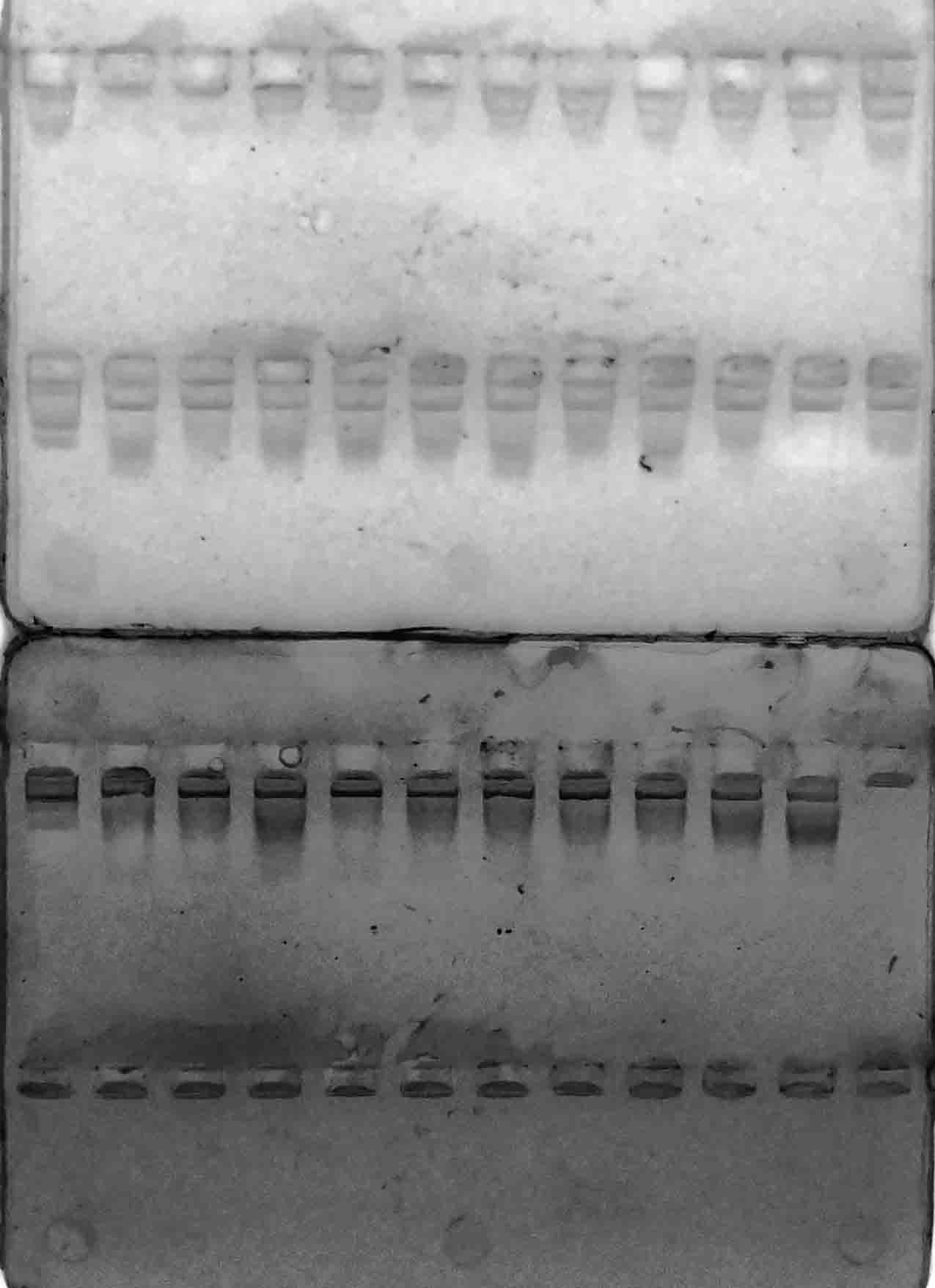
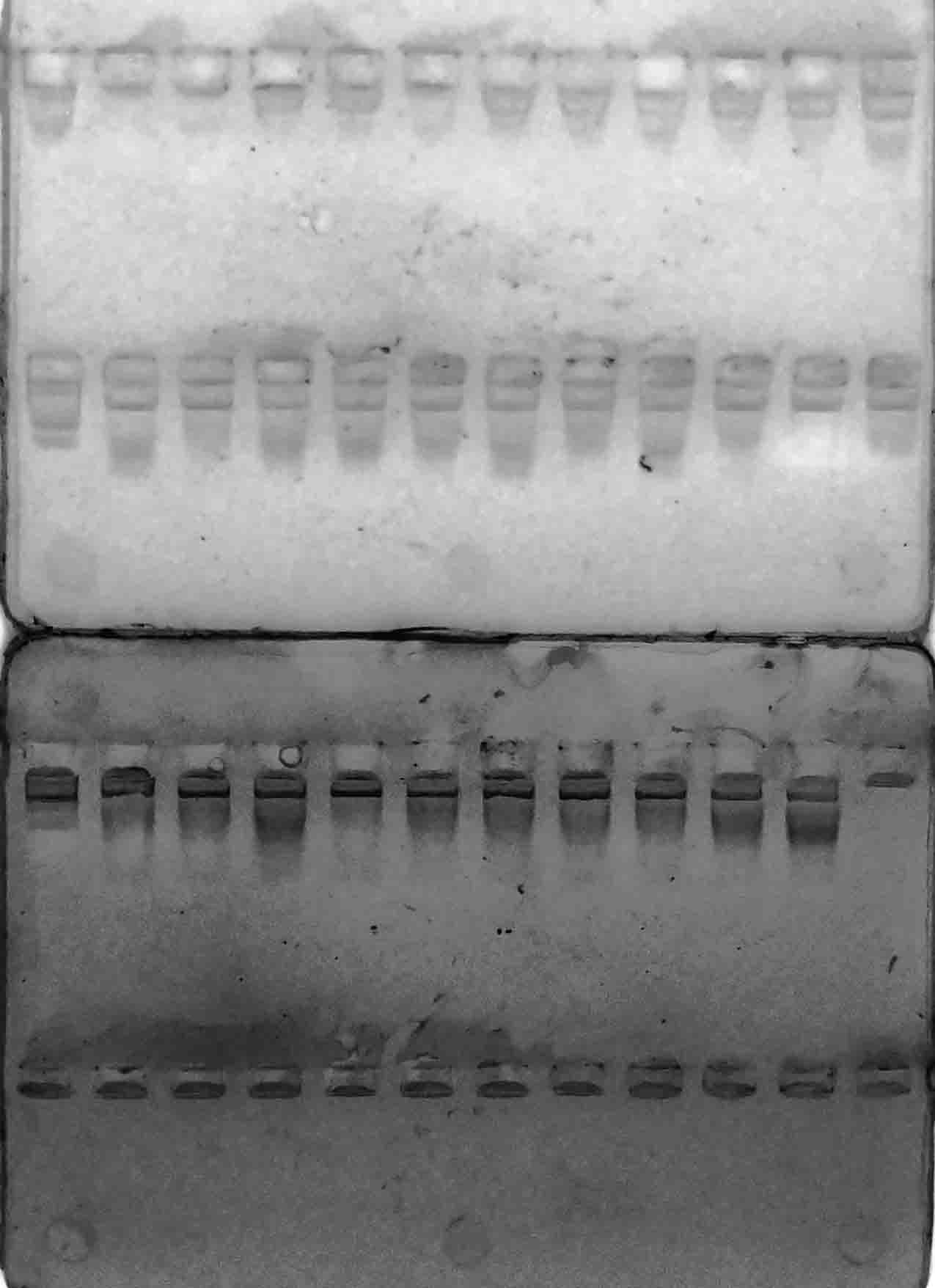
Рисунок 24 – Разделение продуктов амплификации аллелей гена VvMybA2

Рисунок 25 – Разделение продуктов амплификации аллелей гена VvMybA2
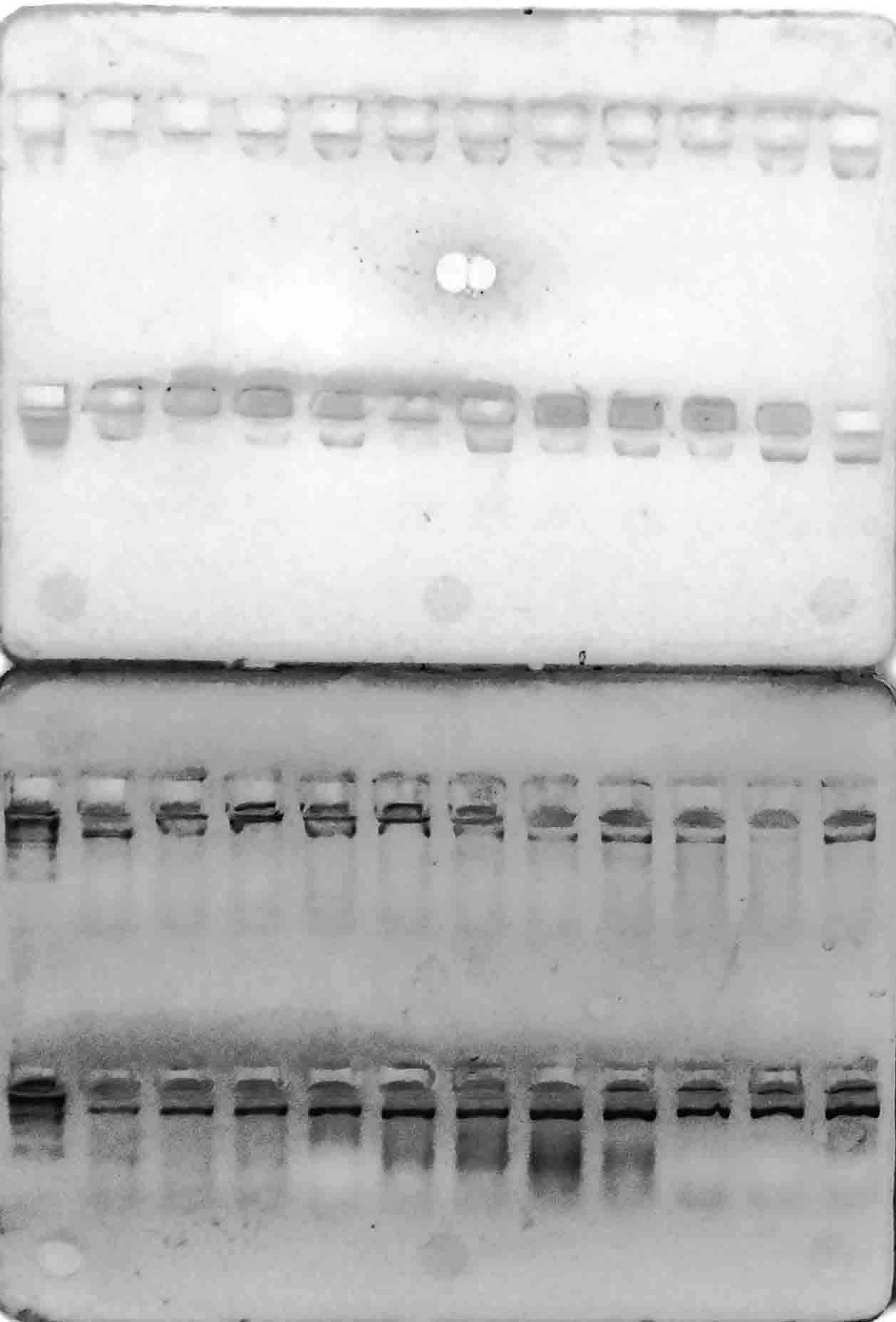
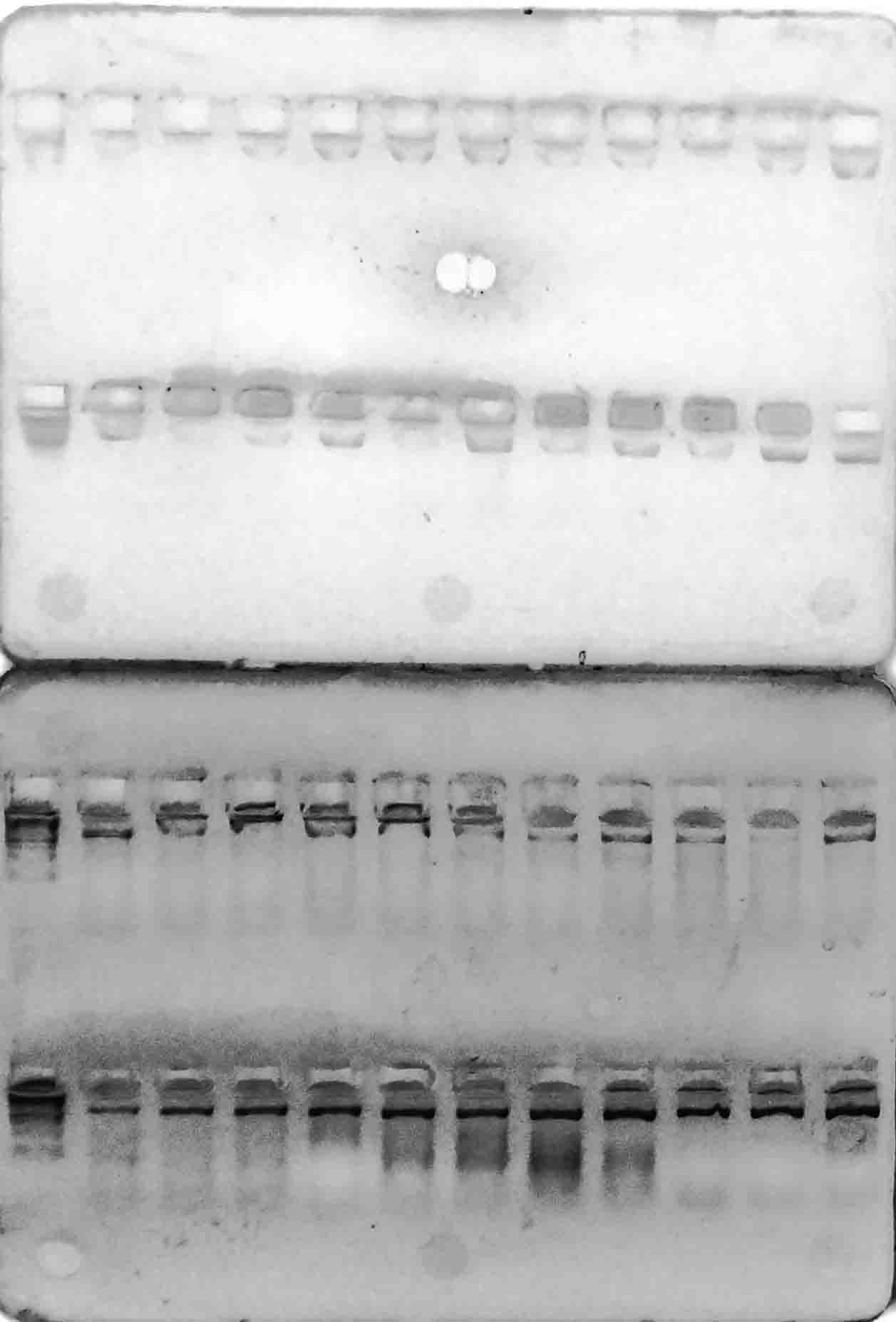
Рисунок 26 – Разделение продуктов амплификации аллелей гена VvMybA2
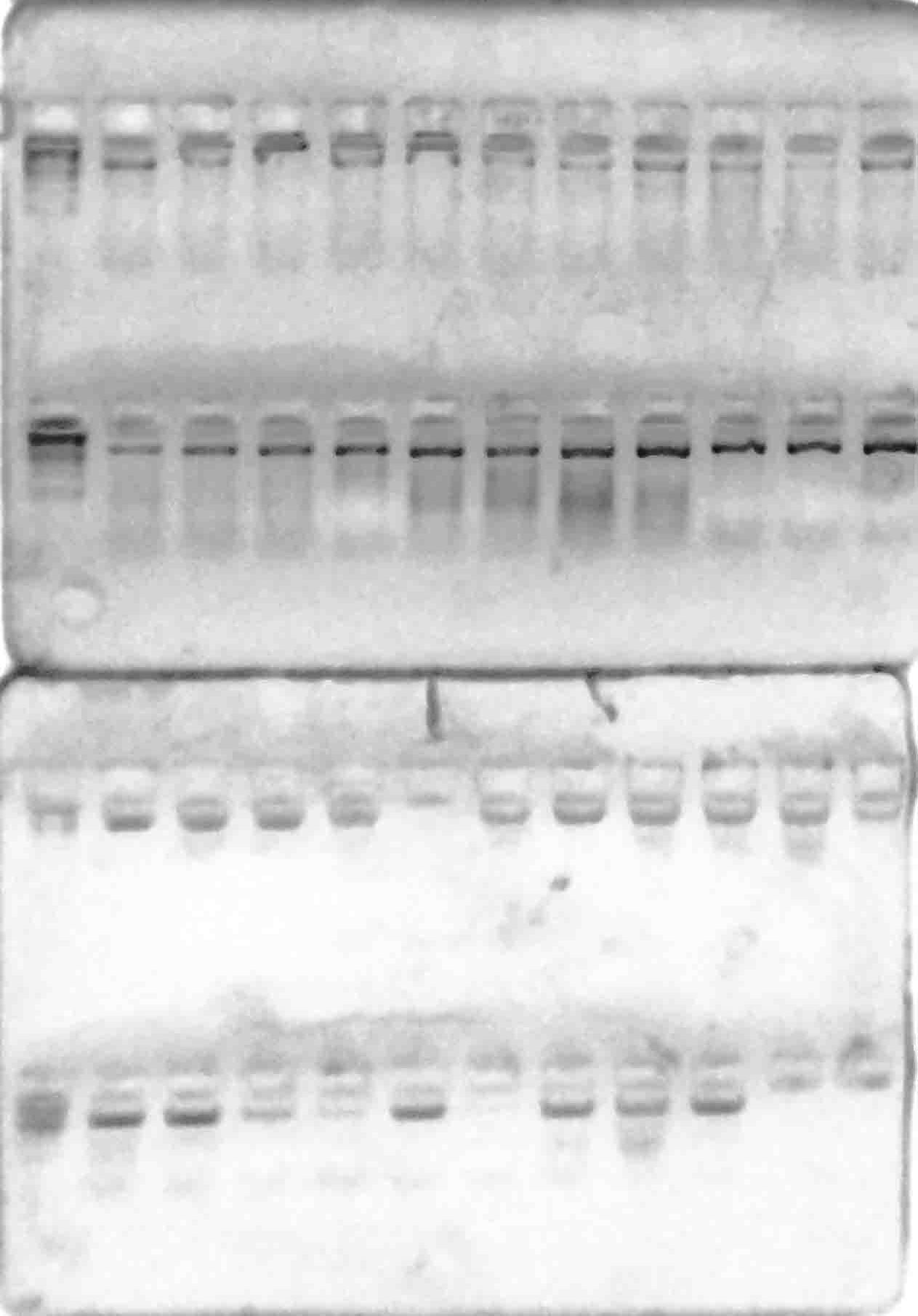
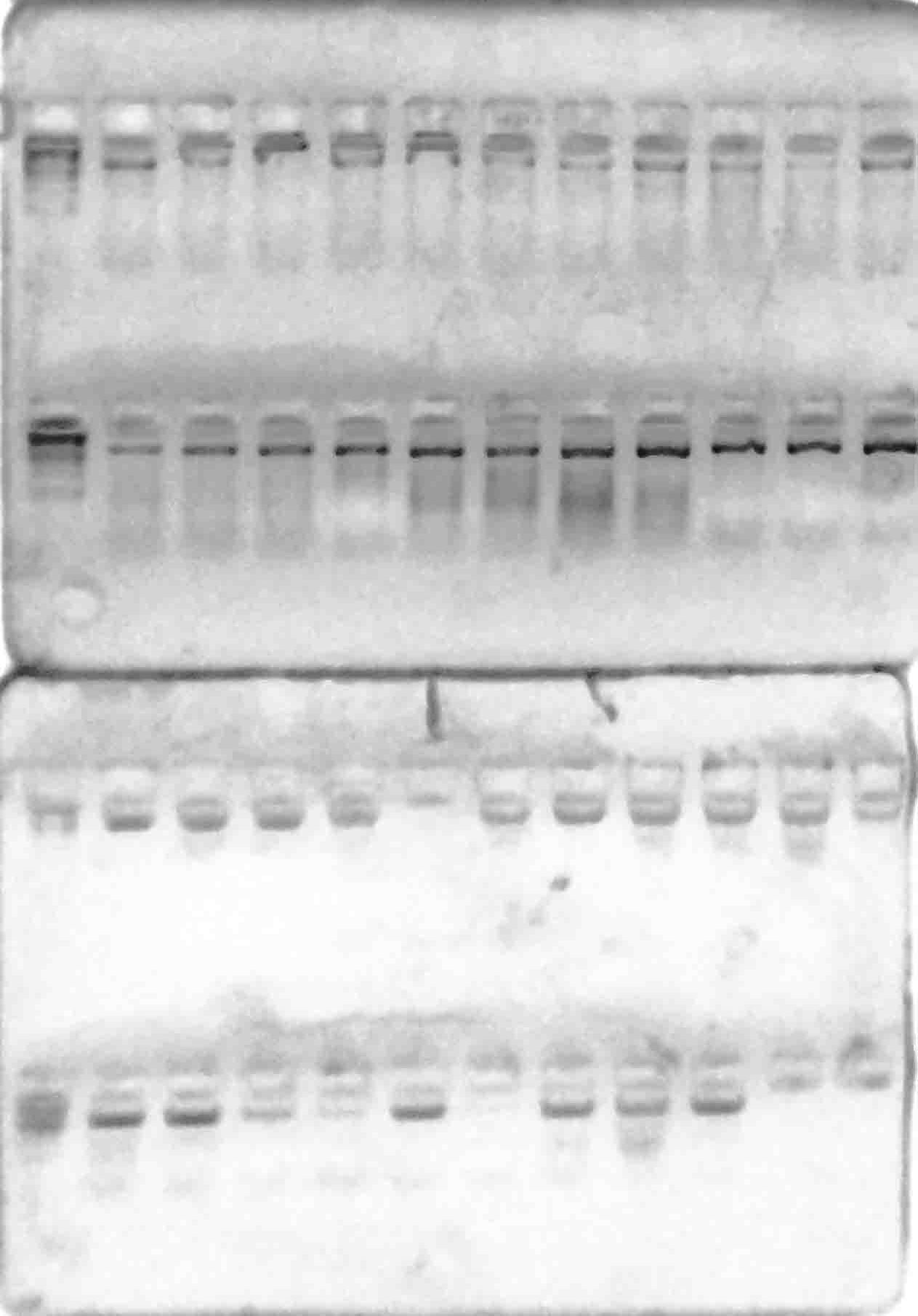
Рисунок 27 – Разделение продуктов амплификации аллелей гена VvMybA2
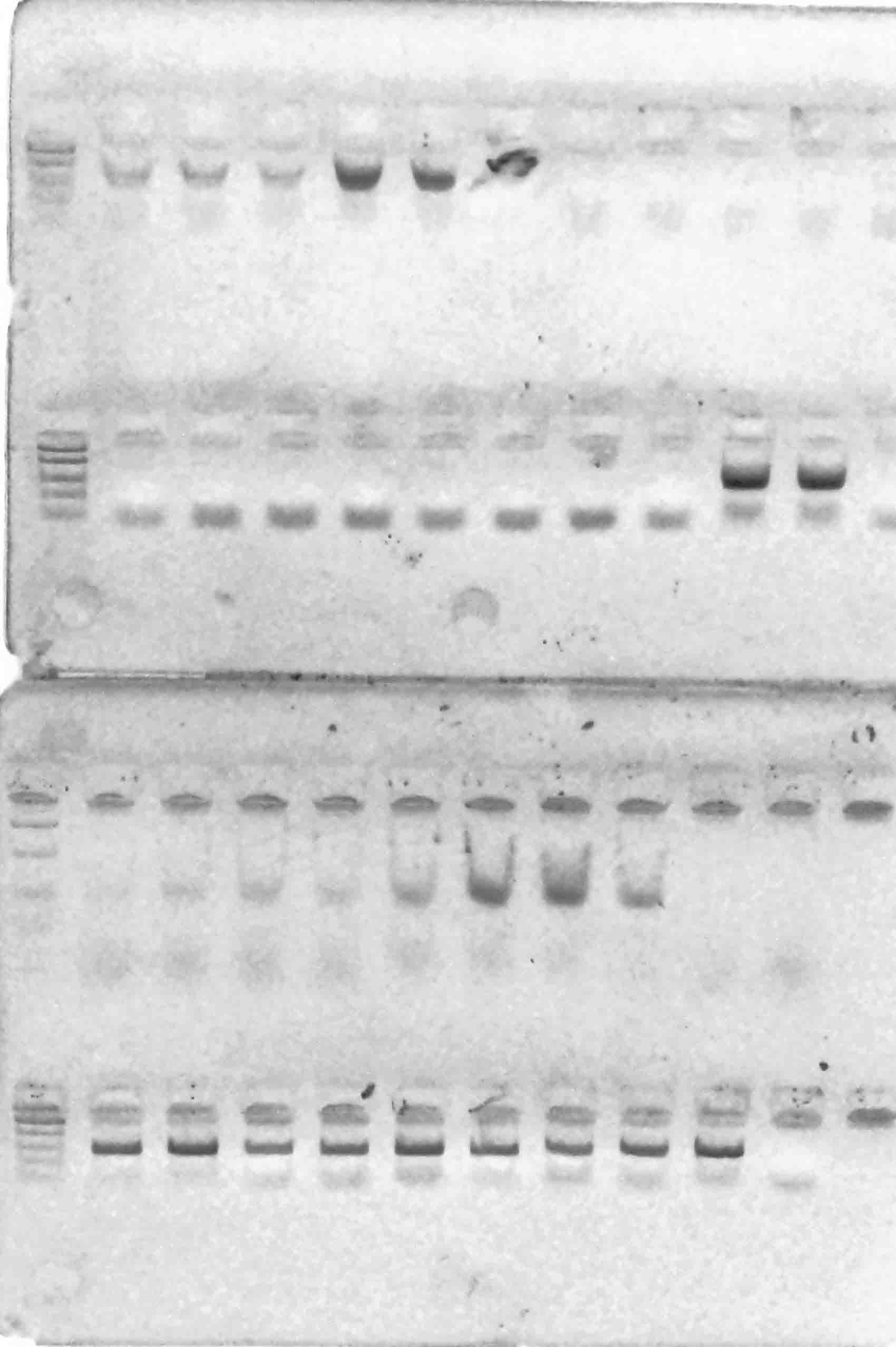
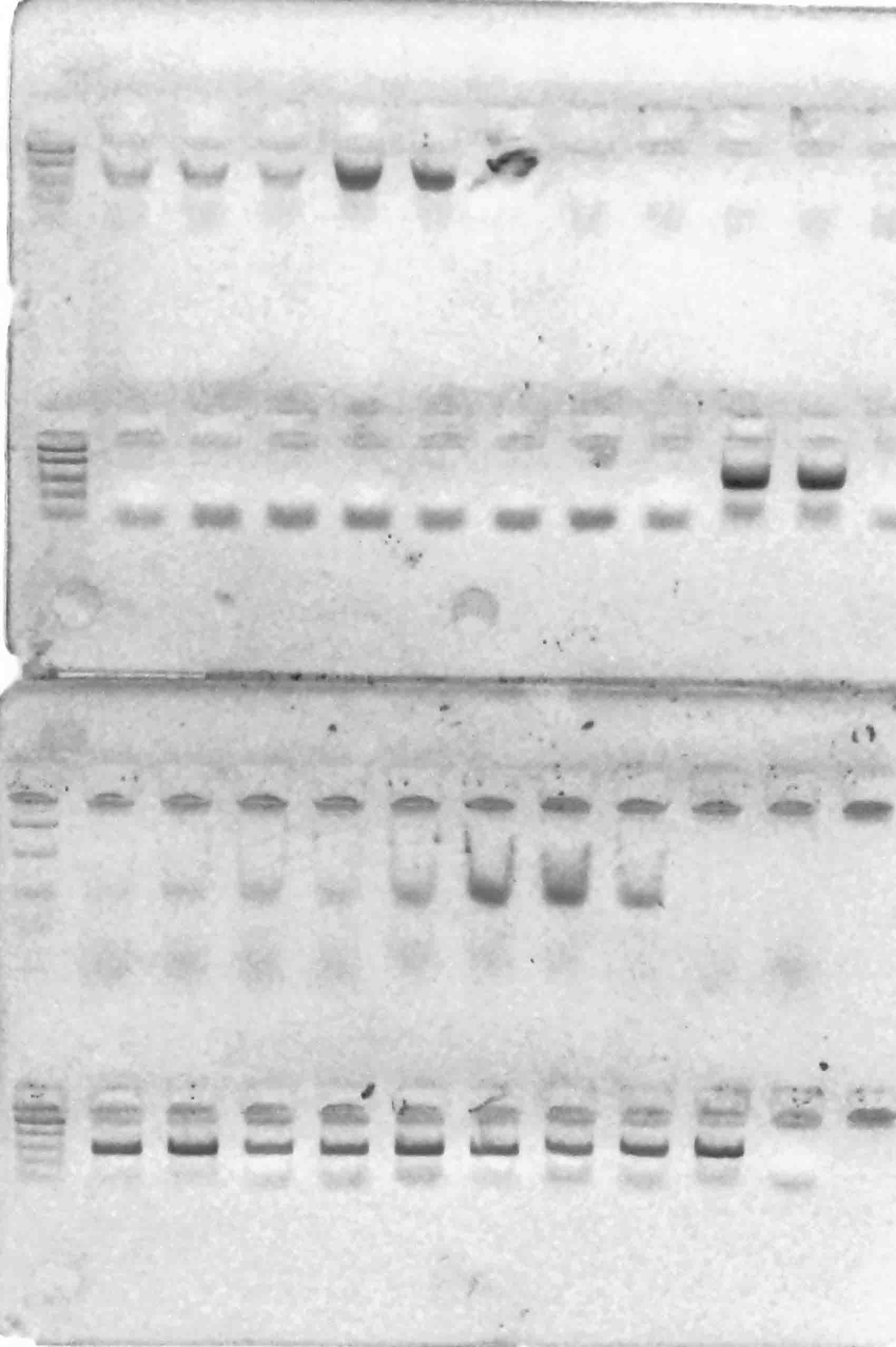
Рисунок 28 – Разделение продуктов амплификации аллелей гена VvMybA2
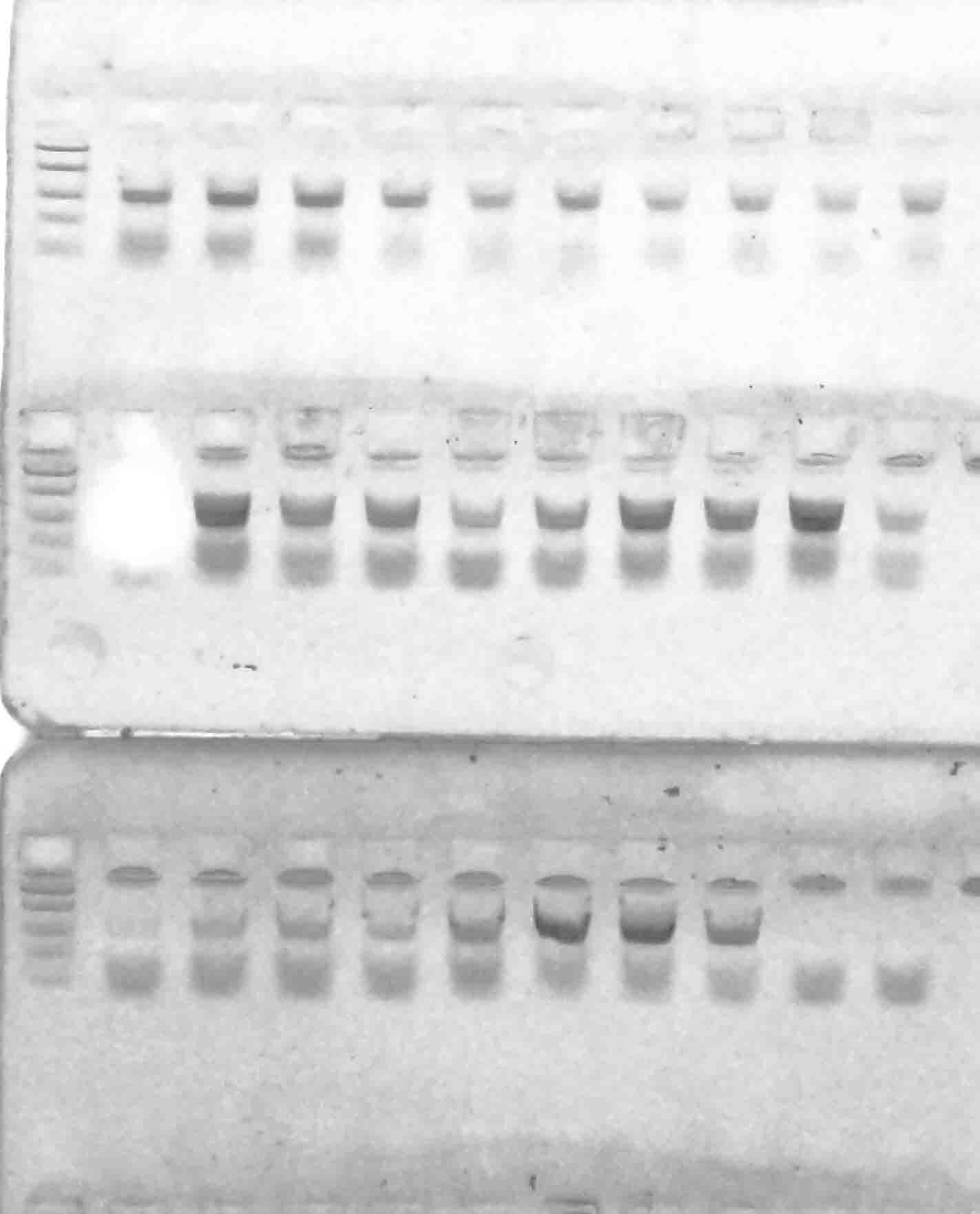
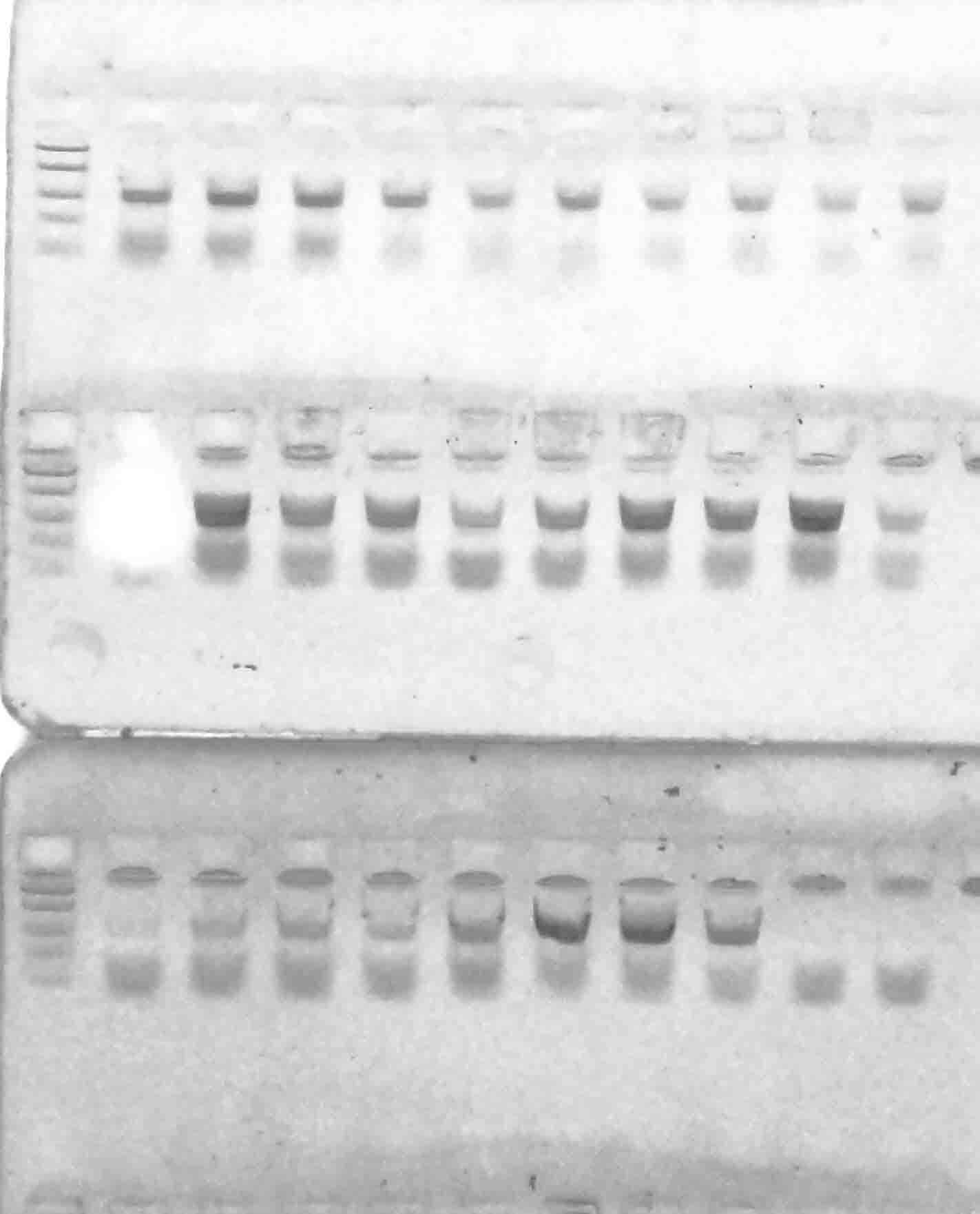
Рисунок 29 – Разделение продуктов амплификации аллелей гена VvMybA2
В результате работы нами были получены аллели искомого гена у изученных сортов. Стоит отметить, что выявленные аллели были визуально одинакового размера (примерно 1000 п.н.). Такой результат является вполне ожидаемым, в связи с тем, что указанные маркеры дают возможность детектировать аллели одинакового размера как у окрашенных, так и у неокрашенных сортов. Однако, как также известно, сами же последовательности нуклеотидов данных аллелей могут быть полиморфны. Поэтому, нами было проведено секвенирование с последующим выравниванием данных сиквенсов при помощи программы Clustal Omega с целью анализа их строения (рисунок 30).
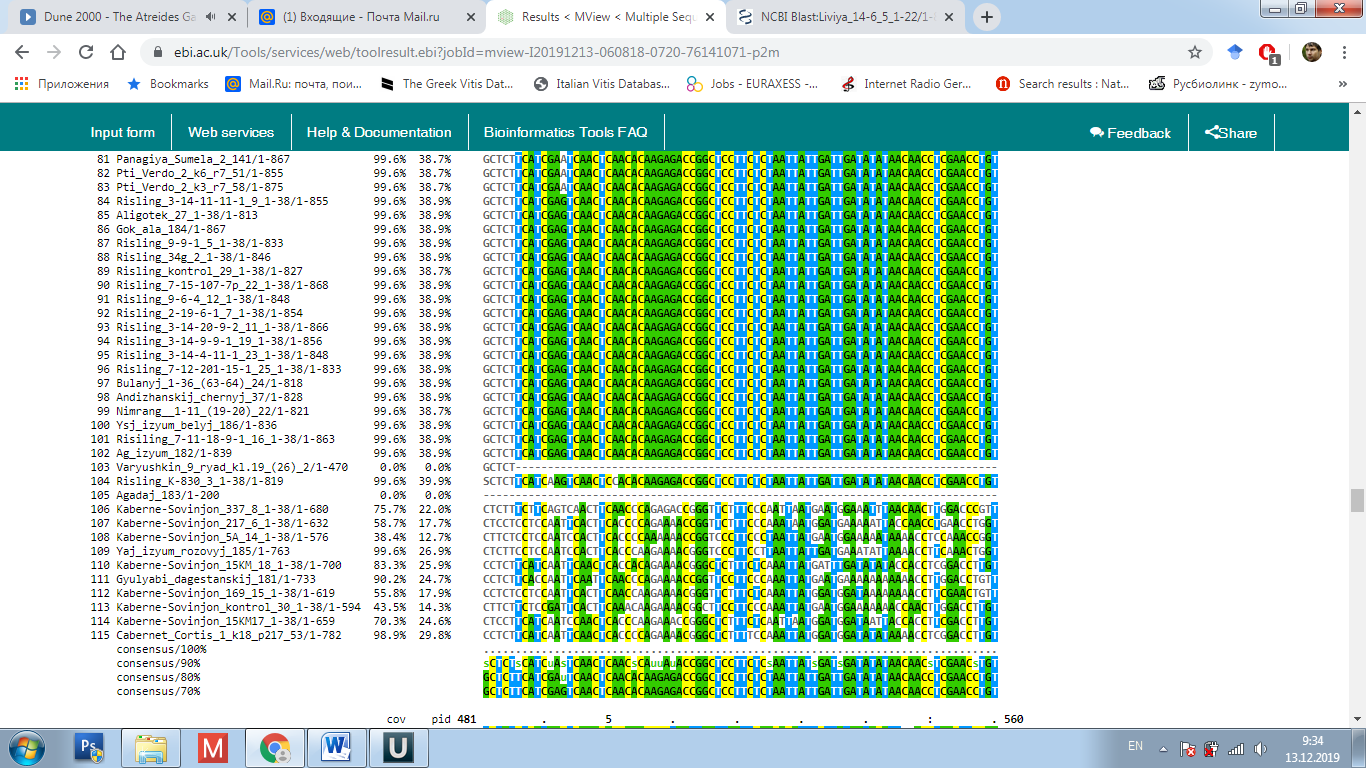
Рисунок 30 – Выравнивание нуклеотидных последовательностей
в программе Clustal Omega
Выравнивание данных последовательностей аллелей изученных сортов показало полиморфное строение у многих образцов (см. рисунок 30). При этом наиболее интересным было обнаружение полиморфности среди красноягодных сортов винограда. В то время как практически все белоягодные сорта имели практически одинаковое строение аллели. Это интересно в виду того, что, на данный момент известно всего несколько аллелей гена VvMybA2, которые ответственны за наличие либо отсутствие окраски (рисунок 31).
Помимо этого, известны также и последовательности у нескольких видов, принадлежащих к семейству Vitaceae, также доступные в NCBI. Тем не менее, для сравнения полученных результатов с уже имеющимися мы провели поиск алгоритмами NCBI BLAST среди семейства Vitaceae по параметрам наибольшего сходства (таблица 5).
Как мы видим из таблицы 5, практически все сорта показали сходство именно с аллелью Vitis heyneana myb-related transcription factor A2 gene, complete cds, при этом сортоклон Каберне-Совиньон 5А продемонстрировал высокую схожесть с формой аллели Vitis vinifera R2R3 MYB transcription factor (MYBA2) gene, MYBA2-white allele, complete cds. С одной стороны это было неожиданным, так как сорт Каберне-Совиньон обладает яркой окраской ягоды, с другой стороны, этот сорт гетерозиготный, так как произошел от белоягодного и красноягодного сортов. И, данный факт только подтверждает это, в связи
с тем, что остальные клоны сорта Каберне-Совиньон показали схожесть с аллелью Vitis heyneana myb-related transcription factor A2 gene, complete cds. Данное совпадение является также интересным, так как это разные виды, тем не менее, как уже было сказано, аллелей VvMybA2 имеется всего несколько в базе данных. Тем более что качество сиквенса было достаточно хорошим (рисунок 32).
Рисунок 31 – Результаты поиска нуклеотидных последовательностей VvMybA2
Рисунок 32 – Нуклеотидная последовательность сорта Рислинг 34г
Для того чтобы проверить имеются ли еще генотипы и, соответственно, аллели с такими же нуклеотидными заменами и насколько высока схожесть
у изученных нами сортов с уже описанными, мы провели поиск в базе данных NCBI (Таблица 5).
Таблица 5 – Результаты поиска последовательностей в базе данных NCBI
| Название генотипа |
Название гена | Процент совпадения |
| 1 | 2 | 3 |
| Траминер | Vitis heyneana myb-related transcription factor A2 gene, complete cds | 98,10 |
| Сибирьковый 14р. Клет. 19с | Vitis heyneana myb-related transcription factor A2 gene, complete cds | 97,66 |
| Сибирьковый | Vitis heyneana myb-related transcription factor A2 gene, complete cds | 96,48 |
| Шаани | Vitis heyneana myb-related transcription factor A2 gene, complete cds | 98,10 |
| Алый терский | Vitis heyneana myb-related transcription factor A2 gene, complete cds | 98,39 |
| Португальский синий |
Vitis heyneana myb-related transcription factor A2 gene, complete cds | 97,51 |
| Кульджинский | Vitis heyneana myb-related transcription factor A2 gene, complete cds | 97,80 |
| Чинури 1-7 (18-19) |
Vitis heyneana myb-related transcription factor A2 gene, complete cds | 98,10 |
| Риш баба 1-8 (33-34) |
Vitis heyneana myb-related transcription factor A2 gene, complete cds | 98,09 |
| Чауш | Vitis heyneana myb-related transcription factor A2 gene, complete cds | 98,10 |
| Уни белый (92-93) |
Vitis heyneana myb-related transcription factor A2 gene, complete cds | 97,95 |
| Нимранг 1-11 (19-20) | Vitis heyneana myb-related transcription factor A2 gene, complete cds | 97,51 |
| Кизил сапак 1-8 (61-62) |
Vitis heyneana myb-related transcription factor A2 gene, complete cds | 97,22 |
| Буланый 1-36 (63-64) | Vitis heyneana myb-related transcription factor A2 gene, complete cds | 97,36 |
| Хатми 1-11 (76-77) |
Vitis heyneana myb-related transcription factor A2 gene, complete cds | 97,95 |
| Воскеат 1-38 (70-71) |
Vitis heyneana myb-related transcription factor A2 gene, complete cds | 98,10 |
| Цоликаури 1-36 (49-50) | Vitis heyneana myb-related transcription factor A2 gene, complete cds | 98,10 |
| Шабаш | Vitis heyneana myb-related transcription factor A2 gene, complete cds | 98,10 |
| Гамэ черный | Vitis heyneana myb-related transcription factor A2 gene, complete cds | 97,80 |
| Чиляки 1-23 (35-36) |
Vitis heyneana myb-related transcription factor A2 gene, complete cds | 97,95 |
| Продолжение таблицы 5 | ||
| 1 | 2 | 3 |
| Тербаш 1-23 (35-34) |
Vitis heyneana myb-related transcription factor A2 gene, complete cds | 98,10 |
| Баян Ширей | Vitis heyneana myb-related transcription factor A2 gene, complete cds | 97,95 |
| Катта-Курган | Vitis heyneana myb-related transcription factor A2 gene, complete cds | 97,95 |
| Андижанский черный |
Vitis heyneana myb-related transcription factor A2 gene, complete cds | 97,80 |
| Босса | Vitis heyneana myb-related transcription factor A2 gene, complete cds | 96,49 |
| Хатми | Vitis heyneana myb-related transcription factor A2 gene, complete cds | 97,66 |
| Мсхвилтвала Тетри | Vitis heyneana myb-related transcription factor A2 gene, complete cds | 97,95 |
| Тита рбили | Vitis heyneana myb-related transcription factor A2 gene, complete cds | 97,95 |
| Пти Вердо 2 к6 р7 |
Vitis heyneana myb-related transcription factor A2 gene, complete cds | 98,10 |
| Пти Вердо 1 к3 р7 |
Vitis heyneana myb-related transcription factor A2 gene, complete cds | 98,54 |
| Кортис 1 к18 р 217 |
Vitis heyneana myb-related transcription factor A2 gene, complete cds | 89,49 |
| Пти Вердо 1 к6 р7 |
Vitis heyneana myb-related transcription factor A2 gene, complete cds | 98,54 |
| Пти Вердо 2 к3 р7 |
Vitis heyneana myb-related transcription factor A2 gene, complete cds | 98,24 |
| Солярис К | Vitis heyneana myb-related transcription factor A2 gene, complete cds | 97,66 |
| Шардоне 1-7 (13-14) |
Vitis heyneana myb-related transcription factor A2 gene, complete cds | 98,10 |
| Панагия Сумела 2 |
Vitis heyneana myb-related transcription factor A2 gene, complete cds | 98,10 |
| Панагия Сумела 4 |
Vitis heyneana myb-related transcription factor A2 gene, complete cds | 98,24 |
| Панагия Сумела 6 |
Vitis heyneana myb-related transcription factor A2 gene, complete cds | 98,24 |
| Панагия Сумела 11 |
Vitis heyneana myb-related transcription factor A2 gene, complete cds | 97,95 |
| Антарис | Vitis heyneana myb-related transcription factor A2 gene, complete cds | 98,24 |
| Жупски Баядисер |
Vitis heyneana myb-related transcription factor A2 gene, complete cds | 98,10 |
| Жупски 3куст | Vitis heyneana myb-related transcription factor A2 gene, complete cds | 98,24 |
| Шампанчик Цимлянский | Vitis heyneana myb-related transcription factor A2 gene, complete cds | 98,10 |
| Кумшацкий белый |
Vitis heyneana myb-related transcription factor A2 gene, complete cds | 98,10 |
| Сыпун черный | Vitis heyneana myb-related transcription factor A2 gene, complete cds | 98,39 |
| Продолжение таблицы 5 | ||
| 1 | 2 | 3 |
| Будай шули | Vitis heyneana myb-related transcription factor A2 gene, complete cds | 97,95 |
| Гок изюм | Vitis heyneana myb-related transcription factor A2 gene, complete cds | 97,95 |
| Гюляби дагестанский |
Vitis heyneana myb-related transcription factor A2 gene, complete cds | 84,86 |
| Аг изюм | Vitis heyneana myb-related transcription factor A2 gene, complete cds | 97,80 |
| Гок ала | Vitis heyneana myb-related transcription factor A2 gene, complete cds | 97,07 |
| Яй изюм розовый |
Vitis heyneana myb-related transcription factor A2 gene, complete cds | 83,33 |
| Яй изюм белый | Vitis heyneana myb-related transcription factor A2 gene, complete cds | 97,66 |
| Рисилинг Алькадар 34а |
Vitis heyneana myb-related transcription factor A2 gene, complete cds | 99,93 |
| Рисилинг 34г | Vitis heyneana myb-related transcription factor A2 gene, complete cds | 97,22 |
| Пино белый 32 | Vitis heyneana myb-related transcription factor A2 gene, complete cds | 97,51 |
| Рисилинг 9-9-1 | Vitis heyneana myb-related transcription factor A2 gene, complete cds | 97,36 |
| Каберне-Совиньон 217 | Vitis heyneana myb-related transcription factor A2 gene, complete cds | 84,15 |
| Рисилинг 2-19-6-1 |
Vitis heyneana myb-related transcription factor A2 gene, complete cds | 97,66 |
| Каберне-Совиньон 337 | Vitis heyneana myb-related transcription factor A2 gene, complete cds | 86,19 |
| Рисилинг 3-14-11-11-1 |
Vitis heyneana myb-related transcription factor A2 gene, complete cds | 96,49 |
| Пино белый 31 | Vitis heyneana myb-related transcription factor A2 gene, complete cds | 97,51 |
| Рисилинг 3-14-20-9-2 |
Vitis heyneana myb-related transcription factor A2 gene, complete cds | 97,80 |
| Рисилинг 9-6-4 | Vitis heyneana myb-related transcription factor A2 gene, complete cds | 97,51 |
| Пино белый 46 | Vitis heyneana myb-related transcription factor A2 gene, complete cds | 97,95 |
| Каберне-Совиньон 5А | Vitis vinifera R2R3 MYB transcription factor (MYBA2) gene, MYBA2-white allele, complete cds | 90,07 |
| Каберне-Совиньон 169 | Vitis heyneana myb-related transcription factor A2 gene, complete cds | 84,75 |
| Рисилинг 7-11-18-9-1 |
Vitis heyneana myb-related transcription factor A2 gene, complete cds | 97,66 |
| Каберне-Совиньон 15КМ | Vitis heyneana myb-related transcription factor A2 gene, complete cds | 87,98 |
| Каберне-Совиньон 15КМ | Vitis heyneana myb-related transcription factor A2 gene, complete cds | 89,41 |
| Рисилинг 3-14-9-9-1 |
Vitis heyneana myb-related transcription factor A2 gene, complete cds | 97,80 |
| Продолжение таблицы 5 | ||
| 1 | 2 | 3 |
| Пино белый 6 | Vitis heyneana myb-related transcription factor A2 gene, complete cds | 97,66 |
| Рисилинг Алькадар 34 |
Vitis heyneana myb-related transcription factor A2 gene, complete cds | 98,10 |
| Рисилинг 7-15-107-7п |
Vitis heyneana myb-related transcription factor A2 gene, complete cds | 97,66 |
| Рисилинг 3-14-4-11-1 |
Vitis heyneana myb-related transcription factor A2 gene, complete cds | 97,80 |
| Мерло 15 | Vitis heyneana myb-related transcription factor A2 gene, complete cds | 97,95 |
| Рисилинг 7-12-201-15-1 |
Vitis heyneana myb-related transcription factor A2 gene, complete cds | 97,80 |
| Клон Грамотенко |
Vitis heyneana myb-related transcription factor A2 gene, complete cds | 97,51 |
| Алиготэк | Vitis heyneana myb-related transcription factor A2 gene, complete cds | 96,93 |
| Мерло 14МГ | Vitis heyneana myb-related transcription factor A2 gene, complete cds | 98,09 |
| Рисилинг контроль |
Vitis heyneana myb-related transcription factor A2 gene, complete cds | 97,66 |
| Каберне-Совиньон контроль |
Vitis heyneana myb-related transcription factor A2 gene, complete cds | 85,66 |
| Алиготе контроль |
Vitis heyneana myb-related transcription factor A2 gene, complete cds | 97,80 |
| Совиньон 1 | Vitis heyneana myb-related transcription factor A2 gene, complete cds | 97,80 |
| Совиньон 2 | Vitis heyneana myb-related transcription factor A2 gene, complete cds | 97,95 |
| Совиньон 3 | Vitis heyneana myb-related transcription factor A2 gene, complete cds | 93,37 |
| Совиньон 4 | Vitis heyneana myb-related transcription factor A2 gene, complete cds | 97,95 |
| Совиньон контроль |
Vitis heyneana myb-related transcription factor A2 gene, complete cds | 97,95 |
| Пино белый контроль |
Vitis heyneana myb-related transcription factor A2 gene, complete cds | 95,31 |
| Мерло контроль | Vitis heyneana myb-related transcription factor A2 gene, complete cds | 97,95 |
| Ливия 14-6 | Vitis heyneana myb-related transcription factor A2 gene, complete cds | 97,95 |
| Ливия 14-5 | Vitis heyneana myb-related transcription factor A2 gene, complete cds | 97,66 |
| Ливия | Vitis heyneana myb-related transcription factor A2 gene, complete cds | 97,07 |
| Ливия Ф | Vitis heyneana myb-related transcription factor A2 gene, complete cds | 98,10 |
| Гелиос 50-5 | Vitis heyneana myb-related transcription factor A2 gene, complete cds | 96,78 |
| Продолжение таблицы 5 | ||
| 1 | 2 | 3 |
| Гелиос 50-6 | Vitis heyneana myb-related transcription factor A2 gene, complete cds | 96,78 |
| Академический К | Vitis heyneana myb-related transcription factor A2 gene, complete cds | 97,80 |
| Долгожданный 6-8 | Vitis heyneana myb-related transcription factor A2 gene, complete cds | 98,10 |
| Долгожданный 6-9 | Vitis heyneana myb-related transcription factor A2 gene, complete cds | 98,10 |
| Долгожданный | Vitis heyneana myb-related transcription factor A2 gene, complete cds | 98,10 |
| Долгожданный Ф | Vitis heyneana myb-related transcription factor A2 gene, complete cds | 98,10 |
| Антоний Великий 30-5 |
Vitis heyneana myb-related transcription factor A2 gene, complete cds | 98,10 |
| Антоний Великий 30-6 |
Vitis heyneana myb-related transcription factor A2 gene, complete cds | 98,10 |
| Антоний Великий |
Vitis heyneana myb-related transcription factor A2 gene, complete cds | 98,10 |
| Антоний Великий Ф |
Vitis heyneana myb-related transcription factor A2 gene, complete cds | 98,10 |
| Аркадия розовая 2-5 |
Vitis heyneana myb-related transcription factor A2 gene, complete cds | 97,95 |
| Аркадия розовая 2-6 |
Vitis heyneana myb-related transcription factor A2 gene, complete cds | 98,10 |
В целом, результаты такой проверки, полученные при сравнении с базой данных NCBI дали, с одной стороны, вполне ожидаемый результат, а с другой – неожиданным было то, что схожесть выявлена именно с другим видом того же семейства. Тем не менее, в виду того, что нами использовалось секвенирование при использовании прямого и обратного праймеров, у некоторых сортов было выявлено гетерозиготное состояние локуса. Это, конечно же, является особенностью нашей выборки генотипов, но в целом не удивительно, так как у них не известно их происхождение. Тем не менее, как показано в таблице 5, были выявлены как процентные, так и качественные различия у изученных генотипов. Это говорит нам о гетерогенном строении гена, который был выбран для изучения, что также подтверждается предыдущими данными, так как при его анализе в прошлых исследованиях было выявлено 19 полиморфных сайтов.
Как показано в таблице 5 у некоторых сортов было выявлено наличие различий в строении изученных аллелей (что также подтверждается и выравниванием в программе Clustal Omega) при сравнении с уже имеющимися в базе данных сортами. Не смотря на это, при анализе результатов было уточнено, что обнаружены не только различия между нуклеотидными последовательностями, но и гетерогенность, очевидно вызванная одновременным наличием, очевидно, двух аллелей (функциональной и не функциональной) в генотипах и, соответственно, их сложным селекционным происхождением, которое до конца пока не установлено из-за того, что данные сорта являются стародавними и, вполне возможно, их родительские формы не могут быть обнаружены в виду того, что являются утерянными. Тем не менее, выявленные особенности их строения являются крайне интересными для последующего филогенетического изучения истории этих сортов и форм, а также уточнению их происхождения.
В остальном стоит отметить, что полученные результаты у генотипов соответствовали ожидаемым и, чтобы проверить влияние строения аллелей на аминокислотную последовательность, мы провели сравнение при помощи алгоритмов blastx (таблица 6).
Таблица 6 – Результаты сравнение аминокислотных последовательностей
изученных сортов винограда с базой данных NCBI
| Название | Результат поиска в NCBI | Процент совпадения |
Код генбанка |
| 1 | 2 | 3 | 4 |
| Траминер | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Сибирьковый 14р. Клет. 19с | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
97,78 | BAD18978.1 |
| Сибирьковый | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
91,11 | BAD18978.1 |
| Шаани | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Алый терский | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
97,78 | BAD18978.1 |
| Португальский синий |
myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Кульджинский | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Чинури 1-7 (18-19) | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Риш баба 1-8 (33-34) |
myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Чауш | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Продолжение таблицы 6 | |||
| 1 | 2 | 3 | 4 |
| Уни белый (92-93) |
myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Нимранг 1-11 (19-20) |
myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Кизил сапак 1-8 (61-62) |
myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
93,33 | BAD18978.1 |
| Буланый 1-36 (63-64) |
myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
91,11 | BAD18978.1 |
| Хатми 1-11 (76-77) |
myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Воскеат 1-38 (70-71) |
myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Цоликаури 1-36 (49-50) |
myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Шабаш | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Гамэ черный | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Чиляки 1-23 (35-36) |
myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Тербаш 1-23 (35-34) |
myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Баян Ширей | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Катта-Курган | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Андижанский черный |
myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
97,78 | BAD18978.1 |
| Босса | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Хатми | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Мсхвилтвала Тетри |
myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Тита рбили | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Пти Вердо 2 к6 р7 | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Пти Вердо 1 к3 р7 | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
93,78 | BAD18978.1 |
| Кортис 1 к18 р 217 | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Пти Вердо 1 к6 р7 | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
97,78 | BAD18978.1 |
| Пти Вердо 2 к3 р7 | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
97,78 | BAD18978.1 |
| Солярис К | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
95,56 | BAD18978.1 |
| Шардоне 1-7 (13-14) |
myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
97,78 | BAD18978.1 |
| Продолжение таблицы 6 | |||
| 1 | 2 | 3 | 4 |
| Панагия Сумела 2 | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
95,67 | BAD18978.1 |
| Панагия Сумела 4 | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
97,78 | BAD18978.1 |
| Панагия Сумела 6 | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
97,78 | BAD18978.1 |
| Панагия Сумела 11 | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
95,56 | BAD18978.1 |
| Антарис | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
97,78 | BAD18978.1 |
| Жупски Баядисер | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
97,78 | BAD18978.1 |
| Жупски 3куст | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
97,78 | BAD18978.1 |
| Шампанчик Цимлянский |
myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Кумшацкий белый |
myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Сыпун черный | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
97,78 | BAD18978.1 |
| Будай шули | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Гок изюм | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Гюляби дагестанский |
myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Аг изюм | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Гок ала | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
95,56 | BAD18978.1 |
| Яй изюм розовый | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
70,37 | RVW21054.1 |
| Яй изюм белый | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Рисилинг Алькадар 34а |
myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
95,56 | BAD18978.1 |
| Рисилинг 34г | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
97,78 | BAD18978.1 |
| Пино белый 32 | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
93,33 | BAD18978.1 |
| Рисилинг 9-9-1 | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Каберне- Совиньон 217 |
myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Рисилинг 2-19-6-1 | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Каберне- Совиньон 337 |
Transcription factor MYB113 [Vitis vinifera] |
77,78 | RVW21054.1 |
| Рисилинг 3-14-11-11-1 |
myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
93,33 | BAD18978.1 |
| Продолжение таблицы 6 | |||
| 1 | 2 | 3 | 4 |
| Пино белый 31 | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
97,78 | BAD18978.1 |
| Рисилинг 3-14-20-9-2 |
myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Рисилинг 9-6-4 | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Пино белый 46 | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Каберне- Совиньон 5А |
myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
51,82 | BAD18978.1 |
| Каберне- Совиньон 169 |
myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Рисилинг 7-11-18-9-1 |
myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Каберне- Совиньон 15КМ |
myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Каберне- Совиньон 15КМ |
myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Рисилинг 3-14-9-9-1 |
myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Пино белый 6 | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
97,78 | BAD18978.1 |
| Рисилинг Алькадар 34 |
myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Рисилинг 7-15-107-7п |
myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
97,78 | BAD18978.1 |
| Рисилинг 3-14-4-11-1 |
myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Мерло 15 | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Рисилинг 7-12-201-15-1 |
myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Клон Грамотенко | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Алиготэк | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
95,56 | BAD18978.1 |
| Мерло 14МГ | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
71,17 | BAD18978.1 |
| Рисилинг контроль | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Каберне- Совиньон контроль |
myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Алиготе контроль | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
97,78 | BAD18978.1 |
| Совиньон 1 | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
97,78 | BAD18978.1 |
| Совиньон 2 | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Совиньон 3 | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
97,78 | BAD18978.1 |
| Продолжение таблицы 6 | |||
| 1 | 2 | 3 | 4 |
| Совиньон 4 | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Совиньон контроль |
myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Пино белый контроль |
myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
93,33 | BAD18978.1 |
| Мерло контроль | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Ливия 14-6 | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Ливия 14-5 | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
97,78 | BAD18978.1 |
| Ливия | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
95,56 | BAD18978.1 |
| Ливия Ф | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Гелиос 50-5 | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
93,33 | BAD18978.1 |
| Гелиос 50-6 | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
93,33 | BAD18978.1 |
| Академический К | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
97,78 | BAD18978.1 |
| Долгожданный 6-8 |
myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Долгожданный 6-9 |
myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Долгожданный | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Долгожданный Ф | myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Антоний Великий 30-5 |
myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Антоний Великий 30-6 |
myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Антоний Великий |
myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Антоний Великий Ф |
myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Аркадия розовая 2-5 |
myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
| Аркадия розовая 2-6 |
myb-relared transcription factor VvMYBA2 [Vitis vinifera] |
100 | BAD18978.1 |
Таким образом, результаты показывают нам распределение выявленных аллелей на три основные группы по аминокислотным последовательностям, относящимся к последовательностям в ген банке BAD18978.1, ABL14065.1
и RVW21054.1. Все исследованные сиквенсы показали схожесть с уже имеющимися в базе данных NCBI сиквенсами. При этом важно отметить, что процент совпадения был разный. Это объясняется двумя фактами: 1) сорта имеют различную окраску, и изменения в гене VvMybA2 влияют на это; 2) сорта имеют гетерогенное строение, т. е. их аллели действительно отличаются от референсных, так как эти генотипы имеют в разной степени окрашенную ягоду.
В целом было выявлено, что данные аминокислотные последовательности консервативны как при сортовом, так и при межвидовом выравнивании. Интересным же было то, что такие клоны как Мерло 14МГ, Каберне-Совиньон 337
и Яй изюм розовый показали наличие большого количества отличий от референсных аллелей, с которыми было проведено сравнение. Тем более что такие белки, обычно, являются консервативными. Также, в виду того, что все три сорта имеют яркую окраску у их ягод, предполагается, что данные различия, очевидно, не повлияли на экспрессию этого признака.
3.5 Филогенетический анализ изученных генотипов
Для изучения филогенетических взаимоотношений между изученными генотипами, мы провели кластеризацию методом Neighbor Join трех кластерных древ: гены VvMybA1 и VvMybA2 по отдельности и оба гена вместе. Такой алгоритм действий был выбран потому, как оба признака по сути являются признаками изученных организмов, но цель именно тройного анализа – сравнение полученных результатом: по отдельности и обобщенных.
Таким образом, нами были построены кластерные древа, содержащие изученные генотипы сортов и форм (рисунки 33–35).
Сравнение трех Neighbor Join кластеризаций, основанных на трех вариантах нуклеотидных сиквенсах, выявило, в целом, схожее распределение генотипов. Тем не менее, наиболее точное древо, как и ожидалось, было построено при использовании полных данных о нуклеотидных последовательностях двух генов.
В целом, все генотипы распределились на четыре макро-кластера, которые представлены основными четырьмя вариациями аллелей. И как мы можем видеть, через середину древа можно провести мнимую линию, которая будет разграничивать два основных сортотипа: с окрашенными и с не окрашенными ягодами.

Рисунок 33 – Neighbor Join древо, построенное на основе данных
о ДНК-последовательностях гена VvMybA1
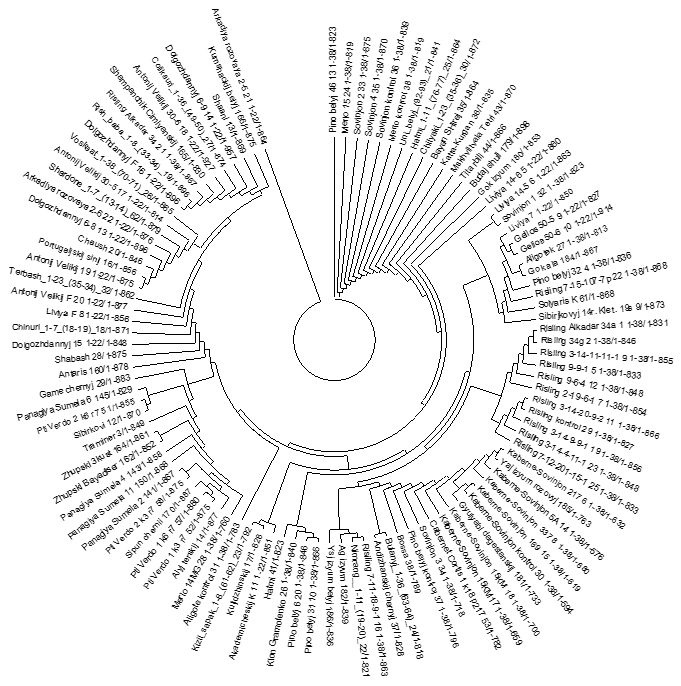
Рисунок 34 – Neighbor Join древо, построенное на основе данных
о ДНК-последовательностях гена VvMybA2
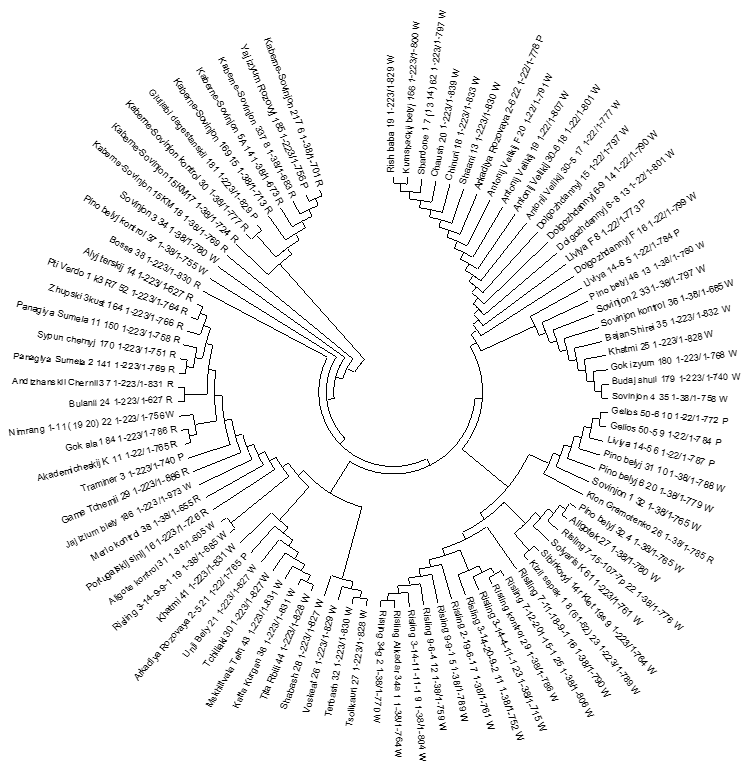
Рисунок 35 – Neighbor Join древо, построенное на основе данных о ДНК-последовательностях генов VvMybA1 и VvMybA2
Тем не менее, имелись и некоторые смешения в кластерах. Например,
к кластеру красноягодных сортов был отнесен генотип сорта Яй изюм белый. Это интересно в виду того, что имеются также такие сорта как Яй изюм черный и Яй изюм розовый. Как мы видим, Яй изюм розовый попал к сортам, имеющим яркую окраску, в то время как белоягодный сорт был дистанцирован от него ближе к макро-кластеру неокрашенных сортов. Отсюда можно предположить, что данный генотип имеет аллель близкородственную окрашенным генотипам и, возможно, произошел в виду появления какой-то мутации, заблокировавшей экспрессию окраски. Также интересно и то, что сорта Яй изюм белый
и Яй изюм розовый не расположены рядом друг с другом, что наводит на мысль об их независимом происхождении, но, тем не менее, близком родстве. То есть вполне возможно они оба произошли от сорта Яй изюм черный, но
в виду независимо произошедших мутаций.
Стоит отметить, что также в эту суб-группу сошел сорт Нимранг. Данный генотип имеет очень интересное проявление окраски, так как ягоды нельзя назвать полностью розовыми или зелеными. Тем не менее, мы отнесли его
к слабоокрашенным сорта, но по результатам объединенных данных было выявлено, что строение его аллелей позволяет отнести его к окрашенным сортам. Также, его положение рядом с окрашенным сортом Гок ала дает возможность предположить, что происхождение сорта также относится к сортам бассейна Черного моря и, вполне возможно, кто-то из его близких или дальних предков был оттуда, отчего он и унаследовал такую аллель.
Неожиданным было то, что кластер белоягодных сортов вошел генотип Мерло клон Грамотенко, являющийся сортоклоном сорта Мерло, который имеет яркую окраску ягод. Тем не менее, выявление наличия «белой» аллели подтверждает его сложное происхождение, установленное в предыдущих исследования. Так как установлено, что его отцом является Каберне фран, а матерью – неизвестный ранее сорт.
Помимо этого, интересным результатом было и то, что такие сорта как Ливия, Гелиос и Аркадия Розовая вошли в макро-кластер белоягодных сортов. Конечно, данные сорта являются близкородственными, так как одним из их родителе был сорт Аркадия. В то время как Аркадия розовая и Гелиос имеют также общего и второго родителя: Кишмиш находка. Тем не менее, не смотря на наличие окраски у ягод, данные сорта были отнесены к группе сортов без нее. Вполне возможно, что тут наблюдается случай, противоположный тому, что мы можем видеть с сортов Нимранг. Не смотря на то, что данные сорта унаследовали наличие окраски от сорта Кишмиш находка, в то время как строение обоих или какого-то одного из генов VvMybA1 и VvMybA2 больше напоминает по своему строению «белую» аллель, что подтверждается также и слабой экспрессией данного признака.
Остальные же сорта, не имеющие окраски ягод, составили основу данного макро-кластера. Например, в отдельный микро-кластер были выделены генотипы, принадлежащие к сортотипу Рислинг. Также, интересным было и то, что аллели таких сортов как Совиньон и Пино белый, имеющие характерную окраску для белоягодных сортов, были разделены на два кластера, в которые вошли и аборигенные сорта. Это дает возможность предполодить, что, очевидно, такие сорта имеют также аллель, наиболее характерную для белоягодных сортов. В то же время как там был присовокуплен и сорт Мерло клон Грамотенко, у которого была выявлена не экспрессируемая аллель.
Таким образом, мы можем заключить, что кластеризация по принципу объединения данных прошла наиболее успешно, так как в таком случае наблюдалось наименьшее количество «примесей». Также важным является и то, что благодаря выполнению такой кластеризации, проливается свет на происхождение некоторых аборигенных сортов. Это также дает возможность определить
и аллели, характерные для групп сортов, что в будущем станет основой как для сортоизучения, так и для фундаментальных исследований, ставящих перед собой цель понимание функционирования метаболических путей.
ВЫВОДЫ
- Впервые выполнено секвенирование нуклеотидных последовательностей аллелей VvMybA1 и VvMybA2 у перспективных аборигенных сортов
и клоновых форм, отобранных и выращиваемых на территории Российской Федерации, что будет важным для проведения дальнейшей селекции опосредованной на получение конкретных агробиологических и производственных признаков. - По результатам секвенирования выбранных аллелей создана база данных ДНК-последовательностей изученных сортов (Прилолжения 1 и 2). Такие последовательности, по результатам сравнения с базой данных NCBI, показали наличие как уникального строения некоторых генотипов, так и выявили аллели, типичные для сортов с наличием или без наличия окраски.
- Дальнейшее сравнение нуклеотидных последовательностей с аминокислотными последовательностями, имеющимися в NCBI, показали наличие как схожих сиквенсов, так и присутствие изменения в их последовательности. Тем не менее, при сравнении генотипов и фенотипов растений нельзя сказать, чтобы такие изменения привели и к сдвигу фенотипических признаков. Не смотря на это, ценным было изучение совокупности строения нуклеотидных и аминокислотных последовательностей, которые показали такие интересные результаты как при кластеризации сортов Аркадия Розовая, Гелиос и Ливия. Данные сорта хоть и имели аллели, сходные по нуклеотидному строению с белоягодными сортами, тем не менее, очевидным является и наличие у них розовой окраски ягод. Или же, говоря иначе, слабой экспрессии генов.
- При изучении аборигенных сортов и строения их генов выявлены некоторые особенности строения. В частности, раскрыты однонуклеотидные полиморфизмы, которые привели к изменению в строении аминокислотной последовательности. Помимо этого, выявлены генотипы, причина потеря окраски ягод у которых вызвана движением ретротранспозона Gret-1 и также генотипы, у которых изменение окраски вызвано иными причинами.
- Изучение филогенитеческих взаимоотношений между исследованными сортами позволило выявить как очевидные, так и неожиданные факты. Например, такие сорта как Нимранг и Яй изюм белый. Очевидно, что генотип Яй изюм белый должен находиться либо же рядом с сортов Яй изюм розовый, либо же, что еще более очевидно, располагаться в кластере белоягодных сортов. Тем не менее, его аллель VvMybA1 имеет уникальную вставку, которая характерна для аллели mybA1-SUB, которая является функциональной. Также, его аллель при сравнении с базой данных является даже более характерной для окрашенных сортов, что у его «собрата» Яй изюм розового, что также, неожиданно. В свою очередь, сорт Нимранг, имеющих окраску ягод более слабую, чем у Аркадии Розовой, Гелиоса или Ливии, был отнесен к группе окрашенных сортов. Это, в свою очередь, также объясняется строением аллели, которая более характерна для окрашенных сортов.
- Остальные же сорта, как и предполагалось, были отнесены к принадлежащим им сортотипам, характерным для окрашенных и неокрашенных сортов. Не смотря на это, попадание к белоягодным сортам такой формы как Мерло клон Грамотенко указывает как и на то, что в процессе работы была выялвена «белая» аллель, так и на то, что такой стародавний сорт как Мерло имеет сложное происхождение, в которое были вовлечены гетерозиготные сорта, что подтверждается и предыдущими исследованиями.
ЛИТЕРАТУРА
- Айвазян, П. К. Селекция виноградной лозы / П. К. Айвазян, Е. Н. Докучаева. – Киев : Изд-во Украинской академии с.-х. наук, 1960. – С. 288–296.
- Альбертс, Б, Молекулярная биология клетки. В 3-х т. / Б. Альбертс,
Д. Брей, Дж. Льюис. – М. : Мир, 1994. – 517 с. - Барышева, И. А. Исследование внутрисортовой изменчивости ДНК винограда ПДРФ и ПЦР методами / И. А. Барышева, М. И. Тулаева, В. С. Чисников // Цитология и генетика. – 2003. – Т. 37, № 6. – С. 31–38.
- Беспалова, Л. А. Фотопериодическая чувствительность и молекулярное маркирование генов PPD и VRN в связи с селекцией сортов пшеницы альтернативного образа жизни / Л. А. Беспалова, В. А. Кошкин, Е. К. Потокина // Доклады Российской академии сельскохозяйственных наук. – 2010. – № 6. – С. 3–6.
- Бороевич, С. А. Принципы и методы селекции растений / С. А. Бороевич. – М. : Колос, 1984. – 344 с.
- Вавилов, Н. И. Генетика и селекция / Н. И. Вавилов // Избр. соч. – М. : Колос, 1966. – 560 с.
- Виноград. Всё о винограде [Электронный ресурс] / ред. В. Ю. Буря; Web-мастер В. Ю. Буря. – Электрон. дан. – Краснодар, 2012. – Режим доступа : http://vinograd.info.
- Волынкин, В. А. Триединство генетики, ампелографии и физиологии
в современной селекции винограда / В. А. Волынкин, В. А. Зленко, Н. П. Олейников // Интерактивная ампелография. – Краснодар, 2012. – С. 14–30. - Гераськин, С. А. Биологический контроль окружающей среды: генетический мониторинг : учеб. пособие / С. А. Гераськин, Е. И. Сарапульцева,
Л. В. Цаценко ; под ред. С. А. Гераськина, Е. И. Сарапульцевой. – М. : Академия, 2010. – 208 с. - Голодрига, П. Я. Понятие «клон» в виноградарстве / П. Я. Голодрига // Садоводство, виноградарство и виноделие Молдавии. – 1972. – № 11. –
С. 28–29. - Грамотенко, П. М. Клоновая селекция как метод повышения урожайности сортов винограда / П. М. Грамотенко // Сорт в виноградарстве. – 1962. –
№ 4. – С. 214–221. - Дивашук, М. Г. Идентичность генов короткостебельности RHT-11
И RHT-B1E / М. Г. Дивашук, А. В. Васильев, Л. А. Беспалова // Генетика. – 2012. – № 7. – С. 897. - Дорошенко, Н. П. Физиологическое обоснование применение препарата «Эмистим» при клональном микроразмножении винограда / Н. П. Дорошенко // Политематический сетевой электронный научный журнал Кубанского государственного аграрного университета (Научный журнал КубГАУ) [Электронный ресурс]. – Краснодар : КубГАУ, 2010. – № 58(04). – Режим доступа : http://ej.kubagro.ru/2010/04/pdf/32.pdf.
- Драгович, А. Ю. Сопоставление двух существующих каталогов аллелей глиадинкодирующих локусов у озимой мягкой пшеницы / А. Ю. Драгович,
В. Г. Зима, А. В. Фисенко // Генетика. – 2006. – Т. 42, № 8. – С. 1107–1116. - Драновский, В. А. Массовая и фитосанитарная селекция – необходимость современного виноградарства / В. А. Драновский, Л. П. Трошин // Виноград и вино России. – 1995. – № 4. – С. 20–23.
- Еремин, Г. В. Размножение клоновых подвоев персика / Г. В. Еремин, В. Г. Еремин // Политематический сетевой электронный научный журнал Кубанского государственного аграрного университета (Научный журнал КубГАУ) [Электронный ресурс]. – Краснодар : КубГАУ, 2010. – № 62(08). – Режим доступа : http://ej.kubagro.ru/2010/08/pdf/08.pdf.
- Звягин, А. С. Выделение ДНК из гербарных листьев Vitis vinifera L. /
А. С. Звягин // Политематический сетевой электронный научный журнал Кубанского государственного аграрного университета (Научный журнал КубГАУ) [Электронный ресурс]. – Краснодар : КубГАУ, 2010. – № 58(04). – Режим доступа : http://ej.kubagro.ru/2010/04/pdf/22.pdf. - Звягин, А. С. Выделение ДНК из листьев Vitis vinifera L. / А. С. Звягин, Л. П. Трошин // Политематический сетевой электронный научный журнал
кубанского государственного аграрного университета (Научный журнал КубГАУ) [Электронный ресурс]. – Краснодар : КубГАУ, 2010. – № 60(06). – Режим доступа : http://ej.kubagro.ru/2010/06/pdf/36.pdf. - Зеленский, Г. Л. Борьба с пирикуляриозом риса путем создания устойчивых сортов / Г. Л. Зеленский // Политематический сетевой электронный научный журнал Кубанский государственный аграрный университет (Научный журнал КубГАУ) [Электронный ресурс]. – Краснодар : КубГАУ, 2013. –
№ 85(01). – Режим доступа : http://ej.kubagro.ru/2013/01/pdf/48.pdf. - Зеленский, Г. Л. Изучение биоразнообразия сорных растений рода Echinochloa на территории Краснодарского края на основе морфологического
и молекулярно-генетического подходов / Г. Л. Зеленский // Труды Кубанского государственного аграрного университета. – 2008. – № 11. – С. 110–114. - Зеленский, Г. Л. Новый исходный материал для селекции риса на повышение продуктивности / Г. Л. Зеленский, М. В. Шаталова // Политематический сетевой электронный научный журнал Кубанского государственного аграрного вниверситета (Научный журнал КубГАУ) [Электронный ресурс]. – Краснодар : КубГАУ, 2013. – № 89(05). – Режим доступа : http://ej.kubagro.ru/2013/05/pdf/60.pdf.
- Зотов, В. В. Улучшить стандартные сорта винограда методом отбора / В. В. Зотов // Виноделие и виноградарство СССР. – 1956. – № 8. – С. 31–35.
- Изучение биоразнообразия возбудителя пирикуляриоза риса молекулярно-генетическими методами / Ж. М. Мухина, С. А. Волкова, Т. М. Коломиец, В. В. Тюрин // Труды Кубанского государственного аграрного университета. – 2008. – № 5(14). – С. 107–110.
- Калайджян, А. А. Российский солнечный цветок / А. А. Калайджян,
Л. В. Хлевной, Н. Н. Нещадим ; под ред. В. М. Пенчукова. – Краснодар : Совет. Кубань, 2007. – 351 с. - Кискин, П. Х. Филлоксера / П. Х. Кискин. – Кишинев : Штиинца, 1977. – 211 с.
- Клочнева, В. И. Идентификация видов, сортов и клонов винограда по белкам как маркерам генов / В. И. Клочнева, Л. П. Трошин, А. В. Шурхал. – М. : Типография ВАСХНИЛ, 1990. – 35 с.
- Клочнева, В. И. Межвидовая и внутривидовая изменчивость винограда по аллозимным локусам / В. И. Клочнева, Л. П. Трошин, А. В. Шурхал // Науч. тр. ВНИИВиПП «Магарач». – 1989. – № 9. – С. 109–114.
- Коваленко, Н. Н. Стимуляторы корнеобразования и их влияние на укоренение зеленых черенков клоновых подвоев плодовых культур / Н. Н. Коваленко, А. П. Кузнецова // Политематический сетевой электронный научный журнал Кубанского государственного аграрного университета (Научный журнал КубГАУ) [Электронный ресурс]. – Краснодар : КубГАУ, 2011. – № 73(09). – Режим доступа : http://ej.kubagro.ru/2011/09/pdf/26.pdf.
- Козаченко, Д. М. Клоновая и санитарная селекция винограда и некоторые элементы питомниководства во Франции / Д. М. Козаченко // Виноград
и вино России. – 1997. – № 6. – С. 56–58. - Лукомец, В. М. Селекционно-генетическое улучшение сои на юге европейской части России (обзор). Масличные культуры / В. М. Лукомец, А. В. Кочегура, С. В. Зеленцов // Научно-технический бюллетень Всероссийского научно-исследовательского института масличных культур. – 2012. – № 2 (151–152). – С. 207–210.
- Лукомец, В. М. Цитологический скрининг мужского гаметофита линий подсолнечника, устойчивых к имидазолиновым гербицидам / В. М. Лукомец,
Н. И. Бочкарев, Л. В. Цаценко // Труды Кубанского государственного аграрного университета. – 2012. – № 35. – С. 117–122. - Медведева, Н. И. Методические рекомендации по микроклональному размножению винограда in vitro / Н. И. Медведева, Н. В. Поливара, Л. П. Трошин // Политематический сетевой электронный научный журнал Кубанского государственного аграрного университета (Научный журнал КубГАУ) [Электронный ресурс]. – Краснодар : КубГАУ, 2010. – № 62(08). – Режим доступа : http://ej.kubagro.ru/2010/08/pdf/31.pdf.
- Медведева, Н. И. Особенности микроклонального размножения интродуцентов и клонов винограда / Н. И. Медведева, Н. В. Поливара, Л. П. Трошин // Политематический сетевой электронный научный журнал Кубанского государственного аграрного университета (Научный журнал КубГАУ) [Электронный ресурс]. – Краснодар : КубГАУ, 2008. – № 40(6). – Режим доступа : http://ej.kubagro.ru/2008/06/pdf/18.pdf.
- Милованов, А. В. Выделение ДНК при помощи PeqGOLD PLANT DNA mini KIT / А. В. Милованов, Л. П. Трошин // Политематический сетевой электронный научный журнал Кубанского государственного аграрного университета (Научный журнал КубГАУ) [Электронный ресурс]. – Краснодар : КубГАУ, 2013. – № 90(06). – Режим доступа : http://ej.kubagro.ru/2013/06/pdf/11.pdf.
- Мухина, Ж. М. Молекулярные маркеры и их использование в селекционно-генетических исследованиях / Ж. М. Мухина, Е. В. Дубина // Политематический сетевой электронный научный журнал Кубанского государственного аграрного университета (Научный журнал КубГАУ) [Электронный ресурс]. – Краснодар : КубГАУ, 2011. – № 66(02). – Режим доступа : http://ej.kubagro.ru/2011/02/pdf/09.pdf.
- Мухина, Ж. М. Создание внутригенных молекулярных маркеров риса для повышения эффективности селекционного и семеноводческого процессов /
Ж. М. Мухина, С. В. Токмаков, Ю. А. Мягких // Политематический сетевой электронный научный журнал Кубанский государственный аграрный университет (Научный журнал КубГАУ) [Электронный ресурс]. – Краснодар : КубГАУ, 2011. – № 67(03). – Режим доступа : http://ej.kubagro.ru/2011/03/pdf/19.pdf. - Овчинников, Ю. А. Биоорганическая химия / Ю. А. Овчинникова. – М. : Просвещение, 1987. – 816 c.
- Панарина, А. М. Изучение изменчивости признаков листа с целью выявления их ценности для ампелографических исследований / А. М. Панарина // Науч. тр. института «Магарач». – 1967. – Т. 16. – C. 167–182.
- Подваленко, П. П. Клоновая селекция – современная основа подъема продуктивности виноградников / П. П. Подваленко, А. С. Звягин, Л. П. Трошин // Политематический сетевой электронный научный журнал Кубанского государственного аграрного университета (Научный журнал КубГАУ) [Электронный ресурс]. – Краснодар : КубГАУ, 2009. – № 51(7). – Режим доступа : http://ej.kubagro.ru/2009/07/pdf/19.pdf.
- Романенко, А. А. Селекция гибридов кукурузы в Краснодарском НИИСХ / А. А. Романенко, А. И. Супрунов // Земледелие. – 2011. – № 2. –
С. 41–42. - Романенко, А. А. Селекция зерновых культур на устойчивость к абиотическим стрессорам / А. А. Романенко, Н. Ф. Лавренчук // Вестник РСАХН. – 2011. – № 1. – С. 17–21.
- Супрун, И. И. Апробация микросателлитных ДНК-маркеров для идентификации клоновых форм сортов яблони / И. И. Супрун, С. Н. Артюх,
С. В. Токмаков // Политематический сетевой электронный научный журнал Кубанского государственного аграрного университета (Научный журнал КубГАУ) [Электронный ресурс]. – Краснодар : КубГАУ, 2012. – № 84(10). – Режим доступа : http://ej.kubagro.ru/2012/10/pdf/21.pdf. - Супрун, И. И. Создание селекционных форм риса, несущих ген широкого спектра устойчивости к пирикуляриозу Pi-40, с использование методов ДНК-маркирования / И. И. Супрун, В. С. Ковалев, В. Н. Шиловский // Политематический сетевой электронный научный журнал Кубанский государственный аграрный университет (Научный журнал КубГАУ) [Электронный ресурс]. – Краснодар : КубГАУ, 2013. – № 86(02). – Режим доступа : http://ej.kubagro.ru/2013/02/pdf/18.pdf.
- Трошин, Л. П. Ампелографическая и селекционная научно-исследовательская работа Кубанского госагроуниверситета / Л. П. Трошин // Политематический сетевой электронный научный журнал Кубанского государственного аграрного университета (Научный журнал КубГАУ) [Электронный ресурс]. – Краснодар : КубГАУ, 2012. – № 07 (081). – Режим доступа : http://ej.kubagro.ru/2012/07/pdf/39.pdf.
- Трошин, Л. П. Ампелографические признаки в изучении таксономических отношений сортов Vitis vinifera sativa pontica Negr. / Л. П. Трошин,
В. И. Рисованная, А. И. Полулях // Труды Научного центра виноградарства и виноделия. – 1999. – С. 10–12. - Трошин, Л. П. Анализ наследственной информации винограда /
Л. П. Трошин // Виноград и вино России. – 1997. – № 1. – С. 17–19. - Трошин, Л. П. Биометрическая оценка полиморфизма сортогрупп винограда Пино и Рислинг по морфологическим признакам листьев среднего яруса кроны / Л. П. Трошин, Е. В. Луценко, П. П. Подваленко // Политематический сетевой электронный научный журнал Кубанского государственного аграрного университета (Научный журнал КубГАУ) [Электронный ресурс]. – Краснодар : КубГАУ, 2009. – № 52(8). – Режим доступа : http://ej.kubagro.ru/2009/08/pdf/01.pdf.
- Трошин, Л. П. Виноград: иллюстрированный каталог. Районированные, перспективные, тиражные сорта / Л. П. Трошин, П. П. Радчевский. – Ростов н/Д. : Феникс, 2010. – 271 с.
- Трошин, Л. П. Изменчивость признаков листа сортов винограда Vitis vinifera pontica balcanica Negr. / Л. П. Трошин, В. И. Рисованная, А. А. Полулях // Виноградарство и виноделие. – 1996. – № 1. – С. 15–20.
- Трошин, Л. П. Использование биометрической оценки морфологических признаков клонов для идентификации генотипов сортогрупп Мерло /
Л. П. Трошин, А. С. Звягин // Политематический сетевой электронный научный журнал Кубанского государственного аграрного университета (Научный журнал КубГАУ) [Электронный ресурс]. – Краснодар : КубГАУ, 2008. – № 38(4) – Режим доступа : http://ej.kubagro.ru/2008/04/pdf/10.pdf. - Трошин, Л. П. Методология клоновой селекции винограда / Л. П. Трошин // Юбилейный тематический сборник научных трудов. Ч. 2. Виноградарство. – Краснодар, 2001. – С. 92–94.
- Трошин, Л. П. О значении метода клоновой селекции винограда, разработанного профессором А. С. Мержанианом / Л. П. Трошин // Политематический сетевой электронный научный журнал Кубанского государственного аграрного университета (Научный журнал КубГАУ) [Электронный ресурс]. – Краснодар : КубГАУ, 2010. – № 57(03). – Режим доступа : http://ej.kubagro.ru/2010/03/pdf/16.pdf.
- Трошин, Л. П. Оценка таксономических отношений сортов V. v. s. p. balcanica Negr. и V. v. s. p. meridionali-balcanica Trosh. по морфометрическим признакам листа / Л. П. Трошин, А. А. Полулях, В. И. Рисованная // Виноград
и вино России. – 1998. – № 3. – C. 41–42. - Трошин, Л. П. Районированные сорта винограда России / Л. П. Трошин, П. П. Радчевский. – Краснодар : ООО «Вольные мастера», 2004. – 176 с.
- Трошин, Л. П. Сортимент винограда России / Л. П. Трошин, П. П. Радчевский // Виноделие и виноградарство. – 2001. – № 3. – С. 24–25.
- Трошин, Л. П. Статистический анализ количественных признаков популяции винограда сорта Рислинг / Л. П. Трошин, И. А. Суятинов, М. А. Чупраков // Пути совершенствования питомниководства и селекционного процесса в виноградарстве / Труды ВНИИВиПП «Магарач». – Ялта, 1986. – Т. 24. –
С. 77–86. - Цаценко, Л. В. Методология научной агрономии : курс лекций /
Л. В. Цаценко, Н. А. Щербаков. – Краснодар : КубГАУ, 2012. – С. 10–11. - Цаценко, Л. В. Молекулярная биология : учеб. пособие / Л. В. Цаценко, Ю. С. Бойко, Д. В. Крутенко. – Краснодар : КубГАУ, 2007. – С. 14–15.
- Цаценко, Л. В. Цитология : учеб. пособие / Л. В. Цаценко, Ю. С. Бойко. – Краснодар : КубГАУ, 2007. – 27 с.
- 1H NMR metabolite fingerprints of grape berry: Comparison of vintage and soil effects in Bordeaux grapevine growing areas / G. E. Pereira [et al.] // Analytica Chimica Acta. – 2006. – Vol. 563, № 1–2. – Р. 346–352.
- A 48 SNP set for grapevine cultivar identification / J. A. Cabezas, J. Ibáñez, D. Lijavetzky, D. Vélez, G. Bravo, V. Rodríguez, I. Carreño, A. M. Jermakow,
J. Carreño, L. Ruiz-García, M. R. Thomas // BMC plant biology. – 2011. –
Vol. 11(1). – Р. 153. - Abubakkar, M. Extraction of DNA suitable for PCR applications from mature leaves of Mangifera indica L. / M. Abubakkar, A. Khan, H. Cheema // Journal of Zhejiang University. – 2012. – Vol. 13. – Р. 239–243.
- Allelic variation in the VvMYBA1 and VvMYBA2 domestication genes in natural grapevine populations (Vitis vinifera subsp. sylvestris) / D. Carrasco [et al.] // Plant Systematics and Evolution. – 2015. – Vol. 301, № 6. – Р. 1613–1624.
- Ampelographic and genetic characterization of Croatian grapevine varieties / E. Maletić, I. Pejić, J. K. Kontić, D. Zdunić, D. Preiner, S. Šimon, D. Stupić // VITIS – Journal of Grapevine Research. – 2015. – Vol. 4(54). – Р. 93–98.
- An extensive study of the genetic diversity within seven French wine grape variety collections / F. Pelsy [et al.] // Theoretical and Applied Genetics. – 2010. – Vol. 120, № 6. – Р. 1219–1231.
- A PCR-based diagnostic tool for distinguishing grape skin color mutants /
S. Giannetto [et al.] // Plant Science. – 2008. – Vol. 175, № 3. – Р. 402–409. - Ashenfelter, O. Predicting the prices and quality of Bordeaux wines /
O. Ashenfelter // The Economic Journal. – 2008. – № 529. – Р. 174–184. - Ashenfelter, O. The economics of wine, weather, and climate change /
O. Ashenfelter, K. Storchmann // Review of Environmental Economics and Policy. – 2016. – № 1. – Р. 25–46. - Ashenfelter, O. Using a hedonic model of solar radiation to assess the economic effect of climate change: the case of Mosel Valley vineyards / O. Ashenfelter, K. Storchmann // National Bureau of Economic Research. – 2006. – № 12. –
Р. 122–130. - A skin color mutation of grapevine, from black-skinned Pinot Noir to white-skinned Pinot Blanc, is caused by deletion of the functional VvmybA1 allele / H. Yakushiji, S. Kobayashi, N. Goto-Yamamoto, S. T. Jeong, T. Sueta, N. Mitani,
A. Azuma // Bioscience, biotechnology, and biochemistry. – 2006. – Vol. 70(6). –
Р. 1506–1508. – doi.org/10.1271/bbb.50647. - Atak, A. Molecular and ampelographic characterization of some grape hybrids (Vitis vinifera L.) / A. Atak, A. Altindisli, A. Fuat // African Journal of Agricultural Research. – 2012. – Vol. 7. – P. 456–466.
- Audeguin, L. L’experimtntation des clones des de vigne en France. Etat des lilex, metohodologie et perspectives / L. Audeguin, R. Boidron, P. Bloy // XXIV Weltkongress fur Rebe und Wein. – 1997. – Vol. 10. – P. 42–52.
- Azuma, A. VvmybA1 genotype determines grape skin color / A. Azuma,
S. Kobayashi, H. Yakushiji // Vitis. – 2007. – Vol. 46. – P. 154–155. - Balthazard, J. Clonal selection and gene pool preservation of traditional grape cultivars / J. Balthazard, P. Huglin // Proceedings of the Third International symposium on grape breeding. – 1980. – P. 63–64.
- Benjak, A. Genome-wide analysis of the «Cut-and-Paste» transposons of grapevine / A. Benjak, A. Forneck, J. Casacuberta // PLoS ONE. – 2008. – Vol. 3. – P. 9–10.
- Berry quality and antioxidant properties in Vitis vinifera cv. Tempranillo as affected by clonal variability, mycorrhizal inoculation and temperature / N. Torres
[et al.] // Crop and Pasture Science. – 2016. – Vol. 67, № 9. – Р. 961–977. - Bodor, P. The separation of grapevine (Vitis vinifera L.) Conculta members with molecular markers / P. Bodor, G. Bisztray, A. Pedryc // Proceedings of International Ph. D. Students Conference. Brno. – 2008. – P. 118–123.
- Bogs, J. The grapevine transcription factor VvMYBPA1 regulates proanthocyanidin synthesis during fruit development / J. Bogs, F. Jaffe, A. Takos // Plant Physiology. – 2007. – Vol. 143. – P. 1347–1361.
- Boss, P. K. Expression of anthocyanin biosynthesis pathway genes in red and white grapes / P. K. Boss, C. Davies, S. P. Robinson // Plant molecular biology. – 1996. – Vol. 32, №. 3. – Р. 565–569.
- Boulton, R. The copigmentation of anthocyanins and its role in the color of red wine: A critical review / R. Boulton // American journal of enology and viticulture. – 2001. – № 2. – Р. 67–87.
- Brown, N. P. View: a web-compatible database search or multiple alignment viewer / N. P. Brown, C. Leroy, C. M. Sander // Bioinformatics (Oxford, England). – 1998. – Vol. 14, № 4. – Р. 380–381.
- Cadle-Davidson, M. Radiation of the grape retroelement (GRET1) in Vitis spp. of the grapevine germplasm repository / M. Cadle-Davidson, C. Owens // Grape Genetics Research. – 2007. – Vol. 10. – P. 24.
- Caetano-Anollks, G. Fast and sensitive silver staining of DNA in polyacrylamide gels / G. Caetano-Anollks, P. Gresshoff // Analyt. Bioch. – 1991. –
Vol. 15. – P. 80–83. - Carcamo, C. Detection of polymorphism in ancient Tempranillo clones using microsatellite and retrotransposon markers / C. Carcamo, I. Provedo, R. Arroyo-Garcha // Iranian Journal of Biotechnology. – 2010. – Vol. 8. – P. 73–75.
- Carputo, D. Use of SSR and retrotransposon-based markers to interpret the population structure of native grapevines from southern Italy Clizia Villano /
D. Carputo, L. Frusciante, X. Santoro // Springer Science. – 2014. – Vol. 131. –
P. 245–250. - Characterization of single nucleotide polymorphism in Tunisian grapevine genome and their potential for population genetics and evolutionary studies /
L. Riahi, N. Zoghlami, A. Fournier-Level, A. Dereeper, L. Le Cunff, V. Laucou,
A. Mliki, P. This // Genetic resources and crop evolution. – 2013. – Vol. 60(3). –
Р. 1139–1151. – doi.org/10.1007/s10722-012-9910-y. - Choudhary, K. Protocol for isolation of genomic DNA from dry and fresh leaves of vigna species suitable for RAPD and restriction digestion / K. Choudhary, N. Mathur, O. Choudhary // Adv. in Biological Research. – 2008. – Vol. 2. –
P. 83–89. - Color recovery in berries of grape (Vitis vinifera L.) ‘Benitaka’, a bud sport of ‘Italia’, is caused by a novel allele at the VvmybA1 locus / A. Azuma [et al.] // Plant Science. – 2009. – № 4. – С. 470–478.
- Derent, X. Clonal selection of grapes / X. Derent, W. Deling, Z. Guoliang // Acta Ноrtic. Sin. – 1991. – Vol. 18(2). – P. 121–125.
- Dettweiler, E. The two Vitis databases as tools for germplasm management / E. Dettweiler, E. Rudolf // Vitis international variety catalogue (http://www.dainet.de/
genres/idb/vitis), European Vitis Database (http://www.dainet.de/eccdb/vitis/). – VIII International Conference on Grape Genetics and Breeding. – Kecskemet, 2002. –
Р. 505–509. – doi.org/10.17660/ActaHortic.2003.603.66. - Duchêne, E. Variation of linalool and geraniol content within two pairs of aromatic and non‐aromatic grapevine clones / E. Duchêne // Australian Journal of Grape and Wine Research. – 2009. – Vol. 15(2). – Р. 120–130. – doi.org/10.1111/j.1755-0238.2008.00039.x.
- Efficiency optimization of CRISPR/Cas9-Mediated targeted mutagenesis in grape / F. Ren, C. Ren, Z. Zhang, W. Duan, D. Lecourieux, S. H. Li, Z. C. Liang // Frontiers in plant science. – 2019. – Vol. 10(1). – Р. 612. – doi.org/10.3389/fpls.2019.00612.
- Evolution of the VvMybA gene family, the major determinant of berry colour in cultivated grapevine (Vitis vinifera L.) / A. Fournier-Level [et al.] // Heredity. – 2010. – Vol. 104, № 4. – Р. 351.
- Fatahi, R. Characterization of Iranian grapevine cultivars using microsatellite markers / R. Fatahi, A. Ebadi, N. Bassil // Vitis. – 2003. – № 42(4). – P. 185–192.
- Fogarty, J. J. To save or savor: A review of approaches for measuring wine as an investment / J. J. Fogarty, R. Sadler // Journal of Wine Economics. – 2014. –
№ 3. – Р. 225–248. - Fogarty, J. J. Wine investment and portfolio diversification gains /
J. J. Fogarty // Journal of Wine Economics. – 2010. – № 1. – Р. 119–131. - Forneck A. Plant breeding: clonality – a concept for stability and variability during vegetative propagation / A. Forneck // Progress in botany. – Springer, Berlin, Heidelberg, 2005. – Р. 164–183.
- Genetic diversity and geographical dispersal in grapevine clones revealed by microsatellite markers / X. Moncada, F. Pelsy, D. Merdinoglu, P. Hinrichsen // Genome. – 2006. – Vol. 49(11). – Р. 1459–1472. – doi.org/10.1139/g06-102.
- Genomic and genetic analysis of Myb-related genes that regulate anthocyanin biosynthesis in grape berry skin / A. Azuma [et al.] // Theoretical and Applied Genetics. – 2008. – Vol. 117, № 6. – С. 1009–1019.
- Genotype identification and inference of genetic relatedness among different purpose grape varieties and rootstocks using microsatellite markers / S. Ajitpal, K. Krishan, I. Manav, I. Gill // African Journal of Biotechnology. – 2013. – Vol. 12. – P. 134–141.
- Gil, J. Extraction of genomic DNA from the lipid–polysaccharide– and polyphenol rich coconut (Cocos nucifera L.) / J. Gil, C. Angeles, A. Laurena // Plant Molecular Biology Reporter. – 2005. – Vol. 23. – P. 297.
- Ginwal, H. S. An efficient genomic DNA isolation protocol for RAPD and SSR analysis in Acorus calamus L. / H. S. Ginwal, N. Mittal // Journal of Biotechnology. – 2010. – Vol. 9. – P. 213–216.
- Grapevine field experiments reveal the contribution of genotype, the influence of environment and the effect of their interaction (G× E) on the berry transcriptome / S. Dal Santo [et al.] // The Plant Journal. – 2018. – Vol. 93, № 6. –
Р. 1143–1159. - Grapevine Non-Vinifera Genetic Diversity Assessed by SSR Markers as
a Starting-Point for New Rootstock Breeding Programs / D. Migliaro, G. De Lorenzis, G. S. Di Lorenzo, B. De Nardi, M. Gardiman, O. Failla, L. Brancadoro,
M. Crespan // American Journal of Enology and Viticulture. – 2019. – Vol. 70(4). – Р. 390–397. – doi.org/10.5344/ajev.2019.18054. - Grapevine powdery mildew resistance and susceptibility loci identified on a high-resolution SNP map / P. Barba, L. Cadle-Davidson, J. Harriman, J. C. Glaubitz, S. Brooks, K. Hyma, B. Reisch // Theoretical and applied genetics. – 2014. – Vol. 127(1). – Р. 73–84. – DOI : 10.1007/s00122-013-2202-x.
- Gray, D. J. Precision breeding of grapevine (Vitis vinifera L.) for improved traits / D. J. Gray, Z. T. Li, S. A. Dhekney // Plant science. – 2014. – Vol. 228(1). –
Р. 3–10. – doi.org/10.1016/j.plantsci.2014.03.023. - Guidoni, S. Effects of seasonal and agronomical practices on skin anthocyanin profile of Nebbiolo grapes / S. Guidoni, A. Ferrandino, V. Novello // American Journal of Enology and Viticulture. – 2008. – Vol. 59, № 1. – Р. 22–29.
- Haplotype composition at the color locus is a major genetic determinant of skin color variation in Vitis× labruscana grapes / A. Azuma, Y. Udo, A. Sato,
N. Mitani, A. Kono, Y. Ban, H. Yakushiji, Y. Koshita, S. Kobayashi // Theoretical and Applied Genetics. – 2011. – Vol. 122, № 7. – Р. 1427–1438. - High through put SNP discovery and genotyping in grapevine (Vitis vinifera L.) by combining a re-sequencing approach and SNPlex technology / D. Lijavetzky, J. A. Cabezas, A. Ibáñez, V. Rodríguez, J. M. Martínez-Zapater // BMC genomics. – 2007. – Vol. 8(1). – Р. 424. – doi.org/10.1186/1471-2164-8-424.
- Huglin, P. L’Evaluation genetique et sanitaire du materiel clonal de la vigne / P. Huglin, R. Guillot, C. Valat // Bull. O. I. V. – 1980. – Vol. 53. – P. 857–882.
- Ibrahim, R. A modified CTAB protocol for DNA extraction from young flower petals of some medicinal plant species / R. Ibrahim // Geneconserve. – 2011. – № 10(40). – P. 165–182.
- Impact of clonal variability in Vitis vinifera Cabernet franc on grape composition, wine quality, leaf blade stilbene content, and downy mildew resistance /
C. Van Leeuwen [et al.] // Journal of agricultural and food chemistry. – 2012. –
Vol. 61, № 1. – Р. 19–24. - Investigation of VvMybA1 and VvMybA2 berry color genes in’Aglianico’biotypes / G. De Lorenzis [et al.] // Progress in Vitis Vinfera diversity evaluation and use. – Bundesanstalt fur Zuchtungsforschung an Kulturpflanzen, 2015, 7–9 October. – Vol. 54, № special issue. – Р. 43–44.
- Jalview Version 2 – a multiple sequence alignment editor and analysis workbench / A. M. Waterhouse, J. B. Procter D. M., Martin, M. Clamp, G. J. Barton // Bioinformatics. – 2009. – Vol. 25, № 9. – Р. 1189–1191.
- Jones, D. T. A model recognition approach to the prediction of all-helical membrane protein structure and topology / D. T. Jones, W. R. Taylor, J. M. Thornton // Biochemistry. – 1994. – Vol. 33, № 10. – Р. 3038–3049.
- Kalendar, R. Analysis of plant diversity with retrotransposon–based molecular markers / R. Kalendar, A. Flavell // Heredity. – 2011. – Vol. 106. – P. 520–530.
- Kalendar, R. iPBS: a universal method for DNA fingerprinting and retrotransposon isolation / R. Kalendar, K. Antonius, P. Smýkal // Theor. Appl. Genet. – 2010. – Vol. 121. – Р. 1419–1430.
- Kalendar, R. IRAP and REMAP for retrotransposon – based genotyping and fingerprinting / R. Kalendar, A. Schulman // Nature Protocols. – 2006. –
Vol. 11. – P. 2478–2484. - Kalendar, R. IRAP and REMAP: two new retrotransposon – based DNA fingerprinting techniques / R. Kalendar, T. Grob, M. Regina // Theor. Appl. Genet. – 1998. – Vol. 109. – P. 704–711.
- Kalendar, R. The Use of Retrotransposon–Based molecular markers to analyze genetic diversity / R. Kalendar // Field Veg. Crop Res. – 2011. – Vol. 48. –
Р. 261–274. - Kalendar, R. Use of retrotransposon-derived genetic markers to analyse genomic variability in plants / R. Kalendar, A. Amenov, A. Daniyarov // Functional plant biology. – 2019. – Vol. 46(1). – Р. 15–29. – doi.org/10.1071/FP18098.
- Ketan, P. Detection of Proteins in Polyacrylamide Gels Using an Ultrasensitive Silver Staining Technique / P. Ketan, D. Easty, M. Dunn // Methods in Molecular Biology. – 1988. – Vol. 3. – P. 159–168.
- Kobayashi, S. Association of VvmybA1 gene expression with anthocyanin production in grape (Vitis vinifera) skin-color mutants / S. Kobayashi, N. Goto-Yamamoto, H. Hirochika // Japanese Society of Horticultural Science. – 2005. –
№ 74. – Р. 196–203. - Kobayashi, S. Retrotransposon-induced mutations in grape skin color /
S. Kobayashi, N. Goto-Yamamoto, H. Hirochika // Science. – 2004. – Vol. 304,
№ 5673. – Р. 982. - Krizamn, M. Robust CTAB-activated charcoal protocol for plant DNA extraction / M. Krizamn, J. Jakse, D. Baricevic // Agriculturae Slovenica. – 2006. –
Vol. 87. – P. 427–433. - Kumar, S. MEGA7: molecular evolutionary genetics analysis version 7.0 for bigger datasets / S. Kumar, G. Stecher, K. Tamura // Molecular biology and evolution. – 2016. – Vol. 33, № 7. – Р. 1870–1874.
- Lawless, H. T. Sensory evaluation of food: principles and practices /
H. T. Lawless, H. Heymann. – Springer Science & Business Media, 2010. - Lefort, F. The Greek Vitis database: a multimedia web-backed genetic database for germplasm management of Vitis resources in Greece / F. Lefort, K. Roubelakis-Angelakis // Journal of Wine research. – 2000. – Vol. 11(3). – Р. 233–242. – doi.org/10.1080/713684241.
- Lemke, L. A simple, inexpensive and environmentally friendly method for high through put DNA extraction from grapevine (Vitis spp.) / L. Lemke, M. Rex,
E. Zyprian // Vitis. – 2011. – Vol. 14. – P. 4–6. - Life technologies company [Электронный ресурс] / Thermo Fisher Scientific Inc. – Электрон. дан. – Waltham : Thermo Fisher Scientific Inc., 2011. – Режим доступа : http://www.lifetechnologies.com.
- Ligia, G. Assessment of diversity in grapevine gene pools from Romania and Republic of Moldova, based on SSR markers analysis / G. Ligia // Horticulture. – 2012. – Vol. 56. – P. 172.
- Loureiro, M. D. Clonal preselection of grapevine cultivars of the appellation «Cangas Quality Wine» / M. D. Loureiro, P. Moreno-Sanz, B. Suarez // Hort. Sci. – 2011. – Vol. 38. – P. 71–80.
- Mannini, F. Heat-treated V. Virus-infected grapevine clones: agronomical and enological modifications / F. Mannini, G. Vincenzo, C. Rino // International Symposium on the Importance of Varieties and Clones in the production of Quality Wine. – Kecskemét, 1997. – Р. 473.
- Maras, V. Research of origin and work on clonal selection of Montenigrin grapevine varieties Vranac and Kratosija / V. Maras, M. Tomic, V. Kodzulovic // Agroznanje. – 2012. – Vol. 13. – Р. 103–112.
- Maria, S. Grape microsatellite markers: Sizing of DNA alleles and genotype analysis of some grapevine cultivars / S. Maria, C. Frisinghelli // Vitis. – 1998. – Vol. 37. – P. 79–82.
- Masset, P. Wine as an alternative asset class / P. Masset, C. Henderson // Journal of Wine Economics. – 2010. – № 1. – Р. 87–118.
- Matus, J. T. Analysis of the grape MYB R2R3 subfamily reveals expanded wine quality-related clades and conserved gene structure organization across Vitis and Arabidopsis genomes / J. T. Matus, F. Aquea, P. Arce-Johnson // BMC plant biology. – 2008. – № 1. – Р. 83.
- McGinnis, S. BLAST: at the core of a powerful and diverse set of sequence analysis tools / S. McGinnis, T. L. Madden // Nucleic acids research. – 2004. –
Vol. 32, № suppl_2. – Р. W20–W25. - Moisy, C. The Tvv1 retrotransposon family is conserved between plant genomes separated by over 100 million years / C. Moisy, A. Schulman, R. Kalendar // Theor. Appl. Genet. – 2014. – Vol. 127. – P. 1223–1235.
- Molecular characterization of berry skin color reversion on grape somatic variants / V. Ferreira [et al.] // Journal of Berry Research. – 2018. – № 3. –
Р. 147–162. - Molecular genetics of berry color variation in table grape / D. Lijavetzky [et al.] // Molecular Genetics and Genomics. – 2006. – Vol. 276, № 5. – Р. 427–435.
- Molecular tools for clone identification: the case of the grapevine cultivar ‘Traminer’ / S. Imazio, M. Labra, F. Grassi, M. Winfield, M. Bardini, A. Scienza // Plant Breeding. – 2002. – Vol. 121(6). – Р. 531–535. – doi.org/10.1046/j.1439-0523.2002.00762.x.
- Muhammad, L. A simple and efficient method for DNA extraction from grapevine cultivars, Vitis species and Ampelopsis / L. Muhammad, Y. Guang-Ning, F. Norman // Plant Molecular Biology Reporter. – 1994. – № 12(1). – P. 6–13.
- Murshudov, G. N. Refinement of macromolecular structures by the maximum-likelihood method / G. N. Murshudov, A. A. Vagin, E. J. Dodson // Acta Crystallographica Section D: Biological Crystallography. – 1997. – Vol. 53, № 3. –
Р. 240–255. - Myb-related genes of the Kyoho grape (Vitis labruscana) regulate anthocyanin biosynthesis / S. Kobayashi [et al.] // Planta. – 2002. – Vol. 215, № 6. – Р. 924–933.
- National Center for Biotechnology Information [Электронный ресурс] / National Center for Biotechnology Information. – Электрон. дан. – Rockville Pick : U. S. National Library of Medicine, 2011. – Режим доступа : www.ncbi.nlm.nih.gov/.
- Optimizing Agrobacterium-mediated transformation of grapevine / Z. Li,
S. Dhekney, M. Dutt, M. Van Aman, J. Tattersall, K. T. Kelley, D. J. Gray // In Vitro Cellular & Developmental Biology Plant. – 2006. – Vol. 42(3). – Р. 220–227. - Parr, W. V. The nose knows: Influence of color on perception of wine aroma / W. V. Parr, K. Geoffrey White, D. A. Heatherbell // Journal of Wine Research. – 2003. – № 2–3. – Р. 79–101.
- Pejic, I. Conservation, evaluation and revitalization of native grapevine varieties in Croatia / I. Pejic, E. Maletic // Mitteilungen Klosterneuburg. – 2010. –
Vol. 60. – P. 363–368. - Pelsy, F. Molecular and cellular mechanisms of diversity within grapevine varieties / F. Pelsy // Heredity. – 2010. – Vol. 104(4). – Р. 331. – doi.org/10.1038/hdy.2009.161.
- PeqLab company [Электронный ресурс] / VWR Company; Web-мастер VWR International GmbH. – Электрон. дан. – Erlangen : Life Science Competence Center, 2011. – Режим доступа : http://www.peqlab.com/wcms/en/index.php.
- Polyphenol content and total antioxidant activity of vini novelli (young red wines) / N. Pellegrini [et al.] // Journal of agricultural and food chemistry. – 2000. – № 3. – Р. 732–735.
- Puchooa, D. A simple, rapid and efficient method for the extraction of genomic DNA from lychee / D. Puchooa // African Journal of Biotechnology. – 2004. – Vol. 3. – P. 253–255.
- Purple-leaved Ficus lyrata plants produced by overexpressing a grapevine VvMybA1 gene / J. Zhao, Z. T. Li, J. Chen, R. J. Henny, D. J. Gray, J. Chen // Plant cell reports. – 2013. – Vol. 32(11). – Р. 1783–1793. – doi.org/10.1007/s00299-013-1491-5.
- Quality criteria and targets for clonal selection in grapevine / E. Rühl
[et al.] // I International Symposium on Grapevine Growing, Commerce and Research 652. – 2003. – Р. 29–33. - Quantitative genetic bases of anthocyanin variation in grape (Vitis vinifera L. ssp. sativa) berry: a quantitative trait locus to quantitative trait nucleotide integrated study / A. Fournier-Level [et al.] // Genetics. – 2009. – Vol. 183, № 3. –
Р. 1127–1139. - Randall, K. Primer-directed enzymatic amplification of DNA with a thermostable DNA polymerase / K. Randall, H. David, S. Susanne // Science. – 1988. – Vol. 29. – P. 487–491.
- Regner, F. Genetic markers for the identification of varieties and clones as a guarantee of quality / F. Regner, K. Sefc, A. Stadlbauer // Acta Horticulturae. – 1998. – Vol. 473. – P. 49–61.
- Regner, F. Highly variable Vitis microsatellite loci for the identification of Pinot Noir clones / F. Regner, R. Hack, J. L. Santiago // Vitis. – 2006. – Vol. 45. –
P. 85–91. - Repetitive DNA of grapevine: classes present and sequences suitable for cultivar identification / M. R. Thomas, S. Matsumoto, P. Cain, N. S. Scott // Theor. Appl. Genet. – 1993. – Vol. 86. – P. 173–180.
- Rusjan, D. Genetic and Phenotypic Diversity and Relations Between Grapevine Varieties: Slovenian Germplasm / D. Rusjan // The Mediterranean Genetic Code – Grapevine and Olive. – 2013. – Vol. 3. – P. 147–176.
- Salmaso, M. Gene pool variation and phylogenetic relationships of an indigenous northeast Italian grapevine collection revealed by nuclear and chloroplast SSRs / M. Salmaso, R. D. Valle, M. Lucchin // Genome. – 2008. – Vol. 51(10). –
Р. 838–855. – doi.org/10.1139/G08-064. - Sanger, F. DNA sequencing with chain–terminating inhibitors / F. Sanger,
S. Nicklen, A. R. Coulson // Proc. Natl. Acad. Sci. USA. – 1977. – Vol. 12. – P. 563–567. - Sanning, L. W. Bordeaux wine as a financial investment / L. W. Sanning, S. Shaffer, J. M. Sharratt // Journal of Wine Economics. – 2008. – № 1. – Р. 51–71.
- Sant’Ana, G. C. Comparison of a retrotransposon-based marker with microsatellite markers for discriminating accessions of Vitis vinifera / G. C. Sant’Ana, J. L. Ferreira, H. S. Rocha // Genetics and Molecular Research. – 2012. – Vol. 11. – P. 1507–1525.
- Schulman, A. The Application of LTR Retrotransposons as Molecular Markers in Plants / A. Schulman, A. Flavell, E. Paux // Methods in Molecular Biology. – 2012. – Vol. 59. – P. 115–153.
- Shahzadi, I. Optimization of DNA extraction from seeds and fresh leaf tissues of wild marigold (Tagetes minuta) for polymerase chain reaction analysis /
I. Shahzadi, R. Ahmed, A. Hassan // Genet. Mol. Res. – 2010. – Vol. 9. –
P. 386–393. - Sievers, F. Clustal Omega, accurate alignment of very large numbers of sequences / F. Sievers, D. G. Higgins // Multiple sequence alignment methods. – Humana Press, Totowa, NJ, 2014. – Р. 105–116.
- Silvia, V. A reference integrated map for cultivated grapevine (Vitis vinifera L.) from three crosses, based on 283 SSR and 501 SNP–based markers / V. Silvia, T. Michela, C. Giuseppina // Springer-Verlag. – 2008. – Vol. 85. – P. 34–45.
- Singleton, V. L. Wine flavor and phenolic substances / V. L. Singleton,
A. C. Noble // Phenolic, sulfur, and nitrogen compounds in food flavors. – 1976. –
№ 2. – Р. 47–70. - Skoog, D. A. Principles of Instrumental Analysis. 6th ed. ThomsonBrooks / D. A. Skoog, F. J. Holler, S. R. Crouch. – Belmont : ColePublishing, 2007. – P. 573.
- Spontaneous variation regarding grape berry skin color: A comprehensive study of berry development by means of biochemical and molecular markers / V. Ferreira [et al.] // Food research international. – 2017. – Р. 149–161.
- Swanepoel, J. J. The influence of rootstock on the rooting pattern of the grapevine / J. J. Swanepoel, J. M. Southey // South African Journal of Enology and Viticulture. – 1989. – Vol. 10, № 1. – Р. 23–28.
- Tassie, L. Vine identification – knowing what you have / L. Tassie // Wine Research and Development. – 2010. – Vol. 19. – P. 56–59.
- Tautz, D. Cryptic simplicity in DNA is a major source of genetic variation / D. Tautz, M. Trick, G. Dover // Nature. – 1986. – Vol. 322. – P. 652–653.
- The Greek Vitis Database [Электронный ресурс] / University of Crete. Wed-мастер S. Monachou. – Электрон. дан. – Heraklion : University of Crete, 2010. – Режим доступа : http://gvd.biology.uoc.gr/gvd/contents/general-info/02a.html.
- The state-of-the-art of grapevine biotechnology and new breeding technologies (NBTS) / L. Dalla Costa, M. Malnoy, D. Lecourieux, L. Deluc, F.Ouaked-Lecourieux, M. R. Thomas, L. J. Torregrosa // OENO One. – 2019. – Vol. 53(2). –
Р. 189–212. – doi.org/10.20870/oeno-one.2019.53.2.2405. - This, P. Development of stable RAPD markers for the identification of grapevine rootstocks and the analysis of genetic relationships / P. This, C. Cuisset,
J. M. Boursiquot // Am. J. Enol. Vitic. – 1997. – Vol. 48. – P. 492–501. - This, P. Historical origins and genetic diversity of wine grapes / P. This,
T. Lacombe, M. R. Thomas // Trends in Genetics. – 2006. – Vol. 22(9). – Р. 511–519. – doi.org/10.1016/j.tig.2006.07.008. - This, P. Towards marker-assisted selection for seedlessness in grapevine /
P. This, F. Lahogue, A. F. Adam-Blondon // Acta Hort. – 2000. – Vol. 528. – P. 221–229. - Thomas, M. R. Microsatellite repeats in grapevine reveal DNA polymorphism when analysed as sequence-tagged sites (STSs) / M. R. Thomas, N. S. Scott // Theor. Appl. Genet. – 1993. – Vol. 86. – P. 985–990.
- Thomas, M. R. Sequence-tagged site markers in grapevine and citrus /
M. R. Thomas, N. S. Scott, R. Botta // Journal of the Japanese Society for Horticultural. – 1998. – Vol. 67. – P. 1189–1192. - Towards a scientific interpretation of the terroir concept: plasticity of the grape berry metabolome / A. Anesi [et al.] // BMC plant biology. – 2015. – Vol. 15, № 1. – Р. 191.
- Transgenic plantlets of ‘Chancellor’ grapevine (Vitis sp.) from biolistic transformation of embryogenic cell suspensions / J. R. Kikkert, D. Hébert-Soulé,
P. G. Wallace, M. J. Striem, B. I. Reisch // Plant Cell Reports. – 1996. – Vol. 15(5). – Р. 311–316. - Transposon‐induced gene activation as a mechanism generating cluster shape somatic variation in grapevine / L. Fernandez, L. Torregrosa, V. Segura,
A. Bouquet, J. M. Martinez‐Zapater // The Plant Journal. – 2010. – Vol. 61(4). –
Р. 545–557. – doi.org/10.1111/j.1365-313X.2009.04090.x. - Tschammer, J. Molecular characterization of grapevine cultivars of Riesling-type and of closely related Burgundies / J. Tschammer, E. Zyprian // Vitis. – 1994. – Vol. 33. – P. 249–250.
- Umehara, Y. Construction and characterization of a rice YAC library for physical mapping / Y. Umehara, A. Inagaki, H. Tanoue // Molecular Breeding. – 1995. – Vol. 15. – P. 79–89.
- Valat, L. Transgenic grapevine rootstock clones expressing the coat protein or movement protein genes of Grapevine fanleaf virus: Characterization and reaction to virus infection upon protoplast electroporation / L. Valat, M. Fuchs, M. Burrus // Plant Science. – 2006. – Vol. 170(4). – Р. 739–747. – doi.org/10.1016/j.plantsci.2005.11.005.
- Verpoorte, R. Engineering secondary metabolite production in plants /
R. Verpoorte, J. Memelink // Current opinion in biotechnology. – 2002. – Vol. 13(2). – Р. 181-187. – doi.org/10.1016/S0958-1669(02)00308-7. - Victor, M. U. A versatile microfabricated platform for electrophoresis of double– and single–stranded DNA / M. U. Victor, L. Rongsheng, S. Nimisha // Electrophoresis. – 2003. – Vol. 24. – P. 151–157.
- Vishal, P. D. A simple method for isolation of genomic DNA from fresh and dry leaves of Terminalia arjuna (Roxb.) / P. D. Vishal, V. T. Prashant, S. C. Uddhav // Electronic Journal of Biotechnology. – 2007. – Vol. 10. – P. 31–35.
- Vos, P. R. AFLP: A new technique for DNA fingerprinting / P. R. Vos,
J. Hogers // Nucl. Acids Res. – 1995. – Vol. 23. – P. 407–414. - VpSTS29/STS2 enhances fungal tolerance in grapevine through a positive feedback loop / W. Xu, F. Ma, R. Li, Q. Zhou, W. Yao, Y. Jiao, C. Zhang, J. Zhang, X. Wang, Y. Xu, Y. Wang // Plant, cell & environment. – 2019. – Vol. 42(11). –
Р. 2979–2998. – doi.org/10.1111/pce.13600. - Walter, В. Consideration on grapevine selection and certification / В. Walter, G. P. Martelli // Vitis. – 1998. – Vol. 37. – P. 87–90.
- Walter, T. W. Isoenzyme analysis of the grape / T. W. Walter,
U. Posluszny, P. G. Kevan // Can. J. Bot. – 1989. – Vol. 67. – P. 294–299. - Wang, Z. Survey of plant short tandem repeats / Z. Wang, J. L. Weber,
G. Zhong // Theor. Appl. Genet. – 1994. – Vol. 88. – P. 3–6. - Warren, F. Using simple sequence repeats (SSRs) for DNA fingerprinting germplasm accessions of grape (Vitis L.) species / F. Warren, C. Alpha // Amer. Soc. Hort. Sci. – 1998. – Vol. 123. – P. 182–188.
- Waterhouse, A. L. The phenolic wine antioxidants / A. L. Waterhouse // Handbook of Antioxidants. – 2001. – № 3. – Р. 401–416.
- Weber, J. L. Human DNA polymorphism and methods of analysis /
J. L. Weber // Curr. Opin. Biotechnology. – 1990. – Vol. 11. – P. 166–171. - White grapes arose through the mutation of two similar and adjacent regulatory genes / A. R. Walker [et al.] // The Plant Journal. – 2007. – Vol. 49, № 5. –
Р. 772–785. - Wine grape (Vitis vinifera L.) color associates with allelic variation in the domestication gene VvmybA1 / P. This, T. Lacombe, M. Cadle-Davidson, C. L. Owens // Theoretical and Applied Genetics. – 2007. – Vol. 114, № 4. – Р. 723–730.
- Xu, H. Identification of grape (Vitis) rootstocks using sequence characterized amplified region DNA markers / H. Xu, A. T. Bakalinsky // Hort. Science. – 1996. – Vol. 32. – P. 267–268.
- Ye, G. N. Analysis of the relationship between grapevine cultivars, sports and clones via DNA fingerprinting / G. N. Ye, G. Soylemezoglu, N. F. Weeden // Vitis. – 1998. – Vol. 37. – P. 33–38.
- Zhang, Q. Molekular marker diversity and hybrid sterility in indica-japonica rice crosses / Q. Zhang, K. D. Liu, G. P. Yang // Theor. Appl. Genet. – 1997. – Vol. 95. – P. 112–118.
- Zibibbo Nero characterization, a red-wine grape revertant of Muscat of Alexandria / G. De Lorenzis, M. Squadrito, L. Brancadoro, A. Scienza // Molecular biotechnology. – 2015. – Vol. 57(3). – Р. 265–274.
- Zietkiewicz, E. Genome fingerprinting by simple sequence repeat (SSR)-anchored polymerase chain reaction amplification / E. Zietkiewicz, A. Rafalski,
D. Labuda // Genomics. – 1994. – Vol. 20. – P. 176–183. - Zlenko, V. A. An optimized medium for clonal micropropagation of grapevine / V. A. Zlenko, L. P. Troshin, I. V. Kotikov // Vitis. – 1995. – Vol. 2. – P. 125–126.
- Zulini, L. Characterization of the grapevine cultivar Picolit by means of morphological descriptors and molecular markers / L. Zulini, E. Fabro, E. Peterlunger // Vitis. – 2005. – Vol. 44. – P. 35–38.
- Zulini, L. Genotyping wine and table grape cultivars from Apulia (Southern Italy) using microsatellite markers / L. Zulini, M. Russo, E. Peterlunger // Vitis. – 2002. – Vol. 41. – P. 183–187.
